| Issue |
Med Sci (Paris)
Volume 30, Number 10, Octobre 2014
|
|
|---|---|---|
| Page(s) | 864 - 873 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20143010013 | |
| Published online | 14 October 2014 | |
Le récepteur Met fête ses 30 ans
De la découverte d’un oncogène au développement de thérapies ciblées
Thirty years of Met receptor research: from the discovery of an oncogene to the development of targeted therapies
CNRS UMR 8161, Institut de biologie de Lille, Institut Pasteur de Lille, université de Lille 1 et 2, SIRIC ONCOLille, IFR142, 1, rue du Professeur Calmette, 59021
Lille, France
En 1984, le récepteur à activité tyrosine kinase Met et son ligand l’HGF (hepatocyte growth factor)/SF (scatter factor) sont découverts grâce à leur aptitude à induire la transformation et la prolifération cellulaires. Trois décennies ont permis de mettre en évidence leur rôle fondamental dans le développement et l’homéostasie de nombreuses structures, notamment des organes épithéliaux. Cette même période a permis de décrypter les bases structurales de leur interaction, ainsi qu’un réseau de signalisation complexe. Parallèlement, l’implication de la dérégulation de Met dans la tumorigenèse, supposée dès sa découverte, a été démontrée ; elle est associée à un mauvais pronostic dans de nombreux cancers. Le rôle de cette dérégulation dans des phénomènes de résistance aux thérapies actuelles commence également à être déchiffré. Sur la base de ces données, des compagnies pharmaceutiques ont développé une grande diversité d’inhibiteurs ciblant l’HGF/SF ou le récepteur Met, dont certains sont actuellement évalués dans des essais cliniques de phase III. Nous proposons dans cette revue de retracer le parcours exemplaire des recherches sur le récepteur Met, qui auront permis en trente ans de passer de la découverte fondamentale d’un oncogène au développement de thérapies ciblant son action. Cependant, de nombreuses questions restent en suspens. Par exemple, l’implication de Met dans plusieurs étapes du développement reste à préciser, de même que les mécanismes faisant de ce récepteur un facteur de résistance aux traitements actuels ou le développement probable de résistances aux thérapies ciblant sa propre activité.
Abstract
In 1984, the Met receptor and its ligand, the HGF/SF, were discovered thanks to their ability to induce cell transformation and proliferation. Thirty years of research highlighted their crucial role in the development and homeostasis of various structures, including many epithelial organs. This period also allowed unraveling the structural basis of their interaction and their complex signaling network. In parallel, Met was shown to be deregulated and associated with a poor prognosis in many cancers. Met involvement in resistance to current therapies is also being deciphered. Based on these data, pharmaceutical companies developed a variety of Met inhibitors, some of which are evaluated in phase III clinical trials. In this review, we trace the exemplary track record of research on Met receptor, which allowed moving from bench to bedside through the development of therapies targeting its activity. Many questions still remain unanswered such as the involvement of Met in several processes of development, the mechanisms involving Met in resistance to current therapies or the likely emergence of resistances to Met-targeted therapies.
© 2014 médecine/sciences – Inserm
La découverte du couple HGF/SF-Met
En 1984, il y a tout juste 30 ans, Cooper et al. identifient l’oncogène TPR (translocated promoter region ou tumor potentiating region)-Met à partir d’une lignée cellulaire traitée par un carcinogène chimique [1]. Cette protéine chimérique résulte d’un réarrangement chromosomique entre le gène Tpr et le gène Met, nommé ainsi d’après l’agent carcinogène utilisé, le N-méthyl-N’-nitro-N-nitrosoguanidine. La partie TPR entraîne la dimérisation et l’activation constitutive du domaine kinase de Met, responsable du caractère oncogénique de TPR-Met. En 1984, Nakamura et al. isolent, à partir de plasma de rats partiellement hépatectomisés, un facteur mitogène des hépatocytes, l’hepatocyte growth factor (HGF) [2]. En 1987, Stocker et al. identifient dans le surnageant de fibroblastes un facteur induisant la dispersion des cellules épithéliales nommé scatter factor (SF) [3]. En 1991, il est montré que l’HGF et le SF sont une seule et même protéine, rebaptisée HGF/SF, capable de lier le récepteur Met [4].
L’HGF/SF est une protéine hétérodimérique de 90 kDa. Elle est composée d’une sous-unité α possédant un domaine amino-terminal (domaine N) et quatre domaines en boucles (domaines kringle, K1 à K4), et d’une chaîne β présentant une forte homologie avec le domaine sérine protéase du plasminogène (domaine SPH, serine proteinase homology), mais dénuée d’activité catalytique [5] (Figure 1A).
 |
Figure 1. Domaines fonctionnels et structure de Met et de son ligand l’HGF/SF. A. L’HGF/SF est constitué de deux chaînes α et β reliées par des ponts disulfures. La sous-unité α possède un domaine amino-terminal (domaine N) et quatre domaines baptisés kringle (domaines K1 à K4) en forme de boucles stabilisées par trois liaisons disulfures intra-chaînes. La chaîne β possède un domaine présentant une forte homologie avec le domaine sérine protéase du plasminogène (domaine SPH, serine proteinase homology). B. Le récepteur Met est constitué de deux chaînes α et β reliées par des ponts disulfures. Sur sa partie extracellulaire, le récepteur est constitué d’un domaine Sema, homologue aux sémaphorines, à cheval sur la chaîne α et β. Ce domaine est suivi d’une région riche en cystéine appelée domaine PSI en raison de son homologie avec des domaines retrouvés dans les plexines, les sémaphorines et les intégrines. Puis la partie extracellulaire est constituée d’une succession de quatre domaines IPT (immunoglobulin-like fold shared by plexins and transcription factors). La structure cristallographique du domaine Sema révèle une organisation en sept pales concentriques bâties à partir de feuillets β. Met possède un domaine transmembranaire unique suivi dans la partie intracellulaire du domaine à activité tyrosine kinase. Figure extraite de [6] et reproduite avec l’autorisation de Proc Natl Acad Sci USA ; Copyright 2003 National Academy of Sciences, États-Unis. C. Structure cristallographique du domaine Sema de Met interagissant avec l’HGF/SF. Deux molécules d’HGF/SF interagissent via leurs domaines N et K1 associés tête-bêche. Chaque molécule d’HGF/SF interagit avec un domaine Sema de Met, permettant de proposer une interaction de type 2:2 (2 récepteurs:2 ligands) entre Met et l’HGF/SF. Figure extraite de [8] et reproduite avec l’autorisation de Proc Natl Acad Sci USA ; Copyright 2006, National Academy of Sciences, États-Unis. |
Le récepteur Met est synthétisé sous la forme d’un précurseur de 170 kDa, glycosylé puis clivé en une chaîne α entièrement extracellulaire et une chaîne β transmembranaire, les deux chaînes étant reliées par des ponts disulfures [5]. La partie extracellulaire est constituée d’un domaine Sema, situé à cheval sur les chaînes α et β, et homologue à un domaine des sémaphorines, des protéines de guidage des axones. Son analyse par cristallographie révèle une organisation en sept pales concentriques constituées de feuillets β. S’ensuivent un domaine PSI, ainsi nommé en raison de son homologie avec certains domaines des plexines, des sémaphorines et des intégrines, puis quatre domaines homologues aux immunoglobulines baptisés IPT (immunoglobulin-plexin-transcription factor) [6]. La partie intracellulaire de Met porte le domaine tyrosine kinase avec une structure bilobée constituée de feuillets β en position amino-terminale et d’hélices α en position carboxy-terminale [7] (Figure 1B).
L’HGF/SF lie la région extracellulaire du récepteur Met au niveau de deux sites : d’une part les domaines IPT3 et IPT4, liant le domaine N de l’HGF/SF avec une haute affinité, et, d’autre part, le domaine Sema, qui lie la chaîne β de l’HGF/SF avec une plus faible affinité. Une fois lié à Met, l’HGF/SF se dimérise via une interaction tête-bêche de ses domaines N et K1, ce qui induit la dimérisation de Met [8] (Figure 1C). L’HGF/SF est également capable de s’associer à des glycoprotéines sulfatées, de type héparine, qui favoriseraient son oligomérisation et faciliteraient ainsi la dimérisation de Met.
Implication du couple HGF/SF-Met dans le développement embryonnaire et l’homéostasie épithéliale
L’activation de Met par l’HGF/SF induit une grande variété de réponses biologiques dans des cellules en culture, touchant la survie, la prolifération, la migration, la morphogenèse, l’angiogenèse ou encore la croissance de neurites [5]. Ces propriétés sont toutes en accord avec le rôle du récepteur Met in vivo. À l’échelle de l’organisme, le couple HGF/SF-Met est impliqué dans la mise en place et l’homéostasie de nombreux tissus. En effet, au cours de l’organogenèse, Met est détecté dans les cellules épithéliales d’un grand nombre d’organes (foie, reins, poumons, etc.) alors que l’HGF/SF est exprimé par les cellules mésenchymateuses proches. Cette expression complémentaire suggère leur implication, via une action paracrine, dans la mise en place de ces organes [9]. En outre, le récepteur Met est détecté dans des myoblastes du dermomyotome et dans des cellules du système nerveux, comme les précurseurs des motoneurones, suggérant son rôle dans la mise en place de structures musculaires et nerveuses.
Dans les années 1990, l’invalidation des gènes hgf/sf et met chez la souris a permis de confirmer leur rôle crucial dans l’embryogenèse. Ainsi, des souris met-/- ou hgf/sf-/- meurent in utero à 15 jours de développement et présentent des phénotypes similaires, confirmant le lien fonctionnel entre l’HGF/SF et Met. Les embryons présentent un défaut d’organisation du trophoblaste labyrinthique du placenta, à l’origine d’une réduction des échanges materno-fœtaux qui est létale [10, 11]. La taille de leur foie est également réduite, et les muscles squelettiques sont absents des bourgeons des membres, du diaphragme et du sommet de la langue ; cette absence est liée à un défaut de prolifération et de migration de leurs précurseurs [11, 12] (Figure 2).
 |
Figure 2. Rôle de Met et de son ligand l’HGF/SF durant le développement et chez l’adulte. L’implication de l’HGF/SF et de Met durant le développement embryonnaire, puis chez l’adulte, a pu être démontrée par l’inactivation des gènes correspondants chez la souris. Ainsi, des animaux déficients pour Met ou l’HGF/SF (Met-/- ou HGF/SF-/-) meurent in utero à partir de 15 jours de développement. Des souris HGF/SF-/- présentent des défauts d’organisation du trophoblaste labyrinthique du placenta et une réduction de la taille du foie [10, 11] (photographie « Placenta » extraite de [10] et reproduite avec l’autorisation de Nature, Copyright 1995). L’extinction conditionnelle de Met spécifiquement dans le système nerveux empêche la survie d’un groupe de motoneurones innervant le muscle du petit pectoral [14], menant à des défauts moteurs (photographie « Neurones » extraite de [14] et reproduite avec l’autorisation de J Neurosci, Copyright 2011). Enfin, son extinction conditionnelle chez l’adulte inhibe la régénération de tissus comme le foie [15] (photographie « Foie » extraite de [15] et reproduite avec l’autorisation de Proc Natl Acad Sci USA, Copyright 2004 National Academy of Sciences, États-Unis). |
À partir des années 2000, l’extinction conditionnelle de Met a permis de détailler son rôle dans la mise en place de structures individuelles. Ainsi, son invalidation dans les poumons inhibe significativement le développement alvéolaire [13] ; dans le système nerveux central, elle induit la perte de sous-groupes de motoneurones [14] (Figure 2).
Chez l’adulte, le couple HGF/SF-Met joue un rôle important dans la régénération des tissus épithéliaux. Une augmentation du taux d’HGF/SF est observable après une lésion d’organes comme le foie, les reins ou la moelle épinière, et l’administration de ce facteur favorise la régénération de ces tissus. Au contraire, la régénération du foie, de l’épiderme ou des reins est inhibée par l’extinction conditionnelle de Met dans ces tissus [15] (Figure 2).
Décryptage des mécanismes moléculaires associés à l’activation de Met
La signalisation de Met commence à être décrite dès l’identification de son ligand en 1991. En réponse à la liaison de l’HGF/SF, Met se dimérise et s’autophosphoryle sur les résidus Y1234 et 1235 du domaine catalytique. Les résidus tyrosine 1349 et 1356 phosphorylés de la région carboxy-terminale constituent quant à eux un site d’ancrage capable de lier de multiples effecteurs, tels que GAB1 (Grb2-associated binding protein 1), GRB2 (growth factor receptor-bound protein 2), SHC ([Src homology 2 domain containing]transforming protein), la PI3K (phosphoinositide 3-kinase), Src, STAT3 (signal transducer and activator of transcription 3) ou la PLCγ (phospholipase Cγ) (Figure 3). La mutation de ce site d’ancrage chez la souris induit des phénotypes similaires à ceux des souris met-/-, ce qui démontre le rôle central de ces résidus dans la signalisation [16]. Une fois ces adaptateurs recrutés, diverses cascades de signalisation sont enclenchées.
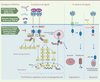 |
Figure 3. Signalisation, dégradation et stratégies d’inhibition de Met. La fixation de son ligand HGF/SF provoque la dimérisation du récepteur Met et son autophosphorylation sur les résidus tyrosine Y1234 et 1235 du domaine kinase, participant à son activation, et les résidus tyrosine 1349 et 1356 de l’extrémité carboxy-terminale constituant un site d’ancrage multisubstrats. Ce site est capable de lier de multiples effecteurs, comme GAB1, GRB2, la PI3K ou STAT3, activant ainsi de multiples voies de signalisation intracellulaire dont l’intégration conduira aux différentes réponses biologiques induites par Met, comme la prolifération, la survie, la migration ou la morphogenèse de branchement. Met s’autophosphoryle également sur la tyrosine 1003 du domaine juxtamembranaire, ce qui va induire le recrutement de Cbl, une E3 ubiquitine ligase responsable de l’ubiquitination du récepteur. Le récepteur ubiquitiné est internalisé, puis transporté vers l’endosome et finalement dégradé par le lysosome, contribuant ainsi à l’atténuation de la signalisation. D’autres sites de régulation négative siègent au niveau du domaine juxtamembranaire, dont divers sites de clivage par des protéases. Ainsi, Met subit un clivage constitutif dans son domaine extracellulaire par des métalloprotéases membranaires de la famille ADAM, puis dans la région juxtamembranaire par la γ-secrétase. Les fragments intracellulaires créés sont labiles puisqu’ils sont dégradés efficacement par le protéasome et le lysosome. Ces clivages sont impliqués dans la régulation de la demi-vie de Met. De plus, en l’absence de son ligand et en réponse à un stress apoptotique, Met est clivé par les caspases en un fragment capable d’amplifier la mort cellulaire par une action sur la perméabilisation de la mitochondrie. Les stratégies d’inhibition de Met visent, soit à interférer avec l’interaction HGF/SF-Met, soit à inhiber l’activité tyrosine kinase de Met. Les inhibiteurs de l’interaction ligand-récepteur sont soit des anticorps bloquants monoclonaux spécifiquement dirigés contre l’HGF/SF ou contre la région extracellulaire de Met, soit des antagonistes capables de lier Met sans l’activer, comme les sous-domaines de l’HGF/SF inspirés du variant naturel NK2, ou encore des sous-domaines de Met utilisés comme leurres pour l’HGF/SF. Les inhibiteurs de l’activité kinase sont en grande majorité des mimétiques de l’ATP capables d’occuper la poche ATP de Met et d’inhiber son activité. FAK : focal adhesion kinase; mTOR : mammalian target of rapamycin; MEK : MAP kinase kinase; β-cat : β-caténine; NK2 : natural splice variant NK2; ETS-1 : v-ets erythroblastosis virus E26 oncogene homolog-1. |
Par exemple, l’activation des petites protéines G Ras, Rac et PAK (p21-activated kinase) contrôle le réarrangement du cytosquelette et la motilité [17]. La voie PI3K, via l’activation d’Akt (protéine kinase B), joue aussi un rôle central dans la survie cellulaire induite par Met [18], tout comme la régulation de p53 par Abl et p38 [19].
Au cours des années 2000, différents mécanismes de régulation négative du récepteur Met ont été mis en évidence. Ainsi, l’ubiquitination de Met, à la suite du recrutement de l’E3 ubiquitine ligase Cbl par l’intermédiaire de la tyrosine juxtamembranaire phosphorylée (Y1003), induit son endocytose, son transport vers l’endosome et sa dégradation [20]. La mutation de la Y1003 induit l’activation du récepteur et augmente ses capacités de transformation cellulaire, soulignant l’importance de cette modification (Figure 3). D’autres sites de régulation siègent au niveau du domaine juxtamembranaire de Met, comme la sérine 983, dont la phosphorylation par les PKC (protéine kinase C) réprime son activité [21]. Ce domaine est également la cible de protéases. Ainsi, lors d’un stress apoptotique, Met est clivé par les caspases en un fragment capable d’amplifier la perméabilisation mitochondriale et la mort cellulaire [22]. Ces capacités pro-apoptotiques de Met, opposées à l’activité anti-apoptotique qu’induit la fixation du ligand, expliquent que Met soit classé parmi les récepteurs à dépendance (Figure 3) [49]. De plus, la demi-vie de Met est régulée par un premier clivage constitutif dans son domaine extracellulaire par des métalloprotéases de la famille ADAM (a disintegrin and metalloproteinase), suivi d’un second clivage par la γ-sécrétase dans la région juxtamembranaire [23] (Figure 3).
L’internalisation de Met n’est pas uniquement synonyme de dégradation, mais elle participe aussi activement à sa signalisation. Ainsi, en réponse à l’HGF/SF, Met se déplace avec le facteur de transcription STAT3 (signal transducer and activator of transcription 3) vers les endosomes périnucléaires. Ce trafic est impliqué dans la translocation nucléaire de STAT3 et son activité, importante notamment pour la transformation cellulaire induite par Met [24].
Le réseau social de Met
Durant les années 2000, les partenaires de Met à la membrane plasmique ont été identifiés ; ils contribuent à l’efficacité de son activation. L’association entre Met et l’intégrine α6β4 est ainsi importante pour l’invasion cellulaire [25]. Met peut aussi interagir avec l’intégrine α3β1, favorisant le recrutement de GAB1 et de la PI3K, et le contrôle de la voie Wnt [26]. L’interaction de Met avec l’isoforme CD44v6 favorise quant à elle la phosphorylation de Met par son ligand, mais aussi une activation plus efficace de la voie Ras-ERK (extracellular signal-regulated kinase), via l’association de la région intracellulaire de CD44v6 avec les protéines ERM (ezrine, radixine et moésine) [27]. L’interaction de Met avec les récepteurs de type plexine stimule par ailleurs l’invasion ou l’angiogenèse induites par les sémaphorines [28]. Met peut aussi dialoguer avec d’autres récepteurs à activité tyrosine kinase comme l’EGFR (epidermal growth factor receptor), ce qui est important dans différents cancers et sera discuté plus loin. Enfin, Met peut interagir avec Fas, un récepteur de mort. L’HGF/SF peut provoquer la dissociation d’un complexe entre Met et Fas, sensibilisant ainsi les cellules à l’apoptose [29].
Mise en évidence des dérégulations du couple HGF/SF-Met dans les cancers
Les capacités du couple HGF/SF-Met à induire la survie et la motilité cellulaires, ou encore l’angiogenèse, sont autant de propriétés qui peuvent contribuer à la tumorigenèse. Le lien formel entre l’activation aberrante de Met et le cancer a été établi en 1997 par l’identification de mutations de Met associées à l’apparition de carcinomes rénaux héréditaires [30]. Depuis, plus de 150 mutations somatiques ponctuelles de Met ont été répertoriées dans divers cancers (d’après le site COSMIC, catalogue of somatic mutations in cancer). La plupart des mutations du domaine catalytique favorisent l’activité kinase, permettant l’induction des voies de signalisation, mais également le recyclage du récepteur aux dépens de sa dégradation [31]. De fait, des souris transgéniques portant ces mutations développent de nombreux cancers [32, 33].
La surexpression de Met a été aussi fréquemment observée, notamment dans près de 80 % des cancers gastriques et rénaux [34, 35]. De nombreux mécanismes peuvent induire une telle surexpression. Outre l’existence d’amplifications géniques, différents oncogènes comme Ras, Ret ou ETS-1 (v-ets erythroblastosis virus E26 oncogene homolog-1) augmentent la transcription du gène Met [36]. L’expression de Met est également réprimée par le microARN miR-34b, qui est régulé par p53 et pourrait donc relier l’inactivation de p53 à la surexpression de Met. L’activation aberrante de Met peut également résulter d’une surexpression de son ligand, l’HGF/SF. De manière générale, la surexpression de l’HGF/SF ou de Met est de mauvais pronostic.
Un dernier élément, et non le moindre dans le contexte des thérapies anticancéreuses, réside dans les résistances aux thérapies ciblant l’EGFR (epidermal growth factor receptor) dans les cancers du poumon et du côlon que caractérise une surexpression de Met [37, 38]. L’activation de Met permet alors de compenser l’inhibition de l’EGFR, et ceci de manière d’autant plus efficace que les réseaux de signalisation des deux récepteurs à activité tyrosine kinase sont très similaires.
Les stratégies thérapeutiques
Les stratégies d’inhibition de Met développées par les laboratoires pharmaceutiques et académiques visent, soit à perturber l’interaction de l’HGF/SF avec Met à l’aide d’anticorps bloquants, soit à inhiber l’activité tyrosine kinase de Met à l’aide d’inhibiteurs de tyrosine kinases (TKI) (Figure 3). Ces stratégies, en développement depuis le début des années 2000, ont clairement démontré leur efficacité dans des modèles précliniques de cancers où Met est dérégulé [48]. L’explosion du nombre d’essais mis en place ces trois dernières années, passant de 95 en 2011 à plus de 240 aujourd’hui, témoigne de la confiance placée par l’industrie pharmaceutique dans le potentiel de ces nouvelles thérapies. Ces essais concernent 18 composés évalués en phases I et II; seuls cinq composés sont actuellement évalués en phase III (Tableau I).
Essais cliniques pour l’évaluation d’inhibiteurs pharmacologiques et d’anticorps bloquant contre Met ou l’HGF dans les cancers. Liste des anticorps bloquants dirigés contre Met ou l’HGF, et des inhibiteurs de tyrosine kinase en cours d’évaluation clinique. Pour chaque molécule, les essais répertoriés sous http://clinicaltrials.gov en mars 2014 sont indiqués avec les pathologies pour lesquelles la molécule est évaluée. CBNPC : cancer bronchique « non à petites cellules ». EGFRi : inhibiteur de l’EGFR
Les inhibiteurs de tyrosine kinases dirigés contre Met sont en grande majorité des mimétiques de l’ATP, capables d’occuper la poche ATP du domaine kinase (Figure 3). Le K252a, un alcaloïde naturel analogue de la staurosporine, a été le premier inhibiteur ciblant Met identifié, mais il présente une faible sélectivité [39]. Rapidement, d’autres compétiteurs de l’ATP, cette fois plus spécifiques, ont été développés comme le PHA-665752 [40], encore largement utilisé en recherche. Aujourd’hui, une douzaine d’inhibiteurs spécifiques de Met sont évalués dans des essais de phase I/II, comme le JNJ38877605.
Les inhibiteurs les plus avancés dans leur développement sont des inhibiteurs à spectre d’action plus large. Le Crizotinib, un inhibiteur de Met, d’ALK (anaplastic lymphoma kinase) et de ROS1 (c-ros oncogène 1) est ainsi prescrit chez les patients atteints d’un cancer bronchique « non à petites cellules » (CBNPC) présentant une translocation d’ALK. La capacité du Crizotinib à inhiber également Met pourrait être un avantage, puisque cela permettrait à la fois de contrecarrer l’activation d’ALK et de réduire les risques de résistance impliquant des amplifications de Met [41].
Le seul inhibiteur spécifique de Met dont l’efficacité est actuellement évaluée dans des essais de phase III est le Tivantinib. Toutefois, un premier essai de phase III, arrêté prématurément en 2012, n’a pas permis de montrer l’efficacité d’une association du Tivantinib avec l’Erlotinib sur la survie globale de patients atteints de cancer bronchique non à petites cellules. Cependant, une amélioration de la survie sans progression tumorale des patients dont la tumeur présente une surexpression et/ou une amplification du gène met a été constatée.
Les anticorps monoclonaux (mAb) bloquants dont le développement a débuté en 1998 avec la mise au point du DN-30 [42], représentent une des stratégies les plus prometteuses1. Ils sont dirigés contre l’HGF/SF ou la région extracellulaire du récepteur Met et empêchent leur interaction (Figure 3) [48]. Par exemple, l’anticorps AMG 102 (Rilotumumab), dirigé contre l’HGF/SF, est en cours d’évaluation dans un essai de phase III chez des patients atteints de cancers gastriques ou d’adénocarcinomes de la jonction œsophagienne qui surexpriment la protéine Met. Parmi les anticorps dirigés contre Met, le MetMab (Onartuzumab) est un anticorps monoclonal humanisé monovalent, pour lequel un seul bras de l’anticorps est utilisé dans le but de prévenir la dimérisation du récepteur. Cet anticorps empêche l’interaction avec l’HGF/SF en liant le domaine Sema de Met et prévient la croissance tumorale des cellules cancéreuses stimulées par l’HGF/SF dans des modèles murins [43]. Cependant, l’association Onartuzumab et Tarceva (inhibiteur de tyrosine kinase anti-EGFR), testée en phase III chez des patients atteints de cancer bronchique non à petites cellules exprimant Met, a été arrêté prématurément puisqu’il n’apportait pas de bénéfice clinique. D’autres essais cliniques de phase III évaluant l’efficacité de cet anticorps sont encore en cours.
De façon intéressante, d’autres mAb dirigés contre le domaine extracellulaire de Met ont une action inhibitrice sans interférer avec l’interaction ligand/récepteur. Par exemple, l’anticorps monoclonal DN30 favorise la dégradation de Met dans des modèles murins en activant son clivage par les métalloprotéases et le complexe γ-sécrétase, et réduit ainsi la croissance tumorale [44].
D’autres stratégies sont développées au niveau préclinique pour inhiber la signalisation HGF/SF-Met. Par exemple, des antagonistes capables de lier Met sans l’activer sont en cours de développement, comme des sous-domaines de l’HGF/SF, inspirés du variant naturel NK2 et capables de se fixer à Met sans induire son activation [45]. De la même façon, des molécules antagonistes ont été développées en utilisant des sous-domaines de Met dans le but de créer des leurres pour l’HGF/SF. Ces sous-domaines, correspondant à la totalité de la région extracellulaire ou au domaine Sema, sont capables d’inhiber des réponses induites par l’HGF/SF et réduisent la croissance de tumeurs expérimentales [46].
Perspectives
En 1984, le récepteur Met et son ligand l’HGF/SF étaient découverts grâce à leur capacité à induire la transformation et la prolifération cellulaires. En trois décennies, le réseau de signalisation nécessaire au développement embryonnaire et à la régénération d’organes épithéliaux a été finement décrypté. Parallèlement, l’implication de la dérégulation de Met dans la tumorigenèse a été clairement démontrée et associée à un mauvais pronostic dans de nombreux types de cancers (www.vai.org/Met/). Sur la base de ces données, des compagnies pharmaceutiques ont développé des inhibiteurs qui, pour certains, sont en cours d’évaluation clinique. La question est souvent posée du temps nécessaire au passage d’une découverte fondamentale vers une application clinique. Pour le récepteur Met, 30 ans ont été nécessaires. Ce parcours est tout à fait représentatif de celui d’autres RTK vis-à-vis desquels des thérapies ciblées sont également en cours de développement (Figure 4).
 |
Figure 4. Chronologie des recherches sur HGF/SF-Met de la découverte de Met jusqu’à aujourd’hui. Principales découvertes depuis 1984 à nos jours ayant permis de décrypter l’implication de Met dans les processus physiologiques, mais également dans les processus pathologiques comme les cancers. PS-RIP : presenilin-dependent regulated intramembrane proteolysis. |
Cependant, il faut noter deux échecs récents dans les essais de phase III évaluant, dans les cancers bronchiques non à petites cellules, un inhibiteur de tyrosine kinase et un anticorps anti-Met en association avec un inhibiteur de l’EGFR. Pour ces deux études, les patients éligibles au traitement ont été sélectionnés sur la base de la surexpression de Met dans les tumeurs, soit environ la moitié des patients atteints de ces cancers. Or, une surexpression de Met n’est pas forcément synonyme d’activation et moins encore d’une sensibilité accrue aux traitements. Une réévaluation des biomarqueurs prédictifs de l’efficacité des traitements semble donc nécessaire. Par exemple, l’activation de Met pourrait être directement évaluée en recherchant par immunohistochimie sa phosphorylation, ou via l’expression de l’HGF/SF dans la tumeur ou dans le sérum. De plus, le dialogue entre Met et les autres récepteurs membranaires est loin d’être bien compris. Ces dialogues sont importants, comme le montrent les liaisons dangereuses qu’entretiennent Met et l’EGFR dans les cancers bronchiques non à petites cellules. Enfin, l’arrivée des thérapies ciblant Met expose à de nouvelles questions, à commencer par le développement de résistances, déjà illustré à partir de modèles cellulaires dans lesquels un traitement avec des anti-Met induit des résistances impliquant les suspects habituels, comme KRas [47]. Enfin, l’avènement des thérapies ciblant Met ne doit pas occulter les nombreuses questions qui restent à élucider. Par exemple, l’implication de Met dans le développement d’organes épithéliaux comme le poumon n’a été démontrée que très récemment par le biais de modèles animaux élaborés. Il est probable que Met soit impliqué dans le développement et l’homéostasie d’autres organes pour lesquels des modèles peuvent être développés.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Les travaux de recherche de l’équipe « signalisation, apoptose et cancer » de l’UMR 8161 sont soutenus par le CNRS, l’Institut Pasteur de Lille et l’Inserm, et par des financements de la Ligue contre le cancer (Comité nord), de l’Association pour la recherche sur le cancer, de l’Institut national du cancer, du Cancéropôle Nord-Ouest et du SIRIC ONCOLille.
Références
- Cooper CS, Park M, Blair DG, et al. Molecular cloning of a new transforming gene from a chemically transformed human cell line. Nature 1984 ; 311 : 29–33. [CrossRef] [PubMed] [Google Scholar]
- Nakamura T, Nawa K, Ichihara A. Partial purification and characterization of hepatocyte growth factor from serum of hepatectomized rats. Biochem Biophys Res Commun 1984 ; 122 : 1450–1459. [CrossRef] [PubMed] [Google Scholar]
- Stoker M, Gherardi E, Perryman M, Gray J. Scatter factor is a fibroblast-derived modulator of epithelial cell mobility. Nature 1987 ; 327 : 239–242. [CrossRef] [PubMed] [Google Scholar]
- Weidner KM, Arakaki N, Hartmann G, et al. Evidence for the identity of human scatter factor and human hepatocyte growth factor. Proc Natl Acad Sci USA 1991 ; 88 : 7001–7005. [CrossRef] [Google Scholar]
- Gherardi E, Birchmeier W, Birchmeier C, Woude GV. Targeting MET in cancer: rationale and progress. Nat Rev Cancer 2012 ; 12 : 89–103. [CrossRef] [PubMed] [Google Scholar]
- Gherardi E, Youles ME, Miguel RN, et al. Functional map and domain structure of MET, the product of the c-met protooncogene and receptor for hepatocyte growth factor/scatter factor. Proc Natl Acad Sci USA 2003 ; 100 : 12039–12044. [CrossRef] [Google Scholar]
- Schiering N, Knapp S, Marconi M, et al. Crystal structure of the tyrosine kinase domain of the hepatocyte growth factor receptor c-Met and its complex with the microbial alkaloid K-252a. Proc Natl Acad Sci USA 2003 ; 100 : 12654–12659. [CrossRef] [Google Scholar]
- Gherardi E, Sandin S, Petoukhov MV, et al. Structural basis of hepatocyte growth factor/scatter factor and MET signalling. Proc Natl Acad Sci USA 2006 ; 103 : 4046–4051. [CrossRef] [Google Scholar]
- Andermarcher E, Surani MA, Gherardi E. Co-expression of the HGF/SF and c-met genes during early mouse embryogenesis precedes reciprocal expression in adjacent tissues during organogenesis. Dev Genet 1996 ; 18 : 254–266. [CrossRef] [PubMed] [Google Scholar]
- Uehara Y, Minowa O, Mori C, et al. Placental defect and embryonic lethality in mice lacking hepatocyte growth factor/scatter factor. Nature 1995 ; 373 : 702–705. [CrossRef] [PubMed] [Google Scholar]
- Schmidt C, Bladt F, Goedecke S, et al. Scatter factor/hepatocyte growth factor is essential for liver development. Nature 1995 ; 373 : 699–702. [CrossRef] [PubMed] [Google Scholar]
- Bladt F, Riethmacher D, Isenmann S, et al. Essential role for the c-met receptor in the migration of myogenic precursor cells into the limb bud. Nature 1995 ; 376 : 768–771. [CrossRef] [PubMed] [Google Scholar]
- Calvi C, Podowski M, Lopez-Mercado A, et al. Hepatocyte growth factor, a determinant of airspace homeostasis in the murine lung. PLoS Genet 2013 ; 9 : e1003228. [CrossRef] [PubMed] [Google Scholar]
- Lamballe F, Genestine M, Caruso N, et al. Pool-specific regulation of motor neuron survival by neurotrophic support. J Neurosci 2011 ; 31 : 11144–11158. [CrossRef] [PubMed] [Google Scholar]
- Huh CG, Factor VM, Sanchez A, et al. Hepatocyte growth factor/c-met signaling pathway is required for efficient liver regeneration and repair. Proc Natl Acad Sci USA 2004 ; 101 : 4477–4482. [CrossRef] [Google Scholar]
- Maina F, Casagranda F, Audero E, et al. Uncoupling of Grb2 from the Met receptor in vivo reveals complex roles in muscle development. Cell 1996 ; 87 : 531–542. [CrossRef] [PubMed] [Google Scholar]
- Royal I, Lamarche-Vane N, Lamorte L, et al. Activation of cdc42, rac, PAK, and rho-kinase in response to hepatocyte growth factor differentially regulates epithelial cell colony spreading and dissociation. Mol Biol Cell 2000 ; 11 : 1709–1725. [CrossRef] [PubMed] [Google Scholar]
- Moumen A, Ieraci A, Patane S, et al. Met signals hepatocyte survival by preventing Fas-triggered FLIP degradation in a PI3k-Akt-dependent manner. Hepatology 2007 ; 45 : 1210–1217. [CrossRef] [PubMed] [Google Scholar]
- Furlan A, Stagni V, Hussain A, et al. Abl interconnects oncogenic Met and p53 core pathways in cancer cells. Cell Death Differ 2011 ; 18 : 1608–1616. [CrossRef] [PubMed] [Google Scholar]
- Peschard P, Fournier TM, Lamorte L, et al. Mutation of the c-Cbl TKB domain binding site on the Met receptor tyrosine kinase converts it into a transforming protein. Mol Cell 2001 ; 8 : 995–1004. [CrossRef] [PubMed] [Google Scholar]
- Hashigasako A, Machide M, Nakamura T, Matsumoto K. Bi-directional regulation of Ser-985 phosphorylation of c-met via protein kinase C and protein phosphatase 2A involves c-Met activation and cellular responsiveness to hepatocyte growth factor. J Biol Chem 2004 ; 279 : 26445–26452. [CrossRef] [PubMed] [Google Scholar]
- Tulasne D, Deheuninck J, Lourenco FC, et al. Proapoptotic function of the MET tyrosine kinase receptor through caspase cleavage. Mol Cell Biol 2004 ; 24 : 10328–10339. [CrossRef] [PubMed] [Google Scholar]
- Ancot F, Leroy C, Muharram G, et al. Shedding-generated Met receptor fragments can be routed to either the proteasomal or the lysosomal degradation pathway. Traffic 2012 ; 13 : 1261–1272. [CrossRef] [PubMed] [Google Scholar]
- Kermorgant S, Parker PJ. Receptor trafficking controls weak signal delivery: a strategy used by c-Met for STAT3 nuclear accumulation. J Cell Biol 2008 ; 182 : 855–863. [CrossRef] [PubMed] [Google Scholar]
- Trusolino L, Bertotti A, Comoglio PM. A signaling adapter function for alpha6beta4 integrin in the control of HGF-dependent invasive growth. Cell 2001 ; 107 : 643–654. [CrossRef] [PubMed] [Google Scholar]
- Liu Y, Chattopadhyay N, Qin S, et al. Coordinate integrin and c-Met signaling regulate Wnt gene expression during epithelial morphogenesis. Development 2009 ; 136 : 843–853. [CrossRef] [PubMed] [Google Scholar]
- Orian-Rousseau V, Morrison H, Matzke A, et al. Hepatocyte growth factor-induced Ras activation requires ERM proteins linked to both CD44v6 and F-actin. Mol Biol Cell 2007 ; 18 : 76–83. [CrossRef] [PubMed] [Google Scholar]
- Sadanandam A, Rosenbaugh EG, Singh S, et al. Semaphorin 5A promotes angiogenesis by increasing endothelial cell proliferation, migration, and decreasing apoptosis. Microvasc Res 2010 ; 79 : 1–9. [CrossRef] [PubMed] [Google Scholar]
- Wang X, DeFrances MC, Dai Y, et al. A mechanism of cell survival: sequestration of Fas by the HGF receptor Met. Mol Cell 2002 ; 9 : 411–421. [CrossRef] [PubMed] [Google Scholar]
- Schmidt L, Duh FM, Chen F, et al. Germline and somatic mutations in the tyrosine kinase domain of the MET proto-oncogene in papillary renal carcinomas. Nat Genet 1997 ; 16 : 68–73. [CrossRef] [PubMed] [Google Scholar]
- Joffre C, Barrow R, Menard L, et al. A direct role for Met endocytosis in tumorigenesis. Nat Cell Biol 2011 ; 13 : 827–837. [CrossRef] [PubMed] [Google Scholar]
- Jeffers M, Fiscella M, Webb CP, et al. The mutationally activated Met receptor mediates motility and metastasis. Proc Natl Acad Sci USA 1998 ; 95 : 14417–14422. [CrossRef] [Google Scholar]
- Graveel C, Su Y, Koeman J, et al. Activating Met mutations produce unique tumor profiles in mice with selective duplication of the mutant allele. Proc Natl Acad Sci USA 2004 ; 101 : 17198–17203. [CrossRef] [Google Scholar]
- Ponzetto C, Giordano S, Peverali F, et al. c-met is amplified but not mutated in a cell line with an activated met tyrosine kinase. Oncogene 1991 ; 6 : 553–559. [PubMed] [Google Scholar]
- Natali PG, Prat M, Nicotra MR, et al. Overexpression of the met/HGF receptor in renal cell carcinomas. Int J Cancer 1996 ; 69 : 212–217. [CrossRef] [PubMed] [Google Scholar]
- Ian M, Bond JA, Prat M, et al. Activated ras and ret oncogenes induce over-expression of c-met (hepatocyte growth factor receptor) in human thyroid epithelial cells. Oncogene 1997 ; 14 : 2417–2423. [CrossRef] [PubMed] [Google Scholar]
- Engelman JA, Zejnullahu K, Mitsudomi T, et al. MET amplification leads to gefitinib resistance in lung cancer by activating ERBB3 signaling. Science 2007 ; 316 : 1039–1043. [CrossRef] [PubMed] [Google Scholar]
- Bardelli A, Corso S, Bertotti A, et al. Amplification of the MET receptor drives resistance to anti-EGFR therapies in colorectal cancer. Cancer Discov 2013 ; 3 : 658–673. [CrossRef] [PubMed] [Google Scholar]
- Morotti A, Mila S, Accornero P, et al. K252a inhibits the oncogenic properties of Met, the HGF receptor. Oncogene 2002 ; 21 : 4885–4893. [CrossRef] [PubMed] [Google Scholar]
- Christensen JG, Schreck R, Burrows J, et al. A selective small molecule inhibitor of c-Met kinase inhibits c-Met-dependent phenotypes in vitro and exhibits cytoreductive antitumor activity in vivo. Cancer Res 2003 ; 63 : 7345–7355. [PubMed] [Google Scholar]
- Gridelli C, Peters S, Sgambato A, et al. ALK inhibitors in the treatment of advanced NSCLC. Cancer Treat Rev 2013 ; 40 : 300–306. [CrossRef] [PubMed] [Google Scholar]
- Prat M, Crepaldi T, Pennacchietti S, et al. Agonistic monoclonal antibodies against the Met receptor dissect the biological responses to HGF. J Cell Sci 1998 ; 111 : 237–247. [PubMed] [Google Scholar]
- Merchant M, Ma X, Maun HR, et al. Monovalent antibody design and mechanism of action of onartuzumab, a MET antagonist with anti-tumor activity as a therapeutic agent. Proc Natl Acad Sci USA 2013 ; 110 : E2987–E2996. [CrossRef] [Google Scholar]
- Foveau B, Ancot F, Leroy C, et al. Downregulation of the Met receptor tyrosine kinase by presenilin-dependent regulated intramembrane proteolysis. Mol Biol Cell 2009 ; 20 : 2494–2506. [CrossRef] [Google Scholar]
- Matsumoto K, Nakamura T, Sakai K, Nakamura T. Hepatocyte growth factor and Met in tumor biology and therapeutic approach with NK4. Proteomics 2008 ; 8 : 3360–3370. [CrossRef] [PubMed] [Google Scholar]
- Coxon A, Rex K, Meyer S, et al. Soluble c-Met receptors inhibit phosphorylation of c-Met and growth of hepatocyte growth factor: c-Met-dependent tumors in animal models. Mol Cancer Ther 2009 ; 8 : 1119–1125. [CrossRef] [PubMed] [Google Scholar]
- Cepero V, Sierra JR, Corso S, et al. MET and KRAS gene amplification mediates acquired resistance to MET tyrosine kinase inhibitors. Cancer Res 2010 ; 70 : 7580–7590. [CrossRef] [PubMed] [Google Scholar]
- Bodmer A, Goetsch L, Laurence Favet L, et al. Anticorps et tumeurs solides. Cibles établies et pistes prometteuses. Med Sci (Paris) 2009 ; 25 : 1090–1098. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Furne C, Mehlen P. Les radeaux de la mort : quand les radeaux lipidiques régulent les récepteurs à dépendance. Med Sci (Paris) 2006 ; 22 : 909–910. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des tableaux
Essais cliniques pour l’évaluation d’inhibiteurs pharmacologiques et d’anticorps bloquant contre Met ou l’HGF dans les cancers. Liste des anticorps bloquants dirigés contre Met ou l’HGF, et des inhibiteurs de tyrosine kinase en cours d’évaluation clinique. Pour chaque molécule, les essais répertoriés sous http://clinicaltrials.gov en mars 2014 sont indiqués avec les pathologies pour lesquelles la molécule est évaluée. CBNPC : cancer bronchique « non à petites cellules ». EGFRi : inhibiteur de l’EGFR
Liste des figures
 |
Figure 1. Domaines fonctionnels et structure de Met et de son ligand l’HGF/SF. A. L’HGF/SF est constitué de deux chaînes α et β reliées par des ponts disulfures. La sous-unité α possède un domaine amino-terminal (domaine N) et quatre domaines baptisés kringle (domaines K1 à K4) en forme de boucles stabilisées par trois liaisons disulfures intra-chaînes. La chaîne β possède un domaine présentant une forte homologie avec le domaine sérine protéase du plasminogène (domaine SPH, serine proteinase homology). B. Le récepteur Met est constitué de deux chaînes α et β reliées par des ponts disulfures. Sur sa partie extracellulaire, le récepteur est constitué d’un domaine Sema, homologue aux sémaphorines, à cheval sur la chaîne α et β. Ce domaine est suivi d’une région riche en cystéine appelée domaine PSI en raison de son homologie avec des domaines retrouvés dans les plexines, les sémaphorines et les intégrines. Puis la partie extracellulaire est constituée d’une succession de quatre domaines IPT (immunoglobulin-like fold shared by plexins and transcription factors). La structure cristallographique du domaine Sema révèle une organisation en sept pales concentriques bâties à partir de feuillets β. Met possède un domaine transmembranaire unique suivi dans la partie intracellulaire du domaine à activité tyrosine kinase. Figure extraite de [6] et reproduite avec l’autorisation de Proc Natl Acad Sci USA ; Copyright 2003 National Academy of Sciences, États-Unis. C. Structure cristallographique du domaine Sema de Met interagissant avec l’HGF/SF. Deux molécules d’HGF/SF interagissent via leurs domaines N et K1 associés tête-bêche. Chaque molécule d’HGF/SF interagit avec un domaine Sema de Met, permettant de proposer une interaction de type 2:2 (2 récepteurs:2 ligands) entre Met et l’HGF/SF. Figure extraite de [8] et reproduite avec l’autorisation de Proc Natl Acad Sci USA ; Copyright 2006, National Academy of Sciences, États-Unis. |
| Dans le texte | |
 |
Figure 2. Rôle de Met et de son ligand l’HGF/SF durant le développement et chez l’adulte. L’implication de l’HGF/SF et de Met durant le développement embryonnaire, puis chez l’adulte, a pu être démontrée par l’inactivation des gènes correspondants chez la souris. Ainsi, des animaux déficients pour Met ou l’HGF/SF (Met-/- ou HGF/SF-/-) meurent in utero à partir de 15 jours de développement. Des souris HGF/SF-/- présentent des défauts d’organisation du trophoblaste labyrinthique du placenta et une réduction de la taille du foie [10, 11] (photographie « Placenta » extraite de [10] et reproduite avec l’autorisation de Nature, Copyright 1995). L’extinction conditionnelle de Met spécifiquement dans le système nerveux empêche la survie d’un groupe de motoneurones innervant le muscle du petit pectoral [14], menant à des défauts moteurs (photographie « Neurones » extraite de [14] et reproduite avec l’autorisation de J Neurosci, Copyright 2011). Enfin, son extinction conditionnelle chez l’adulte inhibe la régénération de tissus comme le foie [15] (photographie « Foie » extraite de [15] et reproduite avec l’autorisation de Proc Natl Acad Sci USA, Copyright 2004 National Academy of Sciences, États-Unis). |
| Dans le texte | |
 |
Figure 3. Signalisation, dégradation et stratégies d’inhibition de Met. La fixation de son ligand HGF/SF provoque la dimérisation du récepteur Met et son autophosphorylation sur les résidus tyrosine Y1234 et 1235 du domaine kinase, participant à son activation, et les résidus tyrosine 1349 et 1356 de l’extrémité carboxy-terminale constituant un site d’ancrage multisubstrats. Ce site est capable de lier de multiples effecteurs, comme GAB1, GRB2, la PI3K ou STAT3, activant ainsi de multiples voies de signalisation intracellulaire dont l’intégration conduira aux différentes réponses biologiques induites par Met, comme la prolifération, la survie, la migration ou la morphogenèse de branchement. Met s’autophosphoryle également sur la tyrosine 1003 du domaine juxtamembranaire, ce qui va induire le recrutement de Cbl, une E3 ubiquitine ligase responsable de l’ubiquitination du récepteur. Le récepteur ubiquitiné est internalisé, puis transporté vers l’endosome et finalement dégradé par le lysosome, contribuant ainsi à l’atténuation de la signalisation. D’autres sites de régulation négative siègent au niveau du domaine juxtamembranaire, dont divers sites de clivage par des protéases. Ainsi, Met subit un clivage constitutif dans son domaine extracellulaire par des métalloprotéases membranaires de la famille ADAM, puis dans la région juxtamembranaire par la γ-secrétase. Les fragments intracellulaires créés sont labiles puisqu’ils sont dégradés efficacement par le protéasome et le lysosome. Ces clivages sont impliqués dans la régulation de la demi-vie de Met. De plus, en l’absence de son ligand et en réponse à un stress apoptotique, Met est clivé par les caspases en un fragment capable d’amplifier la mort cellulaire par une action sur la perméabilisation de la mitochondrie. Les stratégies d’inhibition de Met visent, soit à interférer avec l’interaction HGF/SF-Met, soit à inhiber l’activité tyrosine kinase de Met. Les inhibiteurs de l’interaction ligand-récepteur sont soit des anticorps bloquants monoclonaux spécifiquement dirigés contre l’HGF/SF ou contre la région extracellulaire de Met, soit des antagonistes capables de lier Met sans l’activer, comme les sous-domaines de l’HGF/SF inspirés du variant naturel NK2, ou encore des sous-domaines de Met utilisés comme leurres pour l’HGF/SF. Les inhibiteurs de l’activité kinase sont en grande majorité des mimétiques de l’ATP capables d’occuper la poche ATP de Met et d’inhiber son activité. FAK : focal adhesion kinase; mTOR : mammalian target of rapamycin; MEK : MAP kinase kinase; β-cat : β-caténine; NK2 : natural splice variant NK2; ETS-1 : v-ets erythroblastosis virus E26 oncogene homolog-1. |
| Dans le texte | |
 |
Figure 4. Chronologie des recherches sur HGF/SF-Met de la découverte de Met jusqu’à aujourd’hui. Principales découvertes depuis 1984 à nos jours ayant permis de décrypter l’implication de Met dans les processus physiologiques, mais également dans les processus pathologiques comme les cancers. PS-RIP : presenilin-dependent regulated intramembrane proteolysis. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




