| Issue |
Med Sci (Paris)
Volume 40, Number 4, Avril 2024
|
|
|---|---|---|
| Page(s) | 338 - 341 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2024035 | |
| Published online | 23 April 2024 | |
Les G-quadruplex favorisent la réplication du virus de l’hépatite B en activant la transcription de l’ADNccc et sa séparation de phase dans les hépatocytes
cccDNA phase separates in a G-quadruplex-dependent manner to facilitate its transcription and HBV replication
Inserm U1052, CNRS UMR5286, Centre de recherche en cancérologie de Lyon, Institut d’hépatologie de Lyon, Université Claude Bernard Lyon 1, Lyon, France
Les G-quadruplex (G4) désignent des structures secondaires non canoniques des acides nucléiques impliquant des séquences nucléotidiques particulières riches en guanine (G). L’analyse biophysique de plusieurs G4 identifiées dans le génome humain a permis de prédire la séquence consensus G≥3 – N1-7 - G≥3 - N1-7 - G≥3 – N1-7 - G≥3, dans laquelle les N représentent tous les nucléotides formant une boucle entre les structures planaires constituées par les liaisons hydrogène de Hoogsteen liant les guanines entre elles 1 (Figure 1A). Ces G4 sont des structures très dynamiques, qui subissent des cycles répétés de formation et résolution sous l’effet de protéines qui les reconnaissent [1]. Des expériences d’immuno-précipitation de la chromatine à l’aide d’un anticorps spécifique de ces structures, combinées à du séquençage de l’ADN à haut-débit, ont permis d’en identifier près de 700 000 dans le génome humain, avec une prépondérance dans des régions impliquées dans l’expression génique, telles que les promoteurs2. En accord avec ce constat, plusieurs études montrent l’importance de ces structures G4 dans la régulation transcriptionnelle de nombreux gènes, comme l’oncogène c-MYC [2, 3]. Elles constituent également des plateformes de recrutement de protéines régulatrices de l’expression génique. Certaines de ces protéines, comme la protéine FUS (fused in sarcoma), sont capables de s’oligomériser et de former ainsi, dans le noyau, des « organelles sans membrane » selon un processus de « séparation de phase » (Figure 1A). Ces organelles favorisent les interactions entre plusieurs protéines et acides nucléiques impliqués dans un même processus biologique, rendant ainsi plus efficace une réponse de la cellule à son environnement. Des structures G4 ont également été identifiées dans le génome de nombreux virus, et sont impliquées dans différentes étapes de leur réplication [4]. Une analyse in silico des séquences des génomes viraux a montré l’existence d’une corrélation entre le nombre de structures G4 contenues dans le génome viral et la propension du virus à provoquer une infection chronique chez son hôte [5].
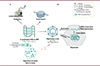 |
Figure 1. Importance fonctionnelle des structures G-quadruplex dans l’expression des gènes et dans la réplication virale : exemple du virus de l’hépatite B. A. Les G-quadruplex (G4) désignent des structures secondaires non canoniques des acides nucléiques se formant dans des régions riches en guanine (G), dont la séquence nucléotidique consensus est indiquée. Leur localisation préférentielle dans des régions régulatrices du génome et des ARN humains en font des acteurs importants du contrôle de l’expression génique, notamment de la transcription et de la traduction des transcrits en protéines. Les structures G4 favorisent la « séparation de phase » dans le noyau cellulaire via le recrutement de protéines capables de s’oligomériser, telles que la protéine FUS (elle s’oligomérise grâce à son domaine PLD et se lie aux structures G4 par son domaine RGG3), et de former ainsi des condensats nucléaires, qui sont impliqués dans différents processus biologiques comme la transcription. Ces structures G4 sont également présentes dans le génome de différents virus, dont ils contribuent à la réplication, comme nous le montrons ici pour le virus de l’hépatite B [9]. B. Le virus de l’hépatite B comporte une enveloppe et une capside contenant le génome viral : un ADN relâché circulaire partiellement double brin. Après l’entrée du virus dans les hépatocytes, la capside est importée dans le noyau et le génome viral est converti en « ADN circulaire clos de façon covalente » (ADNccc), un véritable épisome « chromatinisé ». Cet ADNccc est la matrice unique pour la synthèse, par l’ARN polymérase II cellulaire, des ARNm viraux, dont l’ARN pré-génomique, qui est ensuite rétro-transcrit et encapsidé pour produire de nouvelles particules virales infectieuses. Cette figure a été créée avec le logiciel Biorender (www.biorender.com). |
L’infection chronique de près de 300 millions de personnes dans le monde par le virus de l’hépatite B augmente considérablement leur risque de développer des maladies sévères du foie, notamment un hépatocarcinome. Or les médicaments actuels, malgré leur efficacité pour maintenir l’infection sous contrôle, ne permettent pas d’éliminer complètement le virus à cause de la persistance d’un minichromosome viral : l’ADN circulaire clos de façon covalente ou ADNccc. Une fois l’ADNccc établi dans le noyau des hépatocytes infectés, cet épisome est « chromatinisé », et il constitue alors la matrice unique pour la transcription des ARN viraux par l’ARN polymérase II de la cellule hôte. L’un de ces ARN viraux, l’ARN pré-génomique, est rétro-transcrit en ADN et encapsidé pour former de nouvelles particules virales infectieuses (Figure 1B) [6]. La phase nucléaire du cycle viral est donc essentielle à la réplication du virus de l’hépatite B, et constitue une cible prometteuse pour le développement de nouvelles thérapies visant à guérir les patients de l’infection chronique. Pour tenter d’atteindre cet objectif, il importe de mieux comprendre le contrôle de l’activité transcriptionnelle de l’ADNccc.
Une étude paléogénomique a montré que, comparativement aux génomes ancestraux, les génomes actuels du virus de l’hépatite B présentent une plus grande proportion de structures G4, qui se rapproche ainsi de la proportion dans le génome humain [7]. Ce mimétisme pourrait faciliter la prise en charge de l’ADNccc par la machinerie transcriptionnelle de l’hôte afin de faciliter la réplication du virus. En particulier, deux structures G4 ont été identifiées dans des régions régulatrices de l’ADNccc, sans toutefois établir de lien fonctionnel avec la réplication virale. D’autres travaux de recherche ont montré l’importance de la localisation nucléaire de l’ADNccc pour son activité transcriptionnelle, suggérant, sans pour autant le démontrer, que ce minichromosome pourrait entrer en séparation de phase [8]. En revanche, le lien entre la présence des structures G4 et cette localisation nucléaire de l’ADNccc n’a jamais été établi.
Deux structures G4 situées dans le premier enhancer3 de l’ADNccc favorisent la réplication du virus de l’hépatite B
Nous avons donc cherché à déterminer l’importance de structures G4 de l’ADNccc dans son activité transcriptionnelle et dans la réplication virale. Une analyse in silico de la séquence nucléotidique d’ADNccc du génotype D (majoritaire en Europe) a permis de prédire la présence de douze séquences (nommées PQS1-12) pouvant former des structures G4. Des expériences de dichroïsme circulaire4 ont confirmé que dix de ces séquences forment en effet de telles structures in vitro. Notre intérêt s’est alors porté sur les structures PQS4 et PQS5, qui se situent dans le premier enhancer (enhancer I) de l’ADNccc, une région régulatrice indispensable à la synthèse de l’ensemble des ARN viraux. Des cellules HepG2-NTCP (un modèle cellulaire couramment utilisé pour étudier la réplication du virus de l’hépatite B) ont été transfectées avec des ADNccc portant des mutations dans PQS4 ou PQS5 qui empêchent la formation de structures G4 in vitro, et nous avons comparé la capacité de ces ADNccc mutés à induire une réplication virale avec celle d’ADNccc non muté. La quantification des antigènes viraux sécrétés (par la technique ELISA) et des formes encapsidées d’ADN viraux ainsi que des ARN viraux (par la technique de RT-PCR quantitative) a montré que la mutation de PQS4 ou de PSQ5 s’accompagne d’une diminution de toutes les valeurs mesurées, ce qui témoigne de la contribution de ces deux structures G4 dans la réplication du virus de l’hépatite B. Nous sommes ensuite parvenus à établir que PQS4 et PQS5 activent la transcription de l’ADNccc. En particulier, des expériences d’immunoprécipitation de la chromatine ont montré que la présence de mutations dans PQS4 et PQS5 diminue le recrutement de l’ARN polymérase II par l’ADNccc, et entraîne un changement de marques d’histones au profit de marques répressives de la transcription [9].
L’ADNccc entre en « séparation de phase » de manière dépendante des structures G4
Parmi les protéines recrutées par les structures G4 et susceptibles de s’oligomériser et de former des condensats selon un processus de séparation de phase, figure la protéine FUS, qui reconnaît ces structures grâce à son domaine RGG3 (arginine-glycine-glycine). Cette protéine est impliquée dans le contrôle de l’expression génique notamment par la formation de condensats nucléaires favorisant la transcription via son domaine « prion-like » [10]. Nous avons émis l’hypothèse que PQS4 et PQS5 activent la transcription de l’ADNccc en recrutant la protéine FUS et en favorisant l’entrée de l’ADNccc dans des condensats contenant cette protéine. Plusieurs expériences dans les cellules HepG2-NTCP infectées par le virus de l’hépatite B ont permis de montrer que la protéine FUS est effectivement recrutée par l’ADNccc et induit son activité transcriptionnelle de manière dépendante de la capacité de FUS à se lier aux structures G4, particulièrement à PQS4 et PQS5. D’autre part, une analyse des condensats nucléaires contenant FUS a mis en évidence la présence de l’ADNccc dans ces condensats, de manière dépendante de la liaison de FUS aux G4, particulièrement à PQS5 [9]. Ces résultats apportent donc des arguments convergents en faveur de l’importance fonctionnelle de la séparation de phase de l’ADNccc pour son activité transcriptionnelle. En accord avec ce postulat, une protéine FUS défectueuse quant à sa capacité à entrer en séparation de phase est, contrairement à la protéine FUS non mutée, incapable d’activer la transcription de l’ADNccc [9].
Nous proposons donc un modèle selon lequel l’ADNccc, une fois établi dans le noyau des hépatocytes infectés par le virus de l’hépatite B, s'incorpore à des condensats nucléaires formés par la séparation de phase de la protéine FUS grâce à des structures G4 localisées dans la région régulatrice enhancer I, ce qui, en favorisant une conformation ouverte de la chromatine de l’ADNccc, permet sa transcription et la réplication du virus (Figure 2) [9]. Ce modèle ouvre la voie au développement de nouvelles thérapies ciblant les structures G4 afin d’obtenir une guérison fonctionnelle des patients atteints d’hépatite B chronique.
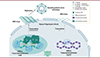 |
Figure 2. Les structures G4 de l’ADNccc viral favorisent son inclusion dans des condensats nucléaires contenant la protéine FUS et sa transcription, donc la réplication active du virus. L’ADNccc, une fois établi dans le noyau des hépatocytes infectés, entre « en séparation de phase » dans des condensats nucléaires contenant la protéine FUS, ce qui implique les structures G4 qu’il contient. Cela maintient une conformation chromatinienne permissive pour sa transcription et favorise la production de nouveaux virions. Cette figure a été créée avec le logiciel Biorender (www.biorender.com). |
Liens d’intérêt
B.T. a reçu des fonds de recherche de l’Inserm de la part de Assembly Biosciences, Beam Therapeutics et Janssen.
Remerciements
Ces travaux de recherche ont été soutenus par le Labex « DevWeCan » (ANR-10-LABX-61) et un financement public de l’Agence nationale de la recherche sur le SIDA et les hépatites virales – Maladies infectieuses émergentes (ANRS-MIE ECTZ134645). G.G. a bénéficié de financements publics de l’ARNS-MIE (ECTZ161842) et de l’Agence nationale de la recherche (ANR-17-RHUS-0003). Les auteurs souhaitent remercier l’ensemble des personnes ayant contribué activement à ces travaux de recherche ainsi que l’ensemble des membres de l’Inserm U1052 pour les discussions fructueuses ayant permis la finalisation de ce projet.
L’appariement de Hoogsteen est un type d’appariement canonique dans les acides nucléiques, impliquant les purines. Les liaisons hydrogène dans les paires de Hoogsteen impliquent une face de la purine différente de la face « Watson-Crick », et en particulier les positions N7 et le groupement oxo ou amino en position 6. Les quadruplexes de G (ou tétrades de G) sont des structures à quatre brins contenant des guanosines (G). Les interactions entre les quatre brins se font par des appariements de type Hoogsteen entre quatre G situées dans le même plan. Cette structure est stabilisée par la présence d’un cation monovalent au centre de la tétrade.
Références
- Huppert JL. Four-stranded nucleic acids : structure, function and targeting of G-quadruplexes. Chem Soc Rev 2008 ; 37 : 1 375–384. [Google Scholar]
- Chambers VS, Marsico G, Boutell JM, et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol 2015 ; 33 : 877–881. [CrossRef] [PubMed] [Google Scholar]
- Siddiqui-Jain A, Grand CL, Bearss DJ, et al. Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription. Proc Natl Acad Sci USA 2002 ; 99 : 11 593–598. [Google Scholar]
- Saranathan N, Vivekanandan P. G-quadruplexes : More than just a kink in microbial genomes. Trends Microbiol 2019 ; 27 : 148–163. [CrossRef] [PubMed] [Google Scholar]
- Bohálová N, Cantara A, Bartas M, et al. Analyses of viral genomes for G-quadruplex forming sequences reveal their correlation with the type of infection. Biochimie 2021; 186 : 13–27. [CrossRef] [PubMed] [Google Scholar]
- Zhang X, Wang Y, Yang G. Research progress in hepatitis B virus covalently closed circular DNA. Cancer Biol Med 2021; 19 : 415–31. [CrossRef] [PubMed] [Google Scholar]
- Brázda V, Dobrovolná M, Bohálová N, et al. G-quadruplexes in the evolution of hepatitis B virus. Nucleic Acids Res 2023; 51 : 7 198–204. [Google Scholar]
- Dias JD, Sarica N, Cournac A, et al. Crosstalk between hepatitis B virus and the 3D genome structure. Viruses 2022; 14 : 445. [CrossRef] [PubMed] [Google Scholar]
- Giraud G, Rodà M, Huchon P, et al. G-quadruplexes control hepatitis B virus replication by promoting cccDNA transcription and phase separation in hepatocytes. Nucleic Acids Res 2023 ; 52 : 2290–305. [Google Scholar]
- Reber S, Jutzi D, Lindsay H, et al. The phase separation-dependent FUS interactome reveals nuclear and cytoplasmic function of liquid-liquid phase separation. Nucleic Acids Res 2021 ; 49 : 7713–31. [Google Scholar]
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Importance fonctionnelle des structures G-quadruplex dans l’expression des gènes et dans la réplication virale : exemple du virus de l’hépatite B. A. Les G-quadruplex (G4) désignent des structures secondaires non canoniques des acides nucléiques se formant dans des régions riches en guanine (G), dont la séquence nucléotidique consensus est indiquée. Leur localisation préférentielle dans des régions régulatrices du génome et des ARN humains en font des acteurs importants du contrôle de l’expression génique, notamment de la transcription et de la traduction des transcrits en protéines. Les structures G4 favorisent la « séparation de phase » dans le noyau cellulaire via le recrutement de protéines capables de s’oligomériser, telles que la protéine FUS (elle s’oligomérise grâce à son domaine PLD et se lie aux structures G4 par son domaine RGG3), et de former ainsi des condensats nucléaires, qui sont impliqués dans différents processus biologiques comme la transcription. Ces structures G4 sont également présentes dans le génome de différents virus, dont ils contribuent à la réplication, comme nous le montrons ici pour le virus de l’hépatite B [9]. B. Le virus de l’hépatite B comporte une enveloppe et une capside contenant le génome viral : un ADN relâché circulaire partiellement double brin. Après l’entrée du virus dans les hépatocytes, la capside est importée dans le noyau et le génome viral est converti en « ADN circulaire clos de façon covalente » (ADNccc), un véritable épisome « chromatinisé ». Cet ADNccc est la matrice unique pour la synthèse, par l’ARN polymérase II cellulaire, des ARNm viraux, dont l’ARN pré-génomique, qui est ensuite rétro-transcrit et encapsidé pour produire de nouvelles particules virales infectieuses. Cette figure a été créée avec le logiciel Biorender (www.biorender.com). |
| Dans le texte | |
 |
Figure 2. Les structures G4 de l’ADNccc viral favorisent son inclusion dans des condensats nucléaires contenant la protéine FUS et sa transcription, donc la réplication active du virus. L’ADNccc, une fois établi dans le noyau des hépatocytes infectés, entre « en séparation de phase » dans des condensats nucléaires contenant la protéine FUS, ce qui implique les structures G4 qu’il contient. Cela maintient une conformation chromatinienne permissive pour sa transcription et favorise la production de nouveaux virions. Cette figure a été créée avec le logiciel Biorender (www.biorender.com). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




