| Issue |
Med Sci (Paris)
Volume 40, Number 4, Avril 2024
|
|
|---|---|---|
| Page(s) | 335 - 337 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2024034 | |
| Published online | 23 April 2024 | |
TMPRSS2 est le récepteur cellulaire du coronavirus saisonnier HKU1
TMPRSS2 is the receptor of seasonal coronavirus HKU1
Institut Pasteur, Université Paris Cité, CNRS UMR3569, Unité virus & immunité, Paris, France
À ce jour, sept coronavirus capables d’infecter les êtres humains ont été identifiés. Trois d’entre eux, SARS-CoV (identifié en 2003), MERS-CoV (identifié en 2012) et SARS-CoV-2 (identifié en 2019) sont hautement pathogènes, et ont provoqué trois récentes épidémies ou pandémies. Quant aux quatre coronavirus saisonniers endémiques, 229E, NL63, OC43 et HKU1, ils causent des maladies bénignes dans la majorité des cas (ils sont impliqués dans 15 à 30 % des rhumes), mais peuvent occasionnellement provoquer des infections sévères chez les nourrissons ou les personnes âgées [1].
Le coronavirus HKU1 a été isolé pour la première fois à Hong-Kong en 2005 chez une personne souffrant d’une pneumopathie [2]. Il fait partie du lignage A des β-coronavirus. Le coronavirus le plus proche de HKU1 du point de vue de l’évolution est le virus de l’hépatite murine (murine hepatitis virus, MHV) [2]. Les séquences d’acides aminés de la protéine du spicule1 de ces deux virus ont 65 % d’identité. La date d’émergence de HKU1, son origine animale et les potentiels hôtes intermédiaires sont inconnus. Le taux de séropositivité contre HKU1 est estimé entre 75 % et 95 % dans la population française, ce qui est comparable aux autres coronavirus saisonniers [3]. Il existe trois génotypes de HKU1 (A, B et C). HKU1C est issu d’une recombinaison entre HKU1A et HKU1B, et son spicule est quasiment identique à celui de HKU1B (99 % d’identité de séquence peptidique) [4], tandis que les spicules de HKU1A et HKU1B ont 85 % d’identité de séquence peptidique.
Le spicule (S) des coronavirus est nécessaire à leur entrée dans les cellules hôtes. La protéine S de HKU1 s’attache aux acides sialiques 9-O-acétylés de la membrane plasmique des cellules hôtes par son domaine N-terminal, ce qui permet une ouverture de la protéine et l’exposition du domaine de liaison à son récepteur (receptor-binding domain, RBD) [5, 6]. La structure de la protéine S dans ses conformations fermée et ouverte a été élucidée récemment [6]. L’état fermé protège notamment le RBD de potentiels anticorps neutralisants. Après la liaison à son récepteur, non identifié jusqu’à récemment, la protéine S peut être clivée par des protéases à sérine2, soit à la surface de la cellule par la protéase transmembranaire TMPRSS2 (type II transmembrane serine protease) ou la kallicréine KLK13, soit dans les endosomes par des cathepsines [7]. D’après les données disponibles pour le virus SARS-CoV-2 et d’autres coronavirus, ce clivage déclenche la projection du peptide de fusion et la fusion de la membrane virale avec la membrane cellulaire (plasmique ou endosomique), ce qui permet l’entrée du virus (Figure 1). La cellule infectée par un coronavirus produit différentes protéines virales, dont la protéine S, qu’elle expose à sa surface, et qui entraîne la fusion des cellules infectées avec les cellules avoisinantes, formant des cellules multinucléées (ou syncytium).
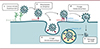 |
Figure 1. Schéma récapitulatif des deux voies d’entrée du coronavirus HKU1 dans la cellule hôte. Les étapes successives (numérotées de 1 à 4) de l’entrée des virions par la voie de surface ou par la voie des endosomes sont représentées. |
Nous avons récemment montré que TMPRSS2 est un récepteur membranaire fonctionnel pour HKU1A et HKU1B [8].
TMPRSS2 permet l’entrée de pseudovirus exprimant la protéine S de HKU1
L’expression de TMPRSS2 est suffisante pour induire la fusion de cellules exprimant le spicule de HKU1, et pour l’entrée de pseudovirus (lentivirus) exprimant ce spicule. Nous avons montré le rôle de TMPRSS2 dans différents types de cellules, dont celles des lignées HEK293T et U2OS3. Sur un ensemble de quinze protéases impliquées dans l’entrée des coronavirus ou de la même famille que TMPRSS2, seule TMPRSS2 permet la fusion membranaire impliquant la protéine S de HKU1. Les cellules de la lignée Caco24 expriment le gène TMPRSS2 de façon endogène et fusionnent spontanément lors de l’expression de la protéine S, mais la délétion de TMPRSS2 empêche la fusion. De plus, ces cellules sont sensibles à l’infection par des pseudovirus de HKU1, mais la délétion de TMPRSS2 empêche l’entrée de ces pseudovirus [8].
L’activité catalytique de TMPRSS2 n’est pas requise pour l’entrée de pseudovirus exprimant la protéine S de HKU1
TMPRSS2 est une protéase à sérine qui devient fonctionnelle par autoclivage [9]. Afin d’exclure la possibilité que le rôle de TMPRSS2 soit dû à son activité enzymatique de clivage de la protéine S et qu’une autre protéine cellulaire serve de récepteur à la protéine S, nous avons produit deux mutants enzymatiquement inactifs de TMPRSS2 : TMPRSS2Arg255Gln, mutée dans le site de clivage, et TMPRSS2Ser441Ala, mutée dans le site catalytique.
Le mutant catalytiquement inactif de TMPRSS2 ne clive pas la protéine S. Son effet sur la fusion cellulaire est réduit par rapport à TMPRSS2 non mutée. Néanmoins, tout comme celle-ci, il permet l’entrée des pseudovirus HKU1 [8]. En effet, le clivage de la protéine S à la surface est nécessaire pour la fusion cellulaire, mais le virus peut pénétrer dans la cellule via les endosomes, dans lesquels d’autres protéases ou l’environnement acide pourront induire la projection du peptide de fusion.
TMPRSS2 et la protéine S de HKU1 interagissent directement
Nous avons également montré l’existence d’une interaction directe entre la protéine S de HKU1 et TMPRSS2 par la technique ELISA. L’affinité de cette interaction a été calculée par interférométrie biocouche5, en utilisant le domaine RBD de la protéine S de HKU1 afin de s’affranchir de la nécessité de disposer de la protéine entière dans sa conformation ouverte. Nous avons mesuré des affinités (Kd) de 334 nM et de 137 nM respectivement pour HKU1A et HKU1B [8]. Ces valeurs sont plus élevées que celle obtenue pour SARS-CoV-2 et son récepteur ACE2 (enzyme de conversion de l’angiotensine 2) (Kd = 37 nM), mais elles restent de l’ordre de grandeur des interactions dites fortes.
Une étude structurale avait précédemment conclu à l’importance d’une région particulière du domaine RBD de la protéine S de HKU1, conservée entre HKU1A et HKU1B, dans la liaison à un récepteur alors inconnu, notamment en montrant que cette région était celle ciblée par des anticorps neutralisant HKU1 [10]. Nous avons montré que cette région était en effet impliquée dans la liaison à TMPRSS2 en mutant deux acides aminés, Trp515 et Arg517 : les deux protéines S mutées ne permettaient plus la fusion cellulaire ou l’entrée des pseudovirus, et avaient une affinité réduite ou non détectable pour TMPRSS2 [8].
L'interaction est bloquée par les nanocorps anti-TMPRSS2
Nous avons aussi isolé des nanocorps6 anti-TMPRSS2 après immunisation d’un alpaga. Trois d’entre eux bloquaient l’interaction de TMPRSS2 avec HKU1, ainsi que la fusion cellulaire et l’entrée des pseudovirus dans les cellules hôtes. Enfin, nous avons isolé du virus HKU1B infectieux à partir de prélèvements nasopharyngés d’une personne infectée, sur une culture primaire de cellules bronchiques différenciées en interface air-liquide. Ces cellules expriment TMPRSS2 sur leurs cils. L’infection de ces cellules par HKU1 était inhibée efficacement par l’un des nanocorps dirigés contre cette protéine [8].
Perspectives
L’ensemble des résultats présentés permet de conclure que TMPRSS2 est le récepteur du coronavirus HKU1 dans l’espèce humaine. Décrypter les voies d’entrée cellulaire des différents coronavirus permet de lutter plus efficacement contre l’infection par des thérapies ciblées. Cela permet également d’isoler et de caractériser plus rapidement de nouveaux coronavirus ou variants des coronavirus connus qui pourraient infecter l’homme. Les résultats de deux études concernant la structure du complexe formé par HKU1 et TMPRSS2 sont en cours de publication [11, 12]. Ils éclairent le mécanisme de l’entrée de HKU1, l’action d’anticorps neutralisants, et l’utilisation, par HKU1, de TMPRSS2 dans d’autres espèces animales qui pourraient être sensibles à l’infection par ce coronavirus. Par ailleurs, il conviendra d’examiner si TMPRSS2 joue également le rôle de récepteur pour d’autres virus, car cette protéase clive non seulement la protéine S des coronavirus, mais aussi les enveloppes d’autres virus respiratoires, tels que les virus influenza et para-influenza, les paramyxovirus, les métapneumovirus et le virus Sendai [13].
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Liu DX, Liang JQ, Fung TS. Human coronavirus-229E, -OC43, -NL63, and -HKU1 (coronaviridae). Encyclopedia of virology 2021; 428–40. [Google Scholar]
- Woo PCY, Lau SKP, Chu C, et al. Characterization and complete genome sequence of a novel coronavirus, coronavirus HKU1, from patients with pneumonia. J Virol 2005 ; 79 : 884–895. [CrossRef] [Google Scholar]
- De Thoisy A, Woudenberg T, Pelleau S, et al. Seroepidemiology of the seasonal human coronaviruses NL63, 229E, OC43 and HKU1 in France. Open Forum Infect Dis 2023; 10 : ofad340. [CrossRef] [PubMed] [Google Scholar]
- Woo PCY, Lau SKP, Yip CCY, et al. Comparative analysis of 22 coronavirus HKU1 genomes reveals a novel genotype and evidence of natural recombination in coronavirus HKU1. J Virol 2006 ; 80 : 7 136–145. [Google Scholar]
- Hulswit RJG, Lang Y, Bakkers MJG, et al. Human coronaviruses OC43 and HKU1 bind to 9-O-acetylated sialic acids via a conserved receptor-binding site in spike protein domain A. Proc Natl Acad Sci USA 2019 ; 116 : 2 681–690. [Google Scholar]
- Pronker MF, Creutznacher R, Drulyte I, et al. Sialoglycan binding triggers spike opening in a human coronavirus. Nature 2023; 624 : 201–6. [CrossRef] [PubMed] [Google Scholar]
- Shirato K, Kawase M, Matsuyama S. Wild-type human coronaviruses prefer cell-surface TMPRSS2 to endosomal cathepsins for cell entry. Virology 2018 ; 517 : 9–15. [CrossRef] [Google Scholar]
- Saunders N, Fernandez I, Planchais C, et al. TMPRSS2 is a functional receptor for human coronavirus HKU1. Nature 2023 ; 624 : 207–14. [CrossRef] [PubMed] [Google Scholar]
- Afar DEH, Vivanco I, Hubert RS, et al. Catalytic cleavage of the androgen-regulated TMPRSS2 protease results in its secretion by prostate and prostate cancer epithelia. Cancer Res 2001 ; 31 : 1 686–92. [Google Scholar]
- Ou X, Guan H, Qin B, et al. Crystal structure of the receptor binding domain of the spike glycoprotein of human betacoronavirus HKU1. Nat Commun 2017 ; 8 : 15 216. [CrossRef] [PubMed] [Google Scholar]
- McCallum M, Park YJ, Stewart C, et al. Human coronavirus HKU1 recognition of the TMPRSS2 host receptor. BioRxiv 2024,01.09.574 565. [PubMed] [Google Scholar]
- Fernández I, Saunders N, Duquerroy S, et al. Structural basis of TMPRSS2 zymogen activation and recognition by the HKU1 seasonal coronavirus. BioRxiv 2024,02.21.581378. [Google Scholar]
- Böttcher-Friebertshäuser E, Garten W, Klenk HD. Activation of viruses by host proteases. Springer Cham, 2018 : 348 p. [Google Scholar]
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Schéma récapitulatif des deux voies d’entrée du coronavirus HKU1 dans la cellule hôte. Les étapes successives (numérotées de 1 à 4) de l’entrée des virions par la voie de surface ou par la voie des endosomes sont représentées. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




