| Issue |
Med Sci (Paris)
Volume 34, Number 10, Octobre 2018
|
|
|---|---|---|
| Page(s) | 813 - 819 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2018215 | |
| Published online | 19 November 2018 | |
Le système CRISPR-Cas
Au-delà de l’édition génomique
The CRISPR-Cas system: beyond genome editing
1
Département de biochimie, de microbiologie, et de bio-informatique, faculté des sciences et de génie, groupe de recherche en écologie buccale, faculté de médecine dentaire, Université Laval, Québec, QC, G1V 0A6, Canada
2
Félix d’Hérelle reference center for bacterial viruses, faculté de médecine dentaire, Université Laval, 1045 avenue de la Médecine, Québec, QC, G1V 0A6, Canada
*
sylvain.moineau@bcm.ulaval.ca
CRISPR-Cas est un système immunitaire adaptatif utilisé par de nombreux microbes pour se défendre contre l’invasion d’acides nucléiques tels que les génomes viraux et autres éléments génétiques mobiles. Le système microbien utilise son locus CRISPR pour stocker de l’information génétique afin de produire des ARN guides. Ces derniers, de concert avec des endonucléases (Cas), empêchent des invasions futures. Des parties de ce système microbien ont été exploitées pour développer un puissant outil d’édition des génomes dans une panoplie d’organismes. La capacité de CRISPR-Cas9 à couper efficacement et à des endroits très précis de l’ADN pourrait peut-être permettre un jour de guérir certaines maladies génétiques humaines. La malléabilité de cet outil d’édition rend possible une variété d’applications allant de la modulation de l’expression de gènes à des modifications épigénétiques. Les locus CRISPR représentent également une mine d’informations pouvant servir de méthode de typage de souches microbiennes ou encore une façon d’étudier les interactions entre les bactéries et leurs habitats.
Abstract
CRISPR-Cas is an adaptive immune system used by many microbes to defend against nucleic acids invasion such as viral genomes. The microbial system uses its CRISPR locus to store genetic information that will generate short CRISPR RNAs. The latter with endonucleases (Cas) prevent future viral infections. Parts of this system were exploited to develop a powerful genome editing tool that was adapted for a variety of organisms. The ability of the CRISPR-Cas9 technology to effectively and precisely cut a targeted genomic DNA region has the potential to may be one day cure genetic diseases. The malleability of this editing tool also offers a wide range of possibilities from modulations of gene expression to epigenetic modifications. The natural CRISPR loci found in bacteria can be used to differentiate microbial strains or to study the interactions between bacteria and its habitat. Addressing CRISPR-Cas fundamentals in microbes and its popular use in eukaryotes, this review presents an update on a system that has revolutionized biological sciences.
© 2018 médecine/sciences – Inserm
Vignette (Photo © Inserm - Bertrand Jordan).
Au cours des dix années suivant la découverte de sa fonction biologique dans des bactéries [1], l’univers CRISPR a progressé à un rythme fulgurant qui a mené à une révolution dans l’ensemble des sciences biologiques.
En raison de sa grande spécificité et simplicité, de nombreux outils moléculaires ingénieux ont en effet été construits et leurs applications offrent maintenant de grands espoirs en thérapie génique mais aussi pour l’étude de problématiques environnementales. Abordant les principes fondamentaux du système, son utilisation populaire chez les eucaryotes et l’essor de ses applications écologiques, cette revue pose un regard actuel sur un sujet suscitant l’intérêt de la communauté scientifique mondiale.
Les bases du système CRISPR-Cas microbien
Le système CRISPR-Cas est constitué du locus CRISPR, de l’anglais clustered regularly interspaced short palindromic repeats, et de gènes cas (CRISPR-associated). Ce locus est composé de courtes séquences nucléotidiques répétées (appelées répétitions) intercalées de courtes séquences nucléotidiques variables et souvent uniques (appelées espaceurs, de l’anglais spacers). Cette structure génétique fut découverte pour la première fois dans le génome de la bactérie Escherichia coli en 1987 [2]. Sa fonction biologique resta inconnue pendant environ 20 ans [1].
L’analyse comparative de nombreux génomes bactériens a démontré la présence de systèmes CRISPR-Cas dans environ la moitié des génomes bactériens connus et près de 90 % des archées1. De tels systèmes sont absents chez les eucaryotes. La taille des courtes répétitions varie selon les espèces microbiennes, mais elles sont habituellement de 20 à 50 paires de base et leurs séquences peuvent partiellement former des structures palindromiques [3]. Ces répétitions flanquent des espaceurs de tailles comparables, mais dont les séquences nucléotidiques sont souvent propres à chaque souche microbienne [3]. En fait, ces séquences distinctes corrrespondent à des éléments génétiques mobiles comme les plasmides (ADN extrachromosomique capable de réplication autonome) ou des génomes de bactériophages (virus bactériens ou phages) [4]. Lorsque les séquences correspondantes sont présentes dans un contexte viral ou plasmidique, elles sont appelées « proto-espaceurs » (Figure 1).
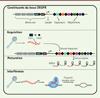 |
Figure 1. Éléments et fonctions de base du système CRISPR-Cas. Le système CRISPR-Cas est constitué des gènes cas (CRISPR-associated), du locus CRISPR (clustered regularly interspaced palindromic repeats), et d’une séquence leader. Le locus CRISPR est composé d’une alternance de courtes séquences nucléotidiques répétées intercalées entre des séquences variables. Lors de l’acquisition d’un nouvel espaceur, les protéines Cas1 et Cas2 dirigent l’ADN exogène vers l’extrémité du locus CRISPR où il sera intégré. Lors d’une infection subséquente, l’endonucléase du système, guidée par des ARNcr, induit une coupure double-brin dans l’ADN étranger. |
Bien que la composition des locus CRISPR soit très variable (à cause des espaceurs), ceux-ci sont fréquemment entourés de gènes codant des protéines nommées « Cas ». Le nom de ces dernières est suivi d’un chiffre révélant une similitude avec d’autres protéines ayant les mêmes fonctions. Le nombre, la combinaison et les fonctions de ces gènes/protéines ont permis de classer les systèmes CRISPR-Cas microbiens en deux grandes classes, incluant six types et de nombreux sous-types [5]. Ce sont les diverses protéines Cas qui sont principalement impliquées dans les différentes étapes du fonctionnement du système microbien.
La fonction des systèmes CRISPR-Cas microbiens, découverte en 2007 [1], ressemble à celle d’un système immunitaire, mais pour les procaryotes. En effet, le système CRISPR-Cas protège les bactéries contre des infections par des phages et des plasmides selon la séquence nucléotidique des espaceurs présents dans le locus CRISPR [1]. Cette protection est induite par la production de petits ARN CRISPR, les ARNcr. En effet, le locus CRISPR est transcrit en un long ARN qui subira ensuite une maturation pour générer des petits ARN, dont chacun contient une séquence complémentaire à une partie ou à l’entièreté d’une répétition et d’un espaceur [6] (Figure 1). Ces ARN non codants servent de guides en s’associant à une ou plusieurs protéines Cas (selon le type de système CRISPR-Cas) et agissent comme complexes de surveillance à l’intérieur des micro-organismes en attente d’une infection ou d’une invasion par un acide nucléique étranger [7]. L’ARNcr reconnaîtra la séquence envahissante à l’aide d’un appariement complémentaire, et la ou les protéines Cas (encore une fois selon le type de système) pourront ensuite couper le matériel génétique envahisseur, protégeant ainsi la cellule [3, 8].
Le système CRISPR-Cas est un système immunitaire adaptatif
Une des particularités du système CRISPR-Cas, qui le différencie des autres mécanismes de défense contre les virus, est sa capacité à acquérir de nouveaux espaceurs (un système d’adaptation) et par le fait même de s’immuniser contre d’éventuelles infections. Ce processus adaptatif se produit naturellement, grâce entre autres aux protéines Cas1 et Cas2 qui sont largement conservées dans les différents types de systèmes CRISPR-Cas. Ces protéines agissent ensemble afin d’induire l’acquisition de nouveaux espaceurs dans le locus CRISPR à partir de l’ADN étranger [9]. Cette acquisition n’est toutefois pas toujours aléatoire. Pour les systèmes de types I et II, de courts motifs (quelques nucléotides) adjacents aux proto-espaceurs, les PAM (protospacer adjacent motifs), ciblent les régions qui sont susceptibles d’être intégrées dans le locus CRISPR [10,11]. Ces motifs PAM, qui peuvent être différents selon le système CRISPR-Cas, seront également essentiels pour la coupure de l’ADN lors de l’étape d’interférence.
Récemment, il a été démontré que l’étape d’adaptation peut se concrétiser plus efficacement lorsque la cellule bactérienne est infectée par des virus défectueux. En effet, lorsque les bactéries sont exposées à des phages inactivés, la génération de bactéries résistantes à ces virus, grâce au système CRISPR-Cas, augmente proportionnellement [12]. Ces virus défectueux créent en fait un environnement dans lequel l’ADN étranger peut entrer dans la cellule sans en causer la mort (par opposition à une infection virale normale), lui donnant ainsi tout le temps nécessaire à l’acquisition de nouveaux espaceurs. Cette étape rappelle étonnamment le processus de vaccination des humains !
La boite à outils CRISPR-Cas
Le système CRISPR-Cas microbien est tellement versatile et relativement facile d’utilisation qu’il est désormais possible d’en faire un outil pédagogique dans un contexte d’enseignement universitaire [13]. La polyvalence de ce système est aussi illustrée par sa capacité à être programmé afin de cibler pratiquement n’importe quelle région d’ADN génomique [14]. Il est également possible d’adapter certaines protéines Cas pour des applications n’impliquant pas la coupure de l’ADN. Les modifications sont souvent réalisées sur l’endonucléase Cas9 du système de type II-A, l’endonucléase la plus couramment utilisée pour l’édition de génomes [15]. Par exemple, une version de la protéine exempte d’activité catalytique, dCas9 (dead Cas9), a été générée afin d’empêcher l’expression de certains gènes [16]. Cet outil, nommé CRISPRi (CRISPR interference), utilise un ARN guide afin de placer dCas9 sur un gène cible ou sur son promoteur. Une fois fixé, ce complexe empêche l’attachement de facteurs de transcription par interférence stérique et permet de bloquer la transcription du gène ciblé.
D’autres éléments peuvent être jumelés à dCas9 et servir de sonde à ADN pour accomplir une activité différente. Par exemple, un outil a été conçu pour étudier la régulation de l’expression de gènes, cette fois fondée sur les modifications épigénétiques chez les eucaryotes. Dans ce cas, dCas9 a été fusionnée à l’enzyme acétyltransférase, créant ainsi un système d’édition épigénétique reposant sur des ARN guides (Figure 2) [17]. Cette protéine de fusion permet l’acétylation des histones associées à certaines séquences d’ADN, induisant le relâchement de la chromatine et permettant ainsi la transcription des régions adjacentes. Ce type de complexe permet l’étude de l’expression des gènes à un niveau jamais atteint auparavant.
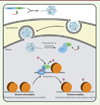 |
Figure 2. La protéine de fusion dCas9-HAT module l’expression des gènes par modifications épigénétiques. La protéine de fusion composée de la version inactive de l’endonucléase Cas9 (dCas9) et d’une histone acétyltransférase (HAT) est injectée dans le noyau au moyen d’un vecteur adénovirus associé (AAV). Cela entraîne l’acétylation localisée des histones. Les histones adjacentes à la séquence ciblée par les ARNg sont acétylées, ce qui relâche la chromatine et permet la transcription des séquences ciblées. |
Le système CRISPR-Cas, lorsque programmé, peut également servir d’outil pour l’immunisation bactérienne [14]. Yosef et al. ont ainsi récemment démontré une preuve de concept pour une stratégie génétique qui vise à sensibiliser les bactéries aux antibiotiques et à tuer sélectivement celles qui sont résistantes. Pour cela, un système CRISPR-Cas fonctionnel a été inséré dans le génome des bactéries (via un phage tempéré qui ne tue pas la bactérie), le CRISPR comprenant des espaceurs ciblant des gènes de résistance aux antibiotiques ainsi que le génome d’un phage virulent [18]. Ce système peut donc couper d’éventuels gènes de résistances aux antibiotiques acquis par la bactérie et, puisqu’il confère aussi une résistance au phage, il peut être utilisé comme agent de sélection afin de conserver uniquement les bactéries ayant intégré le système CRISPR-Cas. Cet ingénieux procédé pourrait permettre de réduire la fréquence de (multi)résistances aux antibiotiques dans une population bactérienne.
CRISPR : outil d’édition de génomes eucaryotes
Un outil d’édition de génomes se doit d’être simple, versatile mais surtout, précis. Le système CRISPR-Cas offre une telle précision. Ce sont les systèmes de types II-A qui ont retenu l’attention des chercheurs en raison de leur simplicité. En effet, seules trois composantes sont nécessaires pour la coupure de l’ADN. Comme nous l’avons déjà vu, l’endonucléase Cas9 et l’ARNcr sont indispensables. L’outil original d’édition de génomes est dérivé de la bactérie Streptococcus pyogenes et utilise SpCas9, la protéine Cas9 de cette dernière [19]. Dans le système CRISPR-Cas de S. pyogenes, un second ARN, l’ARNtracr, est également nécessaire à la précision et à la coupure de l’ADN. Cet ARNtracr est en partie complémentaire des séquences répétées du locus CRISPR et participe à la maturation du long ARN en petits ARNcr et à la prise en charge par Cas9. Toutefois pour l’édition de génome, l’ARNcr a été fusionné à l’ARNtracr afin de former un ARN guide unique (ARNg) qui remplit les fonctions de ces deux composants [15].
Dans un contexte d’édition de génome, une fois l’ADN double brin ciblé par l’ARNg coupé par Cas9, la cellule doit procéder à la réparation de son génome en utilisant ses systèmes de réparation. Cette réparation du génome est essentielle, la coupure pouvant être fatale à la cellule. La cellule peut utiliser un système de réparation appelé jonction d’extrémités non homologues (en anglais non-homologous end-joining ou NHEJ). Ce mécanisme ne restaure pas toujours la séquence initiale de l’ADN, mais il assure la continuité du génome endommagé. Cette réparation conduira fréquemment à l’apparition d’une mutation du gène ciblé et souvent à son inactivation. Un autre mécanisme de réparation, fondé sur la recombinaison homologue, permet aussi de réparer une coupure d’ADN double brin. Dans ce cas, il s’agit d’introduire en parallèle un gabarit de réparation, un fragment d’ADN qui contient des extrémités identiques aux deux portions du génome à refermer, mais qui contient aussi la modification à introduire. C’est de cette façon que l’on peut forcer la réparation selon notre intérêt et ainsi, par exemple, corriger une mutation.
Afin d’agir dans différents types de cellules, ces composants peuvent être injectés aux cellules eucaryotes à l’aide de divers vecteurs comme des virus adénoassociés (AAV) ou des nanoparticules lipidiques [20]. De nombreuses avancées ont été réalisées afin de réduire le nombre et la taille des éléments génétiques à introduire dans ces vecteurs. Les protéines Cas9 hétérologues présentent ainsi un fort potentiel. CjCas9 (Cas9 de Campylobacter jejuni) est active dans des cellules murines et humaines, et le gène qui la code est composé de 2,95 kpb, ce qui est beaucoup plus petit que les 4,1 kpb codant SpCas9, la Cas9 de S. pyogenes. Cette petite taille permet ainsi à CjCas9 d’être introduite dans un vecteur AAV tout en laissant de l’espace pour des éléments supplémentaires tels qu’un gabarit de recombinaison ou un ARNg [21].
Le traitement de maladies génétiques avec le système CRISPR-Cas
Certaines maladies génétiques sont dues à une mutation unique dans un gène modifiant la fonction d’une seule protéine. Elles sont les cibles principales des traitements envisagés à l’aide de l’édition de génome. Un exemple de ces maladies est la dystrophie musculaire de Duchenne, qui est à l’origine d’une dégénérescence musculaire causée par une mutation dans le gène codant pour la dystrophine. Cette protéine assure l’architecture cellulaire, particulièrement au niveau des fibres musculaires. Les mutations causant cette maladie sont souvent des délétions, ce qui modifie le cadre de lecture du gène et aboutit à une protéine non fonctionnelle [22,23]. Des stratégies de corrections utilisant le système CRISPR-Cas9 (Figure 3) ont été testées dans des modèles murins avec pour résultats, dans certains cas, une augmentation des niveaux d’expression de la dystrophine fonctionnelle suffisante pour observer une réduction significative des symptômes de la maladie [24]. Le système CRISPR-Cas9 a aussi été testé dans un modèle murin afin de modifier un gène associé à la dégénérescence maculaire liée à l’âge (DMLA) [21].
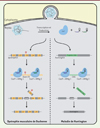 |
Figure 3. Délétion des mutations causales de la dystrophie musculaire de Duchenne et de la maladie de Huntington à l’aide du système CRISPR-Cas9. La mutation causant la dystrophie musculaire de Duchenne dans le modèle murin utilisé se situe dans l’exon 23. En ciblant à l’aide de deux ARNg les régions flanquant cet exon, celui-ci est excisé du génome et la coupure double-brin est réparée par jonction d’extrémités non homologues (NHEJ), créant une protéine tronquée mais fonctionnelle. Les mêmes étapes peuvent être utilisées dans un modèle de maladie de Huntington en utilisant des ARNg spécifiques des régions flanquant la région des triplets CAG dans le gène huntingtin qui cause la maladie. |
La maladie de Huntington2 a récemment été traitée chez la souris en utilisant le système CRISPR-Cas9 [25]. La portion du gène responsable de la maladie contient des répétitions du triplet CAG qui, lorsqu’elles sont présentes en trop grand nombre, rendent la protéine toxique. Une portion de cette région a pu être excisée à l’aide de SpCas9 et de plusieurs ARNg ciblant les régions adjacentes du gène (Figure 3), rétablissant ainsi une protéine saine. La capacité du système à éliminer certaines régions du génome pourrait aussi être utilisée afin de traiter des infections causées par des rétrovirus qui s’insèrent dans le génome humain, comme le virus de l’immunodéficience humaine (VIH). Ce dernier a été éliminé avec succès des génomes dans plusieurs modèles animaux en utilisant un vecteur AAV contenant SpCas9 ainsi que quatre différents ARNg ciblant des régions distinctes du virus intégré [26].
Bien que les applications du système CRISPR-Cas soient multiples, les dangers d’activité hors cible (off target) restent bien présents [15, 19, 21]. En effet, malgré sa grande précision, une des plus grandes craintes associées à l’utilisation de cette technologie est qu’un fragment d’ADN autre que celui ciblé par l’ARNg puisse être coupé, introduisant ainsi une mutation non souhaitée (à la suite de la réparation de la coupure). De très nombreuses études ont été réalisées pour réduire les risques associés à ces coupures hors cibles, mais il reste du travail… Une des options viserait à contrôler le moment où Cas9 est active. La découverte très récente de protéines de bactériophages inhibant spécifiquement Cas9 [27, 28] vient ajouter à la boîte à outils CRISPR un élément qui était alors manquant : un bouton d’arrêt. Bien que le rôle biologique de ces protéines soit de permettre aux bactériophages de désarmer le système de défense CRISPR-Cas bactérien, elles pourraient peut-être aussi permettre, en thérapie génique, d’augmenter la fidélité et l’efficacité du système CRISPR-Cas9. Toutefois, avant de pouvoir utiliser le système CRISPR-Cas9 dans un réel contexte thérapeutique, des avancées permettant d’augmenter sa précision et de mieux contrôler son activité devront être réalisées.
Malgré le fait que le système CRISPR-Cas ne soit pas encore prêt à être utilisé dans un contexte de thérapie germinale chez l’homme, il a récemment été mis à l’épreuve au niveau cellulaire. Des essais cliniques ont récemment été annoncés en Chine [29] et aux États-Unis [30]. Ces études, débutées en 2018, utilisent des cellules immunitaires (les lymphocytes T) modifiées à l’aide du système CRISPR-Cas9 pour cibler plus efficacement les cancers dont souffrent les patients. Ce type de modifications n’implique toutefois que des cellules immunitaires isolées. Au niveau de thérapie germinale globale, une première tentative d’édition de zygotes humains a été réalisée en 2015 [31]. Les auteurs font état d’un succès mitigé : la coupure spécifique de l’ADN est possible, mais la réparation par recombinaison homologue n’est pas observée dans toutes les cellules, produisant une mosaïque de cellules génétiquement différentes. Ces résultats démontrent qu’il reste encore beaucoup à faire avant d’approuver des techniques de thérapie germinale.
Applications écologiques de CRISPR
Bien que le système CRISPR-Cas possède une multitude d’applications grâce à ses capacités endonucléasiques, la séquence naturelle du locus CRISPR chez divers micro-organismes représente une mine d’informations pour des études en écologie microbienne. En effet, la diversité des espaceurs d’un locus CRISPR est phénoménale. Une étude échantillonnant divers microbiotes humains (peau, bouche, etc.) a ainsi démontré que les contenus en espaceurs de chaque site étaient tous très différents les uns des autres. Les espaceurs recensés lors de cette étude ne correspondent presque jamais à des séquences présentes dans les bases de données publiques de séquences nucléotidiques [32]. Ces résultats donnent un aperçu de la diversité jusqu’à présent à peine explorée des plasmides et des phages de l’environnement. Puisque les nouveaux espaceurs sont souvent ajoutés en 5’ du locus CRISPR, ces nouvelles séquences donnent un caractère temporel recensant l’historique des acquisitions d’espaceurs, les plus anciens étant généralement situés dans la portion 3’ du locus [9,10].
En considérant le locus CRISPR comme une série d’espaceurs de séquences nucléotidiques différentes, on peut donc les utiliser afin de distinguer des souches bactériennes (Figure 4). Le premier exemple de différenciation (CRISPR-typing) utilisant le locus CRISPR remonte à 1999 [33], avant même la découverte de sa fonction principale chez les bactéries. Cette méthode est maintenant combinée avec les méthodes traditionnelles afin de distinguer des souches bactériennes avec une très grande précision [34], ce qui a notamment été réalisé chez Salmonella [35, 36]. Ce pouvoir discriminant est d’un grand d’intérêt pour les épidémiologistes. L’organisation chronologique des séquences du locus CRISPR permet de créer une sorte d’« arbre généalogique » des souches bactériennes d’intérêt dans le but de retracer l’origine des épidémies. Cette technique a d’ailleurs été mise en application lors de la survenue brutale d’une épidémie de légionellose à Ulm, en Allemagne [37]. Les locus CRISPR des souches isolées de patients furent analysées et comparées à ceux de souches isolées de tours de refroidissement à travers la ville, permettant ainsi d’identifier l’origine de la contamination.
 |
Figure 4. Discrimination de plusieurs souches bactériennes en utilisant l’information du locus CRISPR. A. Les étapes microbiologiques et biochimiques incluent l’extraction de l’ADN des bactéries ciblées, l’amplification du locus CRISPR au moyen d’amorces spécifiques ainsi que le séquençage des produits de PCR (polymerase chain reaction) obtenus. B. Les étapes bio-informatiques incluent la reconnaissance du motif répétition-espaceur, l’extraction des séquences d’espaceurs individuels, ainsi que la transformation de ces dernières en types CRISPR selon la composition des locus de chaque souche analysée. |
Analyse des écosystèmes microbiens
Les microbiotes humains sont constitués d’une multitude d’organismes différents [38, 39]. Afin de comprendre ces écosystèmes et leurs impacts potentiels sur la santé humaine, il est important d’étudier leurs interactions. Le caractère temporel des locus CRISPR en fait d’excellentes archives moléculaires, une sorte de répertoire des interactions passées entre les bactéries et leur environnement. Une étude réalisée sur les locus CRISPR d’espèces de Streptococcus provenant de salives humaines a révélé deux particularités intéressantes quant à la « population » d’espaceurs. Un groupe d’espaceurs est en effet très conservé chez un individu au cours du temps, alors qu’un second est variable. Dans ce groupe variable, l’analyse des locus CRISPR a montré plusieurs acquisitions et délétions d’espaceurs. Il contient clairement un historique des interactions de ce genre bactérien avec divers éléments génétiques mobiles [39]. Ainsi, la diversité des espaceurs d’un écosystème peut aussi servir à l’identification d’éléments génétiques bactériens pouvant avoir un impact sur la santé. Beaucoup de gènes de résistance aux antibiotiques [40] et de facteurs de virulence [41] sont transmis horizontalement par des plasmides ou par des bactériophages [42]. Les génomes de souches bactériennes ne possédant pas de système CRISPR-Cas sont de taille plus grande et présentent davantage de gènes de résistance aux antibiotiques en regard des bactéries qui possèdent un système CRISPR-Cas [43].
Conclusion
Les connaissances sur les systèmes CRISPR-Cas ont évolué de façon exponentielle au cours des dernières années et les avancées spectaculaires quant à son utilisation comme outil génétique ont révolutionné les sciences biologiques [44] (→). Que ce soit pour caractériser des processus biologiques, pour traiter des maladies génétiques ou pour étudier des écosystèmes microbiens, le système CRISPR-Cas a déjà changé notre façon de faire de la recherche. La versatilité du système CRISPR-Cas ouvre de nombreuses perspectives en recherche fondamentale ou appliquée, incluant le domaine biomédical. La capacité du système CRISPR-Cas à cibler virtuellement n’importe quelle séquence du génome pourrait transformer radicalement les stratégies de traitement de certaines maladies géniques, en passant du contrôle des symptômes à une véritable guérison [45] (→). Lorsque combiné à des méthodes de contrôle de l’activité endonucléase et à de nouveaux vecteurs de transport, le système CRISPR-Cas offre un éventail de possibilités qui ne cessent de s’accroître. Si le passé est garant de l’avenir, les prochaines années devraient offrir de nouvelles applications de plus en plus performantes et stimulante.
(→) Voir la Nouvelle de H. Gilgenkrantz, m/s n° 12, décembre 2014, page 1066
(→) Voir la Chronique génomique de B. Jordan, m/s n° 11, novembre 2016, page 1035
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données pubiées dans cet article.
Les archées sont des organismes aussi différents des bactéries que des eucaryotes. Les enzymes réalisant la réplication de l’ADN, la transcription de l’ADN en ARN ainsi que la traduction de l’ARN messager en protéines chez les archées sont apparentées à celles des eucaryotes. De plus, la présence d’histones dans le matériel génétique des archées les rapproche des eucaryotes et les distingue des bactéries. Comme ceux des eucaryotes, les gènes des archées possèdent des introns et leurs ARN messagers subissent des modifications post-transcriptionnelles.
La maladie de Huntington est associée à la dégénérescence de neurones d’une partie du cerveau impliqués dans des fonctions motrices, cognitives et comportementales. Si le gène dont l’altération provoque la maladie est connu (gène codant une protéine nommée huntingtine), les mécanismes qui mènent à cette neurodégénérescence ne sont pas encore élucidés.
Références
- Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistanc against viruses in prokaryotes. Science 2007 ; 315 : 1709–1712. [CrossRef] [Google Scholar]
- Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol 1987 ; 169 : 5429–5433. [CrossRef] [PubMed] [Google Scholar]
- Barrangou R. CRISPR-Cas systems and RNA-guided interference. Wiley Interdiscip Rev RNA 2013 ; 4 : 267–278. [CrossRef] [PubMed] [Google Scholar]
- Mojica FJM, Díez-Villaseñor C, García-Martínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol 2005 ; 60 : 174–182. [CrossRef] [PubMed] [Google Scholar]
- Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems. Curr Opin Microbiol 2017 ; 37 : 67–78. [CrossRef] [PubMed] [Google Scholar]
- Brouns SJJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 2008 ; 321 : 960–964. [CrossRef] [Google Scholar]
- Tremblay JP. CRISPR, un système qui permet de corriger ou de modifier l’expression de gènes responsables de maladies héréditaires. Med Sci (Paris) 2015 ; 31 : 1014–1022. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Garneau JE, Dupuis ME, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature 2010 ; 468 : 67–71. [CrossRef] [PubMed] [Google Scholar]
- Nuñez JK, Lee ASY, Engelman A, et al. Integrase-mediated spacer acquisition during CRISPR–Cas adaptive immunity. Nature 2015 ; 519 : 193–198. [CrossRef] [PubMed] [Google Scholar]
- Deveau H, Barrangou R, Garneau JE, et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J Bacteriol 2008 ; 190 : 1390–1400. [CrossRef] [PubMed] [Google Scholar]
- Mojica FJM, Díez-Villaseñor C, García-Martínez J, et al. Short motif sequences determine the targets of the prokaryotic CRISPR defence system. Microbiology 2009 ; 155 : 733–740. [CrossRef] [PubMed] [Google Scholar]
- Hynes AP, Villion M, Moineau S. Adaptation in bacterial CRISPR-Cas immunity can be driven by defective phages. Nat Commun 2014 ; 5 : 467–477. [CrossRef] [Google Scholar]
- Hynes AP, Lemay M, Trudel L, et al. Detecting natural adaptation of the Streptococcus thermophilus CRISPR-Cas systems in research and classroom settings. Nat Protoc 2017 ; 12 : 547–565. [CrossRef] [PubMed] [Google Scholar]
- Hynes AP, Labrie SJ, Moineau S. Programming native CRISPR arrays for the generation of targeted immunity. MBio 2016 ; 7 : e00202–e00216. [CrossRef] [PubMed] [Google Scholar]
- Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science 2013 ; 339 : 823–826. [CrossRef] [PubMed] [Google Scholar]
- Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell 2013 ; 152 : 1173–1183. [CrossRef] [PubMed] [Google Scholar]
- Hilton IB, D’Ippolito AM, Vockley CM, et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol 2015 ; 33 : 510–517. [CrossRef] [PubMed] [Google Scholar]
- Yosef I, Manor M, Kiro R, et al. Temperate and lytic bacteriophages programmed to sensitize and kill antibiotic-resistant bacteria. Proc Natl Acad Sci USA 2015 ; 112 : 7267–7272. [CrossRef] [Google Scholar]
- Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science 2013 ; 339 : 819–823. [CrossRef] [PubMed] [Google Scholar]
- Yin H, Song CQ, Dorkin JR, et al. Therapeutic genome editing by combined viral and non-viral delivery of CRISPR system components in vivo. Nat Biotechnol 2016 ; 34 : 328–333. [CrossRef] [PubMed] [Google Scholar]
- Kim E, Koo T, Park SW, et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nat Commun 2017 ; 8 : 1–12. [CrossRef] [Google Scholar]
- Hoffman EP, Brown RH, Kunkel LM. Dystrophin: the protein product of the Duchenne muscular dystrophy locus. Cell 1987 ; 51 : 919–928. [CrossRef] [PubMed] [Google Scholar]
- Nelson CE, Hakim CH, Ousterout DG, et al. In vivo genome editing improves muscle function in a mouse model of Duchenne muscular dystrophy. Science 2016 ; 351 : 403–407. [CrossRef] [PubMed] [Google Scholar]
- Bengtsson NE, Hall JK, Odom GL, et al. Muscle-specific CRISPR/Cas9 dystrophin gene editing ameliorates pathophysiology in a mouse model for Duchenne muscular dystrophy. Nat Commun 2017 ; 8 : 14454. [CrossRef] [PubMed] [Google Scholar]
- Yang S, Chang R, Yang H, et al. CRISPR/Cas9-mediated gene editing ameliorates neurotoxicity in mouse model of Huntington’s disease. J Clin Invest 2017 ; 127 : 2719–2724. [CrossRef] [PubMed] [Google Scholar]
- Yin C, Zhang T, Qu X, et al. In Vivo Excision of HIV-1 provirus by saCas9 and multiplex single-guide RNAs in animal models. Mol Ther 2017 ; 25 : 1168–1186. [CrossRef] [PubMed] [Google Scholar]
- Rauch BJ, Silvis MR, Hultquist JF, et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell 2017 ; 168 : 150–158. [CrossRef] [PubMed] [Google Scholar]
- Hynes AP, Rousseau GM, Lemay ML, et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nat Microbiol 2017 ; 2 : 1374–1380. [CrossRef] [PubMed] [Google Scholar]
- Cyranoski D. CRISPR gene-editing tested in a person for the first time. Nature 2016 ; 539 : 479. [CrossRef] [PubMed] [Google Scholar]
- Reardon S. First CRISPR clinical trial gets green light from US panel. Nature 2016 ; 539 : 479. [CrossRef] [PubMed] [Google Scholar]
- Liang P, Xu Y, Zhang X, et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell 2015 ; 6 : 363–372. [CrossRef] [PubMed] [Google Scholar]
- Rho M, Wu YW, Tang H, et al. Diverse CRISPRs evolving in human microbiomes. PLoS Genet 2012 ; 8 : e1002441. [CrossRef] [PubMed] [Google Scholar]
- Hoe N, Nakashima K, Grigsby D, et al. Rapid molecular genetic subtyping of serotype M1 group A Streptococcus strains. Emerg Infect Dis 1999 ; 5 : 254–263. [CrossRef] [PubMed] [Google Scholar]
- Shariat N, Dudley EG. CRISPRs: molecular signatures used for pathogen subtyping. Appl Environ Microbiol 2014 ; 80 : 4309. [CrossRef] [Google Scholar]
- Shariat N, DiMarzio MJ, Yin S, et al. The combination of CRISPR-MVLST and PFGE provides increased discriminatory power for differentiating human clinical isolates of Salmonella enterica subsp. enterica serovar Enteritidis. Food Microbiol 2013 ; 34 : 164–173. [CrossRef] [Google Scholar]
- Zheng H, Hu Y, Li Q, et al. Subtyping Salmonella enterica serovar Derby with multilocus sequence typing (MLST) and clustered regularly interspaced short palindromic repeats (CRISPRs). Food Control 2017 ; 73 : 474–484. [CrossRef] [Google Scholar]
- Lück C, Brzuszkiewicz E, Rydzewski K, et al. Subtyping of the Legionella pneumophila “Ulm” outbreak strain using the CRISPR-Cas system. Int J Med Microbiol 2015 ; 305 : 828–837. [CrossRef] [PubMed] [Google Scholar]
- Turnbaugh PJ, Ley RE, Hamady M, et al. The human microbiome project. Nature 2007 ; 449 : 804–810. [CrossRef] [PubMed] [Google Scholar]
- Pride DT, Sun CL, Salzman J, et al. Analysis of streptococcal CRISPRs from human saliva reveals substantial sequence diversity within and between subjects over time. Genome Res 2011 ; 21 : 126–136. [CrossRef] [PubMed] [Google Scholar]
- Stokes HW, Gillings MR, CW C, et al. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens. FEMS Microbiol Rev 2011 ; 35 : 790–819. [CrossRef] [PubMed] [Google Scholar]
- Saunders JR, Allison H, James CE, et al. Phage-mediated transfer of virulence genes. J Chem Technol Biotechnol 2001 ; 76 : 662–666. [CrossRef] [Google Scholar]
- Minot S, Sinha R, Chen J, et al. The human gut virome: Inter-individual variation and dynamic response to diet. Genome Res 2011 ; 21 : 1616–1625. [CrossRef] [PubMed] [Google Scholar]
- Van Belkum A, Soriaga LB, LaFave MC, et al. Phylogenetic distribution of CRISPR-Cas systems in antibiotic-resistant Pseudomonas aeruginosa. MBio 2015 ; 6 : 1–13. [CrossRef] [Google Scholar]
- La Gilgenkrantz H. révolution des CRISPR est en marche. Med Sci (Paris) 2014 ; 30 : 1066–1069. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Jordan B. Les débuts de CRISPR en thérapie génique. Med Sci (Paris) 2016 ; 32 : 1035–1037. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Éléments et fonctions de base du système CRISPR-Cas. Le système CRISPR-Cas est constitué des gènes cas (CRISPR-associated), du locus CRISPR (clustered regularly interspaced palindromic repeats), et d’une séquence leader. Le locus CRISPR est composé d’une alternance de courtes séquences nucléotidiques répétées intercalées entre des séquences variables. Lors de l’acquisition d’un nouvel espaceur, les protéines Cas1 et Cas2 dirigent l’ADN exogène vers l’extrémité du locus CRISPR où il sera intégré. Lors d’une infection subséquente, l’endonucléase du système, guidée par des ARNcr, induit une coupure double-brin dans l’ADN étranger. |
| Dans le texte | |
 |
Figure 2. La protéine de fusion dCas9-HAT module l’expression des gènes par modifications épigénétiques. La protéine de fusion composée de la version inactive de l’endonucléase Cas9 (dCas9) et d’une histone acétyltransférase (HAT) est injectée dans le noyau au moyen d’un vecteur adénovirus associé (AAV). Cela entraîne l’acétylation localisée des histones. Les histones adjacentes à la séquence ciblée par les ARNg sont acétylées, ce qui relâche la chromatine et permet la transcription des séquences ciblées. |
| Dans le texte | |
 |
Figure 3. Délétion des mutations causales de la dystrophie musculaire de Duchenne et de la maladie de Huntington à l’aide du système CRISPR-Cas9. La mutation causant la dystrophie musculaire de Duchenne dans le modèle murin utilisé se situe dans l’exon 23. En ciblant à l’aide de deux ARNg les régions flanquant cet exon, celui-ci est excisé du génome et la coupure double-brin est réparée par jonction d’extrémités non homologues (NHEJ), créant une protéine tronquée mais fonctionnelle. Les mêmes étapes peuvent être utilisées dans un modèle de maladie de Huntington en utilisant des ARNg spécifiques des régions flanquant la région des triplets CAG dans le gène huntingtin qui cause la maladie. |
| Dans le texte | |
 |
Figure 4. Discrimination de plusieurs souches bactériennes en utilisant l’information du locus CRISPR. A. Les étapes microbiologiques et biochimiques incluent l’extraction de l’ADN des bactéries ciblées, l’amplification du locus CRISPR au moyen d’amorces spécifiques ainsi que le séquençage des produits de PCR (polymerase chain reaction) obtenus. B. Les étapes bio-informatiques incluent la reconnaissance du motif répétition-espaceur, l’extraction des séquences d’espaceurs individuels, ainsi que la transformation de ces dernières en types CRISPR selon la composition des locus de chaque souche analysée. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




