| Issue |
Med Sci (Paris)
Volume 30, Number 1, Janvier 2014
|
|
|---|---|---|
| Page(s) | 47 - 54 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20143001013 | |
| Published online | 24 January 2014 | |
Nouvelles fonctions de la peptide N-glycanase indépendantes de sa capacité de déglycosylation
Novel deglycosylation-independent roles for peptide N-glycanase
1
Inserm U773, centre de recherche Bichat Beaujon CRB3, Faculté de médecine Xavier Bichat, 75018 Paris, France
2
Université Paris 7 Denis Diderot, site Bichat, 16, rue Henri Huchard, 75018, Paris, France
La première fonction qui a été attribuée à la peptide N-glycanase (PNGase) est de déglycosyler les N-glycosylprotéines mal repliées avant leur dégradation par le protéasome. Il s’avère cependant que l’inhibition de cette enzyme modifie peu le taux de dégradation de ces N-glycosylprotéines. Des données récentes montrent que cette enzyme a également la capacité de moduler la morphogenèse de façon indépendante de sa fonction de déglycosylation. La caractérisation du premier déficit en PNGase devrait apporter une meilleure compréhension du rôle de cette enzyme multifonctionnelle dans la physiologie humaine.
Abstract
The primary function of peptide N-glycanase (PNGase) is thought to be the deglycosylation of endoplasmic reticulum associated degradation (ERAD) substrates. However, inhibition of PNGase appears to have little effect upon the destruction rate of many ERAD substrates, and recent data demonstrate deglycosylation-independent functions for PNGase. Whatever the roles of PNGase turn out to be, the identification of a patient presenting with PNGase deficiency will advance our understanding of the importance of this multifunctional protein in human physiology.
© 2014 médecine/sciences – Inserm
Chez les mammifères, la dégradation des N-glycosylprotéines se déroule dans le cytoplasme ou dans les lysosomes. La dégradation cytoplasmique concerne surtout des N-glycosylprotéines qui n’ont pu acquérir leur conformation tridimensionnelle après leur biosynthèse dans le réticulum endoplasmique (RE). En revanche, la dégradation lysosomale concerne des N-glycosylprotéines constitutives des organites intracellulaires ou provenant de la membrane plasmique et du milieu extérieur [1].
La dégradation cytoplasmique ou lysosomale des N-glycosylprotéines met en jeu deux types d’enzymes qui ont la capacité de débrancher les chaînes N-glycanes en clivant la liaison entre le premier résidu N-acétyl-glucosamine (GlcNAc) de l’oligosaccharide et l’asparagine (Asn) porteuse de la chaîne N-glycane (Figure 1A). Il s’agit, respectivement, de la peptide N-glycanase (PNGase, également appelée N-glycanase, glycoamidase ou peptide-N 4-[N-acétyl-β-D-glucosaminyl], EC3.5.1.52) et de l’aspartylglucosaminidase (AGA, également appelée glycosylasparaginase ou N 4-[β-N-acétylglucosaminyl]-1-asparaginase, EC 3.5.1.26).
 |
Figure 1. Les différentes enzymes capables de déglycosyler les N-glycosylprotéines et le rôle de la PNGase pendant le processus ERAD-L. A. Deux activités enzymatiques intracellulaires majeures sont capables de débrancher les chaînes N-glycanes en clivant la liaison entre le premier résidu de N-acétylglucosamine (GlcNAc) de l’oligosaccharide et l’asparagine (Asn) porteuse de la chaîne N-glycane. Au niveau du cytoplasme, une des activités de déglycosylation est portée par la peptide N-glycanase (PNGase), qui génère de façon concomitante un oligosaccharide libre (fOS) et la conversion du résidu Asn porteur de la chaîne N-glycane en résidu aspartate (Asp, panneau de gauche). Dans les lysosomes, une autre activité est portée par la N 4-(β-N-acétylglucosaminyl)-1-asparaginase qui ne peut agir que si la dégradation de la partie peptidique de la glycoprotéine a eu lieu au préalable (panneau de droite). Certaines données suggèrent également qu’il existerait une activité PNGase à l’intérieur même du réticulum endoplasmique (RE), mais bien que cette activité soit sensible à l’inhibiteur Z-VAD-fmk, la protéine responsable n’a pas encore été caractérisée [32, 33]. À ces activités de déglycosylation, il faut ajouter l’endo-β-N-acétylglucosaminidase cytosolique (ENGase) qui est également capable d’hydrolyser les chaînes N-liées en clivant la liaison entre les deux résidus de N-acétylglucosamine (GlcNAc) de l’oligosaccharide (panneau du milieu), et qui pourrait prendre le relais de la PNGase. B. Le processus ERAD est une machinerie moléculaire complexe ; elle permet de repérer les N-glycosylprotéines qui restent mal repliées après leur biosynthèse dans le RE, puis de les dégrader via le protéasome. Ce processus a été conservé au cours de l’évolution et les connaissances qui ont été établies à partir du modèle de la levure sont applicables aux eucaryotes supérieurs, avec toutefois un niveau de complexité plus élevé. Trois voies ERAD ont été décrites qui reconnaissent des domaines peptidiques mal repliés localisés, soit dans le cytosol (ERAD-C), soit dans la membrane (ERAD-M), soit dans la lumière du RE (ERAD-L). N’est représentée sur le schéma que la voie ERAD-L chez S. cerevisiae. Cette voie ERAD-L est la mieux connue et repose sur trois propriétés : la reconnaissance de la région peptidique, qui reste mal repliée, par l’apparition de régions hydrophobes accessibles ; la modification de la structure oligosaccharidique par l’action successive de différentes glycosidases (Mns1p [mannosidase réticulaire I] et le complexe Mnl1p/Htm1p-Pdi1p) ; et la reconnaissance de la structure oligosaccharidique Man7GlcNAc2 qui en résulte comme signal de dégradation. Cette structure comporte un résidu mannose en α1,6, démasqué sur la branche C de l’oligosaccharide et qui est reconnu spécifiquement par la lectine Yos9p/Os9p. Des complexes transmembranaires protéiques organisés autour de différentes E3 ubiquitine ligases (Hrd1p, Hrd3p, et Der1p) jouent un rôle stratégique, car ils coordonnent les différents évènements qui ont lieu de part et d’autre de la membrane du RE, tels que l’ubiquitination des N-glycosylprotéines mal repliées et leur rétrotranslocation vers le cytosol. Les substrats ubiquitinés sont alors déglycosylés par la PNGase cytosolique avant leur dégradation par le protéasome [22]. Les fOS, qui sont issus de l’hydrolyse des chaînes N-glycanes, sont eux-mêmes soumis à une voie métabolique : chez la levure, ils sont démannosylés de façon progressive par une mannosidase cytosolique codée par le gène Ams1 jusqu’à la structure ne comportant plus que deux résidus de GlcNAc et un résidu de mannose [23]. |
L’aspartylglucosaminurie, une maladie autosomique récessive décrite dès 1968, résulte d’un déficit en activité aspartylglucosaminidase. C’est la maladie de stockage lysosomal la plus fréquente, et elle se manifeste par une accumulation de petits glycopeptides dans les lysosomes. Les symptômes cliniques sont un retard psychomoteur progressif, ainsi que des anomalies du squelette et du visage. De très nombreuses mutations du gène AGA codant pour cette hydrolase lysosomale ont été caractérisées [2]. La première description d’un patient porteur d’un déficit en PNGase ne date, elle, que de 2012 [3]. Les symptômes de ce patient peuvent être la conséquence d’un dysfonctionnement, soit de l’activité d’hydrolyse de la chaîne N-glycane, soit de fonctions nouvellement décrites qui sont indépendantes de l’activité de déglycosylation de l’enzyme.
Dans cette revue, seront donc successivement exposées les données conséquentes concernant le rôle de la PNGase dans la dégradation des N-glycosylprotéines mal repliées, puis les données plus restreintes concernant les autres fonctions potentielles de cette enzyme.
Principales caractéristiques de la PNGase
Une activité PNGase s’exerçant sur une N-glycosylprotéine produit de façon concomitante un oligosaccharide libre (fOS pour free oligosaccharide) et la conversion du résidu asparagine porteur de la chaîne N-glycane en résidu aspartate (Asp) (Figure 1A). Bien que des activités PNGase aient été trouvées depuis de nombreuses années chez les plantes et les bactéries, la découverte d’activités PNGase cytosoliques chez les mammifères [4], puis chez la levure Saccharomyces cerevisiae [5], est plus récente. Les caractéristiques biochimiques de ces dernières s’avéraient différentes des premières activités PNGase rapportées. Le clonage du premier gène (ScPNG1) codant pour cette enzyme a pu être réalisé chez S. cerevisiae [6]. L’existence d’orthologues de ce gène dans de nombreuses espèces, y compris chez les mammifères, a permis d’établir que ces enzymes représentent une nouvelle classe de PNGase qui est conservée au cours de l’évolution.
Structure, expression et localisation subcellulaire
ScPNG1 code pour une protéine soluble (ScPng1p) de 363 acides aminés [6] : le domaine central transglutaminase-like et les deux motifs thioredoxin-like CXXC constituent la structure commune à toutes les espèces (Figure 2). La localisation cytosolique et nucléaire de ScPng1p se superpose à celle du protéasome [6].
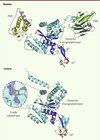 |
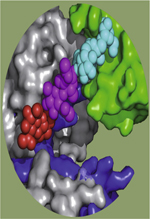
Figure 2. Comparaison des structures tridimensionnelles des PNGases de levure et humaine. La PNGase de Saccharomyces cerevisiae (ScPng1p) est codée par le gène PNG1 : le domaine central transglutaminase, qui comporte le site actif (en bleu foncé), et les deux motifs thioredoxin-like CXXC de liaison au zinc (dont les acides aminés sont indiqués en rouge), constituent une structure hautement conservée qui est commune à toutes les espèces. Un agrandissement du site actif de ScPng1p montre que la triade catalytique (C191, H218 et D235) se situe au fond d’une structure qui a la forme d’un sillon. Le modèle tridimensionnel de la PNGase humaine (HsNgly1p) a été construit à l’aide du logiciel Modeller v9.11 (http://salilab.org/modeller/) en se basant sur l’homologie de séquence des différents domaines de HsNgly1p, de l’enzyme murine (PUB, pdb 2CCQ et 2D5U ; PAW, pdb 2C9G) et du complexe murin PNGaseI/HR23 (pdb 2F4M). La PNGase de mammifère est codée par le gène NGLY1 : en plus du domaine transglutaminase, elle comporte, respectivement, aux extrémités amino- et carboxy-terminales, le domaine PUB (en jaune, domaine d’interaction protéique) et le domaine PAW (en vert, domaine de liaison aux résidus de mannose). |
La PNGase des eucaryotes supérieurs contient, dans les régions amino- et carboxyterminales, deux autres domaines qui sont respectivement le domaine PUB (peptide N-glycanase/UBA ou UBX-containing proteins) et le domaine PAW (PNGases and other worm proteins) (Figure 2). Chez les mammifères, le gène orthologue à ScPNG1 est le gène NGLY1 (N-glycanase) dont l’expression est ubiquiste [7]. Au niveau subcellulaire, l’enzyme Ngly1p est majoritairement présente dans le cytosol, mais certaines études suggèrent également une association de la protéine à la membrane du RE [8].
Les différentes caractéristiques de l’activité de déglycosylation
La comparaison de la séquence peptidique de ScPng1p avec celle d’un membre de la famille des transglutaminases (le facteur XIIIa) a permis de mettre en exergue la conservation de nombreux acides aminés [9], dont ceux qui correspondent à la triade catalytique des transglutaminases : Cys191, His218 et Asp235 [10]. Cette triade catalytique et les motifs CXXC, qui sont des sites de liaison du zinc [11], jouent un rôle important dans l’activité de la PNGase. La mutation des acides aminés de la triade catalytique [9, 11], ou celle des cystéines des motifs thioredoxin-like [9], entraînent une perte complète de l’activité enzymatique.
Il a été montré que le domaine carboxy-terminal PAW est un domaine de liaison au mannose qui stimule fortement l’activité de déglycosylation de l’enzyme Ngly1p de souris (mNgly1p) [12]. Il est possible que le domaine PAW inter-agisse d’abord avec les résidus mannosylés des chaînes N-glycanes. Puis, après un changement de conformation du bras reliant le domaine transglutaminase au domaine PAW, la partie chitobiose1 de l’oligosaccharide se positionne dans le site actif (Figure 3), puis la chaîne N-glycane est alors hydrolysée.
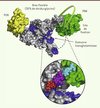 |
Figure 3. Repliement hypothétique du domaine PAW de la PNGase humaine permettant la continuité du site de liaison des résidus mannose avec le sillon du site actif. Le bras reliant le domaine transglutaminase et le domaine PAW (en vert) est riche en résidus glycine (environ 30 %), lui conférant une large flexibilité. Sur la base de l’hypothèse émise par Zhou et al. [12], le domaine PAW du modèle 3D de HsNgly1p (voir Figure 2 ) est manuellement amené au niveau du sillon du site actif du domaine transglutaminase. Un agrandissement de la région comprenant la zone de contact entre les deux domaines montre la fixation d’un ligand hypothétique formé par la juxtaposition de l’inhibiteur Z-VAD-fmk (rouge), de GlcNAcβ1,4GlcNAc (pourpre) et de résidus mannose (bleu turquoise). |
La PNGase déglycosyle les N-glycosylprotéines mal repliées
Une des caractéristiques remarquables de cette enzyme est sa capacité de discriminer entre une N-glycosylprotéine correctement repliée et une N-glycosylprotéine dénaturée ; elle préfère déglycosyler cette dernière [13]. Cette propriété est liée à la conformation tridimensionnelle tout à fait particulière de la région qui englobe le site actif. Cette région, qui est à l’interface entre le domaine central et le domaine de liaison au zinc, adopte la structure d’un sillon au fond duquel réside le site actif [11] (Figure 2).
Les parois de ce sillon empêcheraient l’accès au site actif des chaînes N-glycanes des glycoprotéines natives, mais pas des glycoprotéines dénaturées dont la structure est plus flexible. Une étude effectuée sur ScPng1p conforte cette hypothèse : la délétion des 33 premiers acides aminés de la région aminoterminale induit un élargissement du sillon et confère la capacité de déglycosyler des N-glycosylprotéines natives [14].
La PNGase interagit avec différentes protéines impliquées dans le processus ERAD
Les N-glycosylprotéines qui n’ont pas pu acquérir leur conformation tridimensionnelle sont reconnues par un système de contrôle de qualité qui empêche leur accumulation dans la lumière du RE et entraîne leur dégradation par le protéasome. Il s’agit du processus ERAD (ER associated degradation) [15] (Figure 1B).
La PNGase interagit indirectement avec le protéasome via Rad23p chez la levure [16] ou via Hr23bp chez les mammifères [17]. La protéine Rad23/Hr23b joue un rôle clé dans le processus de réparation par excision de nucléotides (NER ou nucleotide excision repair). Elle forme un complexe avec Rad4p/XPCp qui se lie spécifiquement à l’ADN endommagé de façon dépendante de l’ATP. Le domaine amino-terminal ubiquitine-like (Ubl) permet également à la protéine Rad23/Hr23b de se lier à la sous-unité 26 S du protéasome.
Le domaine PUB de l’enzyme de mammifère permet d’établir de plus nombreuses interactions avec différentes protéines impliquées dans la voie ERAD : à proximité de la membrane du RE, mNGly1p fait partie d’un complexe de cinq protéines (mP97p, mAmfrp, mY33kp, mNgly1p et mHr23bp) [18] qui pourrait être responsable des activités d’ubiquitination, de rétrotranslocation, de déglycosylation et d’interaction avec le protéasome.
La PNGase est l’enzyme qui déglycosyle les substrats ERAD
La démonstration que la PNGase est responsable de la déglycosylation in vivo des substrats ERAD a été apportée en 2004. La diminution de l’expression de Ngly1p entraîne une diminution de la déglycosylation des chaînes lourdes du complexe majeur d’histocompatibilité (CMH) de classe I, et celle d’autres substrats ERAD, comme la chaîne α du récepteur T (TCRα, T-cell receptor α-chain). Dans ces conditions, la présence de molécules glycosylées de ces substrats a pu être observée dans le cytosol [19, 20].
Les paradoxes
Compte tenu de son activité de déglycosylation de N-glycosylprotéines mal repliées et de sa localisation subcellulaire stratégique entre le RE et le protéasome, il avait été envisagé que la PNGase puisse jouer un rôle crucial pendant le processus ERAD. Afin d’évaluer l’importance de cette enzyme dans ce processus, deux approches complémentaires ont été développées qui consistent à étudier l’effet d’une inhibition de l’expression de la PNGase, soit sur le taux de dégradation de substrats ERAD, soit sur la génération de fOS (free oligosaccharide). La 2e approche permet d’analyser de façon plus globale l’ensemble des N-glycosylprotéines qui sont déglycosylées par cette enzyme (Figure 1).
La PNGase modifie peu le taux de dégradation des substrats ERAD
Des études in vivo montrent que le taux de dégradation de la majorité des substrats ERAD étudiés n’est pas profondément altéré par un grand nombre de conditions qui induisent une inhibition de la PNGase, comme la délétion du gène correspondant chez la levure [6], ou l’utilisation d’ARNi (interférents) spécifiques [20], ou de l’inhibiteur Z-VAD-fmk (un inhibiteur des caspases) [19]. In vitro, la présence de la chaîne N-glycane n’empêche pas la dégradation de glycoprotéines dénaturées ou de substrats ERAD par les structures 20 S et 26 S du protéasome, mais la ralentit [21].
Bien que la présence d’une chaîne N-glycane ne constitue pas un obstacle majeur, son hydrolyse améliorerait la dégradation de glycoprotéines mal repliées en facilitant l’insertion de la partie peptidique dans la chambre de dégradation qui se situe à l’intérieur même du protéasome [22].
Paradoxes concernant la génération des fOS et l’expression de PNG1 chez la levure
Alors que plus de 95 % des fOS sont libérés par Png1p chez S. cerevisiae, seulement 50 % de ces structures résultent de la dégradation de N-glycosylprotéines via les voies ERAD connues [23]. Ces données suggèrent qu’il existe d’autres voies ERAD encore non découvertes ou que ScPng1p est impliquée dans un autre processus de dégradation des N-glycosylprotéines.
Il semble également paradoxal, d’une part, que l’activité PNGase [5] et la génération de fOS [23] augmentent en fin de phase exponentielle de croissance au moment où les activités protéasomales diminuent et, d’autre part, que l’expression de Png1p soit maximale pendant la phase stationnaire de croissance [24], alors que le protéasome est complètement déstructuré [25].
Les structures libérées par la PNGase sont différentes de celles reconnues par Os9p
La voie ERAD-L est dépendante des chaînes N-glycanes des N-glycosylprotéines mal repliées et nécessite une démannosylation partielle de la branche C de l’oligosaccharide (voir Figure 1A pour la description des branches de l’oligosaccharide et Figure 1B pour la voie ERAD-L), modification qui va alors être reconnue comme signal de dégradation par le domaine CRD (carbohydrate recognition domain) de Yos9p/Os9p [26] (Figure 4). In vitro, le domaine PAW de mNgly1p a une affinité plus élevée pour les structures oligosaccharidiques (Glc1-0Man9-8GlcNAc2) dont la branche C de l’oligosaccharide est intacte. In vivo, les fOS dont le taux a diminué après l’inhibition de cette enzyme dans la lignée d’hépatocarcinome humain HepG2, sont également des structures (Glc1-0Man9-8GlcNAc2) [27].
 |
Figure 4. Les différentes structures oligosaccharidiques reconnues par le domaine CRD de Os9p ou le domaine PAW de Ngly1p. Les structures oligosaccharidiques partiellement démannosylées, dont la branche C, ne possèdent plus le résidu mannose terminal et sont préférentiellement reconnues par Os9p : c’est le résidu de mannose en α1,6 démasqué sur la même branche (indiqué par un astérisque rouge) qui est reconnu spécifiquement par le domaine CRD de Os9p. De façon paradoxale, les structures oligosaccharidiques (Glc1-0Man9-8GlcNAc2) qui sont reconnues par le domaine PAW de la PNGase murine possèdent le résidu mannose terminal de la branche C. Ces structures n’ont donc pas pu interagir avec le domaine CRD de Os9p [26]. |
Ainsi les chaînes N-glycanes qui sont reconnues par le domaine CRD de Yos9p/Os9p sont différentes de celles qui sont reconnues ou libérées par la PNGase, alors que cette enzyme intervient au cours du processus ERAD après la protéine Yos9p/Os9p (Figure 1B).
Autres fonctions et autres isoformes potentielles de la PNGase
Ces différents paradoxes suggèrent que le rôle de PNG1 est plus complexe que ce qui avait été initialement décrit.
PNG1 joue un rôle dans la mise en place de la polarité cellulaire
Il avait été observé que la délétion du gène PNG1 n’était pas associée à un phénotype particulier, en dehors de l’absence d’activité de déglycosylation, chez les organismes unicellulaires comme la levure S. cerevisiae [6].
Des articles apparus récemment dans la littérature décrivent cependant que la délétion du gène PNG1 dans des organismes pluricellulaires entraîne l’apparition de défauts morphogéniques. Chez Dictyostelium discoideum, PNG1 est en effet indispensable à la formation d’agrégats cellulaires pendant le développement multicellulaire [28]. Des mutants de délétion du gène PNG1 chez Drosophila melanogaster montrent un arrêt ou un ralentissement du développement et une stérilité [29]. La délétion du gène PNG1 entraîne des défauts de polarisation pendant la croissance hyphale du champignon Neurospora crassa [30] et pendant la croissance neuronale chez Caenorhabditis elegans [31].
Ces activités morphogéniques de la PNGase sont indépendantes de son activité de déglycosylation : les protéines Png1p de Drosophila melanogaster et de Neurospora crassa sont en effet inactives par défaut d’un motif CXXC (D. melanogaster) [29] ou par l’absence d’un des acides aminés de la triade catalytique (N. crassa) [30].
Ces propriétés nouvellement décrites de la PNGase semblent avoir été conservées au cours de l’évolution : la réversion des phénotypes des mutants de délétion a été obtenue, chez D. melanogaster, par l’expression de mNgly1p, [29] et, chez N. crassa, par l’expression de ScPng1p [30]. Ces données excluent donc que ces propriétés soient portées par les régions amino- ou carboxyterminales de la PNGase des eucaryotes supérieurs qui sont différentes de celles de l’enzyme de levure.
Différents transcrits du gène NGLY1 coderaient pour des isoformes dont la structure primaire est différente
Il existe différents transcrits générés par épissage alternatif du gène humain NGLY1 qui coderaient pour des isoformes de la PNGase. Il existe dans ces isoformes une modification importante dans un des trois domaines de l’enzyme qui devrait entraîner, respectivement, une perturbation des interactions protéiques de la PNGase, de son activité de déglycosylation ou de la reconnaissance de la structure oligosaccharidique (Figure 5). Bien que l’existence même de ces isoformes reste à prouver, des clones EST (expressed sequences tag) de ces différentes isoformes ont été trouvés dans différentes espèces de mammifères, suggérant que chacune de ces isoformes assure une fonction physiologique qui a été conservée.
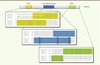 |
Figure 5. Comparaison de la séquence peptidique des domaines PUB, transglutaminase et PAW de différentes isoformes putatives de la PNGase humaine. Les séquences de différents clones EST (expressed sequences tag) correspondant à des transcrits générés par épissage alternatif du gène humain NGLY1 ont été compilées dans le site genome browser (http://www.ensembl.org/index.html). Alors que la séquence peptidique de la PNGase humaine comporte 655 acides aminés (CCDS33719), une isoforme de seulement 613 acides aminés (CCDS46777) correspond à la délétion des 42 premiers acides aminés dans la région amino-terminale ; une isoforme de 637 acides aminés (CCDS46778) ne possède pas les deux acides aminés histidine et aspartate de la triade catalytique (indiquée par des cadres en pointillé) ; et une isoforme de 559 acides aminés (CCDS46779) est tronquée dans la région carboxy-terminale. Ces différentes isoformes montrent des modifications des domaines PUB, transglutaminase et PAW. Les lettres indiquées en rouge correspondent aux acides aminés qui sont spécifiques d’une des isoformes. |
La lignée d’hépatocarcinome humain HepG2 exprime deux transcrits différents du gène NGLY1 : un transcrit majoritaire codant pour la protéine Ngly1p de 655 aa (voir CCDS33719 dans la Figure 5 ), et un autre transcrit qui coderait pour une isoforme dont le domaine PAW serait tronqué (voir CCDS46779 dans la Figure 5 ). Dans nos conditions expérimentales de marquage métabolique en présence de [3H]mannose et en utilisant la stratégie ARNi, nous avons montré que le transcrit minoritaire ne serait pas impliqué dans la génération de fOS radiomarqués [27].
Déficit en Ngly1p : une nouvelle pathologie
Le séquençage de l’ensemble des exons de l’ADN génomique a permis d’identifier des mutations du gène NGLY1 chez un premier patient. La protéine Ngly1p est indétectable dans les cellules du patient, et son taux est fortement réduit dans les leucocytes des parents de ce patient. Ce dernier souffre d’un retard de développement mental, d’épilepsie multifocale, de mouvements involontaires, d’absence de larmes et d’un dysfonctionnement hépatique. Une biopsie du foie a montré l’accumulation d’une substance amorphe dans le cytoplasme qui pourrait correspondre à l’accumulation de N-glycosylprotéines mal repliées [3].
Les atteintes hépatiques et celles du système nerveux central pourraient résulter directement de défauts morphogéniques ou indirectement de l’accumulation d’agrégats protéiques. De nombreuses maladies neurodégénératives sont associées à un dysfonctionnement du contrôle de qualité qui joue un rôle déterminant dans l’accumulation des protéines mal repliées et dans l’évolution de la maladie. Il est donc légitime de se demander si un dysfonctionnement de cette enzyme ne peut pas être associé à une neurodégénérescence comme cela avait été observé dans les dysfonctionnements de certains représentants de la famille des transglutaminases [10].
Alors que l’expression de Ngly1p est ubiquiste, il est également surprenant que certains organes (comme le foie ou le système nerveux central) soient plus particulièrement affectés. Une des explications possibles est qu’une autre enzyme - l’endo-β-N-acétylglucosaminidase (ENGase) - puisse prendre le relais de la PNGase pour assurer la déglycosylation des N-glycosylprotéines mal repliées dans les autres tissus (Figure 1A), comme semblent le suggérer nos études d’expression de l’ENGase humaine chez la levure dans le mutant de délétion Png1∆ [27].
Conclusions et perspectives
La PNGase a été clonée chez la levure sur la base de son activité de déglycosylation des N-glycosylprotéines. Pendant de nombreuses années, le rôle majeur qui lui a été attribué, aussi bien chez les levures que chez les mammifères, est de déglycosyler les glycoprotéines mal repliées avant leur dégradation par le protéasome [34]. Cependant, certaines données semblent contradictoires avec un rôle unique de la PNGase dans le processus ERAD. Il s’avère en effet que cette enzyme n’est pas indispensable pour la dégradation protéasomale de la majorité de substrats ERAD, et qu’elle est capable de moduler d’autres activités biologiques fondamentales chez les eucaryotes supérieurs, telles que la morphogenèse et la mise en place de la polarité cellulaire. Ainsi, au fur et à mesure de l’avancée des connaissances, d’autres fonctions de la PNGase sont révélées, qui sont indépendantes de la capacité de déglycosylation de l’enzyme.
À l’avenir, trois questions fondamentales doivent être posées :
-
Quelle est la fonction de ScPNG1 pendant la phase stationnaire de croissance ?
-
Quels sont les mécanismes moléculaires qui peuvent rendre compte des effets morphogéniques d’un déficit en PNGase ?
-
Quelles sont les activités portées par les différentes isoformes ?
L’analyse des mécanismes moléculaires liés à un déficit de la PNGase humaine devrait permettre de distinguer ceux qui résultent d’un défaut de l’activité de déglycosylation de ceux qui résultent de défauts morphogéniques.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Winchester B. Lysosomal metabolism of glycoproteins. Glycobiology 2005 ; 15 : 1R–15R. [CrossRef] [PubMed] [Google Scholar]
- Aronson NN, Jr. Aspartylglycosaminuria: biochemistry and molecular biology. Biochim Biophys Acta 1999 ; 1455 : 139–154. [CrossRef] [PubMed] [Google Scholar]
- Need AC, Shashi V, Hitomi Y, et al. Clinical application of exome sequencing in undiagnosed genetic conditions. J Med Genet 2012 ; 49 : 353–361. [CrossRef] [PubMed] [Google Scholar]
- Suzuki T, Seko A, Kitajima K, et al. Identification of peptide: N-glycanase activity in mammalian-derived cultured cells. Biochem Biophys Res Commun 1993 ; 194 : 1124–1130. [CrossRef] [PubMed] [Google Scholar]
- Suzuki T, Park H, Kitajima K, Lennarz WJ. Peptides glycosylated in the endoplasmic reticulum of yeast are subsequently deglycosylated by a soluble peptide: N-glycanase activity. J Biol Chem 1998 ; 273 : 21526–21530. [CrossRef] [PubMed] [Google Scholar]
- Suzuki T, Park H, Hollingsworth NM, et al. PNG1, a yeast gene encoding a highly conserved peptide: N-glycanase. J Cell Biol 2000 ; 149 : 1039–1052. [CrossRef] [PubMed] [Google Scholar]
- Suzuki T, Kwofie MA, Lennarz WJ. Ngly1, a mouse gene encoding a deglycosylating enzyme implicated in proteasomal degradation: expression, genomic organization, and chromosomal mapping. Biochem Biophys Res Commun 2003 ; 304 : 326–332. [CrossRef] [PubMed] [Google Scholar]
- Katiyar S, Li G, Lennarz WJ. A complex between peptide: N-glycanase and two proteasome-linked proteins suggests a mechanism for the degradation of misfolded glycoproteins. Proc Natl Acad Sci USA 2004 ; 101 : 13774–13779. [CrossRef] [Google Scholar]
- Katiyar S, Suzuki T, Balgobin BJ, Lennarz WJ. Site-directed mutagenesis study of yeast peptide: N-glycanase. Insight into the reaction mechanism of deglycosylation. J Biol Chem 2002 ; 277 : 12953–12959. [CrossRef] [PubMed] [Google Scholar]
- Lismaa SE, Mearns BM, Lorand L, Graham RM. Transglutaminases and disease: lessons from genetically engineered mouse models and inherited disorders. Physiol Rev 2009 ; 89 : 991–1023. [CrossRef] [PubMed] [Google Scholar]
- Lee JH, Choi JM, Lee C, et al. Structure of a peptide: N-glycanase-Rad23 complex: insight into the deglycosylation for denatured glycoproteins. Proc Natl Acad Sci USA 2005 ; 102 : 9144–9149. [CrossRef] [Google Scholar]
- Zhou X, Zhao G, Truglio JJ, et al. Structural and biochemical studies of the C-terminal domain of mouse peptide-N-glycanase identify it as a mannose-binding module. Proc Natl Acad Sci USA 2006 ; 103 : 17214–17219. [CrossRef] [Google Scholar]
- Hirsch C, Misaghi S, Blom D, et al. Yeast N-glycanase distinguishes between native and non-native glycoproteins. EMBO Rep 2004 ; 5 : 201–206. [CrossRef] [PubMed] [Google Scholar]
- Wang S, Xin F, Liu X, et al. N-terminal deletion of peptide: N-glycanase results in enhanced deglycosylation activity. PLoS One 2009 ; 4 : e8335. [CrossRef] [PubMed] [Google Scholar]
- Olzmann JA, Kopito RR, Christianson JC. The mammalian endoplasmic reticulum-associated degradation system. Cold Spring Harb Perspect Biol 2013. doi: 10.1101/cshperspect.a013185. [Google Scholar]
- Suzuki T, Park H, Kwofie MA, Lennarz WJ. Rad23 provides a link between the Png1 deglycosylating enzyme and the 26 S proteasome in yeast. J Biol Chem 2001 ; 276 : 21601–21607. [CrossRef] [PubMed] [Google Scholar]
- Park H, Suzuki T, Lennarz WJ. Identification of proteins that interact with mammalian peptide: N-glycanase and implicate this hydrolase in the proteasome-dependent pathway for protein degradation. Proc Natl Acad Sci USA 2001 ; 98 : 11163–11168. [CrossRef] [Google Scholar]
- Li G, Zhao G, Zhou X, et al. The AAA ATPase p97 links peptide: N-glycanase to the endoplasmic reticulum-associated E3 ligase autocrine motility factor receptor. Proc Natl Acad Sci USA 2006 ; 103 : 8348–8353. [CrossRef] [Google Scholar]
- Misaghi S, Pacold ME, Blom D, et al. Using a small molecule inhibitor of peptide: N-glycanase to probe its role in glycoprotein turnover. Chem Biol 2004 ; 11 : 1677–1687. [CrossRef] [PubMed] [Google Scholar]
- Blom D, Hirsch C, Stern P, et al. A glycosylated type I membrane protein becomes cytosolic when peptide: N-glycanase is compromised. EMBO J 2004 ; 23 : 650–658. [CrossRef] [PubMed] [Google Scholar]
- Kario E, Tirosh B, Ploegh HL, Navon A. N-linked glycosylation does not impair proteasomal degradation but affects class I major histocompatibility complex presentation. J Biol Chem 2008 ; 283 : 244–254. [CrossRef] [PubMed] [Google Scholar]
- Andermarcher E, Bossis G, Farras R, et al. La dégradation protéasomique : de l’adressage des protéines aux nouvelles perspectives thérapeutiques. Med Sci (Paris) 2005 ; 21 : 141–149. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Chantret I, Kodali VP, Lahmouich C, et al. Endoplasmic reticulum-associated degradation (ERAD) and free oligosaccharide generation in Saccharomyces cerevisiae. J Biol Chem 2011 ; 286 : 41786–41800. [CrossRef] [PubMed] [Google Scholar]
- Davidson GS, Joe RM, Roy S, et al. The proteomics of quiescent and nonquiescent cell differentiation in yeast stationary-phase cultures. Mol Biol Cell 2011 ; 22 : 988–998. [CrossRef] [PubMed] [Google Scholar]
- Laporte D, Salin B, Daignan-Fornier B, Sagot I. Reversible cytoplasmic localization of the proteasome in quiescent yeast cells. J Cell Biol 2008 ; 181 : 737–745. [CrossRef] [PubMed] [Google Scholar]
- Kamiya Y, Satoh T, Kato K. Molecular and structural basis for N-glycan-dependent determination of glycoprotein fates in cells. Biochim Biophys Acta 2012 ; 1820 : 1327–1337. [CrossRef] [PubMed] [Google Scholar]
- Chantret I, Fasseu M, Zaoui K, et al. Identification of roles for peptide: N-glycanase, endo-beta-N-acetylglucosaminidase (Engase1p) during protein N-glycosylation in human HepG2 cells. PLoS One 2010 ; 5 : e11734. [CrossRef] [PubMed] [Google Scholar]
- Gosain A, Lohia R, Shrivastava A, Saran S. Identification characterization of peptide: N-glycanase from Dictyostelium discoideum. BMC Biochem 2012 ; 13 : 9. [CrossRef] [PubMed] [Google Scholar]
- Funakoshi Y, Negishi Y, Gergen JP, et al. Evidence for an essential deglycosylation-independent activity of PNGase in Drosophila melanogaster. PLoS One 2010 ; 5 : e10545. [CrossRef] [PubMed] [Google Scholar]
- Maerz S, Funakoshi Y, Negishi Y, et al. The Neurospora peptide: N-glycanase ortholog PNG1 is essential for cell polarity despite its lack of enzymatic activity. J Biol Chem 2010 ; 285 : 2326–2332. [CrossRef] [PubMed] [Google Scholar]
- Habibi-Babadi N, Su A, de Carvalho CE, Colavita A. The N-glycanase png-1 acts to limit axon branching during organ formation in Caenorhabditis elegans. J Neurosci 2010 ; 30 : 1766–1776. [CrossRef] [PubMed] [Google Scholar]
- Alonzi DS, Kukushkin NV, Allman SA, et al. Glycoprotein misfolding in the endoplasmic reticulum: identification of released oligosaccharides reveals a second ER-associated degradation pathway for Golgi-retrieved proteins. Cell Mol Life Sci 2013 ; 70 : 2799–2814. [CrossRef] [PubMed] [Google Scholar]
- Chantret I, Moore SE. Free oligosaccharide regulation during mammalian protein N-glycosylation. Glycobiology 2008 ; 18 : 210–224. [CrossRef] [PubMed] [Google Scholar]
- Andermarcher E, Bossis G, Farras R, et al. La dégradation protéomique : de l’adressage des protéines aux nouvelles perspectives thérapeutiques. Med Sci (Paris) 2005 ; 21 : 141–149. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Les différentes enzymes capables de déglycosyler les N-glycosylprotéines et le rôle de la PNGase pendant le processus ERAD-L. A. Deux activités enzymatiques intracellulaires majeures sont capables de débrancher les chaînes N-glycanes en clivant la liaison entre le premier résidu de N-acétylglucosamine (GlcNAc) de l’oligosaccharide et l’asparagine (Asn) porteuse de la chaîne N-glycane. Au niveau du cytoplasme, une des activités de déglycosylation est portée par la peptide N-glycanase (PNGase), qui génère de façon concomitante un oligosaccharide libre (fOS) et la conversion du résidu Asn porteur de la chaîne N-glycane en résidu aspartate (Asp, panneau de gauche). Dans les lysosomes, une autre activité est portée par la N 4-(β-N-acétylglucosaminyl)-1-asparaginase qui ne peut agir que si la dégradation de la partie peptidique de la glycoprotéine a eu lieu au préalable (panneau de droite). Certaines données suggèrent également qu’il existerait une activité PNGase à l’intérieur même du réticulum endoplasmique (RE), mais bien que cette activité soit sensible à l’inhibiteur Z-VAD-fmk, la protéine responsable n’a pas encore été caractérisée [32, 33]. À ces activités de déglycosylation, il faut ajouter l’endo-β-N-acétylglucosaminidase cytosolique (ENGase) qui est également capable d’hydrolyser les chaînes N-liées en clivant la liaison entre les deux résidus de N-acétylglucosamine (GlcNAc) de l’oligosaccharide (panneau du milieu), et qui pourrait prendre le relais de la PNGase. B. Le processus ERAD est une machinerie moléculaire complexe ; elle permet de repérer les N-glycosylprotéines qui restent mal repliées après leur biosynthèse dans le RE, puis de les dégrader via le protéasome. Ce processus a été conservé au cours de l’évolution et les connaissances qui ont été établies à partir du modèle de la levure sont applicables aux eucaryotes supérieurs, avec toutefois un niveau de complexité plus élevé. Trois voies ERAD ont été décrites qui reconnaissent des domaines peptidiques mal repliés localisés, soit dans le cytosol (ERAD-C), soit dans la membrane (ERAD-M), soit dans la lumière du RE (ERAD-L). N’est représentée sur le schéma que la voie ERAD-L chez S. cerevisiae. Cette voie ERAD-L est la mieux connue et repose sur trois propriétés : la reconnaissance de la région peptidique, qui reste mal repliée, par l’apparition de régions hydrophobes accessibles ; la modification de la structure oligosaccharidique par l’action successive de différentes glycosidases (Mns1p [mannosidase réticulaire I] et le complexe Mnl1p/Htm1p-Pdi1p) ; et la reconnaissance de la structure oligosaccharidique Man7GlcNAc2 qui en résulte comme signal de dégradation. Cette structure comporte un résidu mannose en α1,6, démasqué sur la branche C de l’oligosaccharide et qui est reconnu spécifiquement par la lectine Yos9p/Os9p. Des complexes transmembranaires protéiques organisés autour de différentes E3 ubiquitine ligases (Hrd1p, Hrd3p, et Der1p) jouent un rôle stratégique, car ils coordonnent les différents évènements qui ont lieu de part et d’autre de la membrane du RE, tels que l’ubiquitination des N-glycosylprotéines mal repliées et leur rétrotranslocation vers le cytosol. Les substrats ubiquitinés sont alors déglycosylés par la PNGase cytosolique avant leur dégradation par le protéasome [22]. Les fOS, qui sont issus de l’hydrolyse des chaînes N-glycanes, sont eux-mêmes soumis à une voie métabolique : chez la levure, ils sont démannosylés de façon progressive par une mannosidase cytosolique codée par le gène Ams1 jusqu’à la structure ne comportant plus que deux résidus de GlcNAc et un résidu de mannose [23]. |
| Dans le texte | |
 |
Figure 2. Comparaison des structures tridimensionnelles des PNGases de levure et humaine. La PNGase de Saccharomyces cerevisiae (ScPng1p) est codée par le gène PNG1 : le domaine central transglutaminase, qui comporte le site actif (en bleu foncé), et les deux motifs thioredoxin-like CXXC de liaison au zinc (dont les acides aminés sont indiqués en rouge), constituent une structure hautement conservée qui est commune à toutes les espèces. Un agrandissement du site actif de ScPng1p montre que la triade catalytique (C191, H218 et D235) se situe au fond d’une structure qui a la forme d’un sillon. Le modèle tridimensionnel de la PNGase humaine (HsNgly1p) a été construit à l’aide du logiciel Modeller v9.11 (http://salilab.org/modeller/) en se basant sur l’homologie de séquence des différents domaines de HsNgly1p, de l’enzyme murine (PUB, pdb 2CCQ et 2D5U ; PAW, pdb 2C9G) et du complexe murin PNGaseI/HR23 (pdb 2F4M). La PNGase de mammifère est codée par le gène NGLY1 : en plus du domaine transglutaminase, elle comporte, respectivement, aux extrémités amino- et carboxy-terminales, le domaine PUB (en jaune, domaine d’interaction protéique) et le domaine PAW (en vert, domaine de liaison aux résidus de mannose). |
| Dans le texte | |
 |
Figure 3. Repliement hypothétique du domaine PAW de la PNGase humaine permettant la continuité du site de liaison des résidus mannose avec le sillon du site actif. Le bras reliant le domaine transglutaminase et le domaine PAW (en vert) est riche en résidus glycine (environ 30 %), lui conférant une large flexibilité. Sur la base de l’hypothèse émise par Zhou et al. [12], le domaine PAW du modèle 3D de HsNgly1p (voir Figure 2 ) est manuellement amené au niveau du sillon du site actif du domaine transglutaminase. Un agrandissement de la région comprenant la zone de contact entre les deux domaines montre la fixation d’un ligand hypothétique formé par la juxtaposition de l’inhibiteur Z-VAD-fmk (rouge), de GlcNAcβ1,4GlcNAc (pourpre) et de résidus mannose (bleu turquoise). |
| Dans le texte | |
 |
Figure 4. Les différentes structures oligosaccharidiques reconnues par le domaine CRD de Os9p ou le domaine PAW de Ngly1p. Les structures oligosaccharidiques partiellement démannosylées, dont la branche C, ne possèdent plus le résidu mannose terminal et sont préférentiellement reconnues par Os9p : c’est le résidu de mannose en α1,6 démasqué sur la même branche (indiqué par un astérisque rouge) qui est reconnu spécifiquement par le domaine CRD de Os9p. De façon paradoxale, les structures oligosaccharidiques (Glc1-0Man9-8GlcNAc2) qui sont reconnues par le domaine PAW de la PNGase murine possèdent le résidu mannose terminal de la branche C. Ces structures n’ont donc pas pu interagir avec le domaine CRD de Os9p [26]. |
| Dans le texte | |
 |
Figure 5. Comparaison de la séquence peptidique des domaines PUB, transglutaminase et PAW de différentes isoformes putatives de la PNGase humaine. Les séquences de différents clones EST (expressed sequences tag) correspondant à des transcrits générés par épissage alternatif du gène humain NGLY1 ont été compilées dans le site genome browser (http://www.ensembl.org/index.html). Alors que la séquence peptidique de la PNGase humaine comporte 655 acides aminés (CCDS33719), une isoforme de seulement 613 acides aminés (CCDS46777) correspond à la délétion des 42 premiers acides aminés dans la région amino-terminale ; une isoforme de 637 acides aminés (CCDS46778) ne possède pas les deux acides aminés histidine et aspartate de la triade catalytique (indiquée par des cadres en pointillé) ; et une isoforme de 559 acides aminés (CCDS46779) est tronquée dans la région carboxy-terminale. Ces différentes isoformes montrent des modifications des domaines PUB, transglutaminase et PAW. Les lettres indiquées en rouge correspondent aux acides aminés qui sont spécifiques d’une des isoformes. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




