| Issue |
Med Sci (Paris)
Volume 28, Number 4, Avril 2012
|
|
|---|---|---|
| Page(s) | 423 - 429 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2012284021 | |
| Published online | 25 April 2012 | |
Stratégies de formation de la structure coiffe chez les virus à ARN
Capping strategies in RNA viruses
Laboratoire CNRS - Architecture et fonction des macromolécules biologiques (AFMB), UMR 7257, groupe réplication virale, structures, mécanismes et drug-design, école supérieure d’ingénieurs de Luminy (ESIL) - Case 925, 163, avenue de Luminy, 13288 Marseille Cedex 09, France
*
etienne.decroly@afmb.univ-mrs.fr
La plupart des virus utilisent la machinerie de traduction dépendante de la coiffe pour assurer l’expression de leurs ARN messagers (ARNm). L’addition d’une structure coiffe à l’extrémité 5’ des ARNm viraux est par conséquent une étape essentielle pour la réplication de nombreux virus. En effet, la coiffe protège les ARNm de la dégradation par les nucléases cellulaires et neutralise la détection des ARNm viraux par les mécanismes de l’immunité innée. L’acquisition de la coiffe des ARN viraux se fait soit en utilisant les enzymes cellulaires de formation de la coiffe de la cellule infectée ou en subtilisant la coiffe des ARNm de la cellule infectée, soit par des machineries enzymatiques virales dédiées. De nombreuses enzymes virales impliquées dans la synthèse de la coiffe ont récemment été caractérisées du point de vue structural et fonctionnel. Ces études ont révélé des mécanismes de synthèse originaux qui ouvrent la voie pour le développement d’inhibiteurs spécifiques à potentiel antiviral.
Abstract
Most viruses use the mRNA-cap dependent cellular translation machinery to translate their mRNAs into proteins. The addition of a cap structure at the 5’ end of mRNA is therefore an essential step for the replication of many virus families. Additionally, the cap protects the viral RNA from degradation by cellular nucleases and prevents viral RNA recognition by innate immunity mechanisms. Viral RNAs acquire their cap structure either by using cellular capping enzymes, by stealing the cap of cellular mRNA in a process named “cap snatching”, or using virus-encoded capping enzymes. Many viral enzymes involved in this process have recently been structurally and functionally characterized. These studies have revealed original cap synthesis mechanisms and pave the way towards the development of specific inhibitors bearing antiviral drug potential.
© 2012 médecine/sciences – Inserm / SRMS
Mécanismes de formation de la coiffe chez les eucaryotes
Les ARN pré-messagers transcrits dans le noyau par l’ARN polymérase II ADN-dépendante (ARN Pol II) subissent plusieurs modifications co- et post-transcriptionnelles, puis sont exportés vers le cytoplasme et traduits en protéines par les ribosomes. Parmi ces modifications, on trouve l’addition d’une structure coiffe à l’extrémité 5’ des ARNm, l’épissage des introns et la synthèse d’une queue poly A [1, 2]. La coiffe est constituée d’une guanosine méthylée en position N7 liée par une liaison 5’-5’ triphosphate au premier nucléotide transcrit de l’ARNm (7MeGpppN) (Figure 1). Son rôle principal est de recruter le facteur eIF4E (eukaryotic translation initiation factor 4E), afin de permettre l’initiation de la traduction des ARNm en protéines [3]. La coiffe protège également les ARNm de leur dégradation par les nucléases cellulaires, facilite l’excision de séquences introniques [4] et empêche la détection des ARNm par les senseurs de l’immunité innée RIG-1 (retinoic acid inducible gene 1 protein) et MDA5 (melanoma-differentiation-associated gene 5) [2, 5, 6, 31].
 |
Figure 1. Structure de la coiffe. La structure coiffe-0 (fond bleu) est composée d’un résidu guanosine méthylé en position N7 (cercle vert à gauche) qui est lié par une liaison 5’-5’ triphosphate (fond rose) au nucléotide en aval de la coiffe. Un groupement méthyl (cercle vert à droite) peut être ajouté à la position 2’O du ribose du premier nucléotide de l’ARNm pour former la coiffe-1. |
La formation de la coiffe chez les eucaryotes nécessite l’action séquentielle de plusieurs activités enzymatiques (Figure 2A). La première étape consiste en l’hydrolyse du phosphate γ de l’extrémité 5’ triphosphate de l’ARNm naissant par une ARN triphosphatase (RTPase). L’ARN guanylyltransférase (GTase) recrute une molécule de GTP et forme un intermédiaire covalent via sa lysine catalytique, avant de transférer le groupement GMP sur l’extrémité 5’ de l’ARN diphosphate. Enfin, une N7-méthyltransférase (N7-MTase) méthyle la guanosine sur la position N7 en utilisant comme donneur de méthyl le S-adénosyl-L-méthionine (SAM ou AdoMet) [1, 2]. L’inactivation d’une seule des trois enzymes impliquées dans la formation de la coiffe est létale pour les cellules, ce qui démontre l’importance de la coiffe pour la survie cellulaire [1].
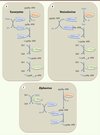 |
Figure 2. Différents mécanismes de formation de la coiffe. A. Ce panneau représente le mécanisme « conventionnel » de formation de la coiffe chez les eucaryotes. Le phosphate γ à l’extrémité 5’ de l’ARN pré-messager est hydrolysé par l’ARN triphosphatase (RTPase). L’ARN guanylyltransférase (GTase) fixe ensuite une molécule de GMP à partir du GTP (Gppp) en libérant du pyrophosphate inorganique (PPi). La GTase transfère le GMP sur l’extrémité diphosphate de l’ARN prémessager formant une liaison 5’-5’ triphosphate. La synthèse de la coiffe se termine par la méthylation du résidu guanosine en position N7 par la N7-MTase à partir du donneur de méthyl (SAM). Enfin, chez les eucaryotes supérieurs, la formation de la structure coiffe-1 fait intervenir une 2’O-MTase qui méthyle la position 2’O du ribose du premier nucléotide de l’ARN. B.Ce panneau illustre le mécanisme de formation de la coiffe chez les Vésiculovirus. La PRNTase recrute l’ARN triphosphate pour former un lien covalent avec l’ARN 5’ diphosphate et le transfère ensuite sur une molécule de GDP (Gpp). L’ordre de la séquence de méthylation est inversé par rapport au mécanisme canonique retrouvé chez les eucaryotes (A.). C. Ce panneau illustre le mécanisme de coiffe des Alphavirus où la N7-méthylation précède le transfert du GMP sur l’ARN diphosphate. |
La synthèse nucléaire de la structure coiffe est couplée à la transcription des ARNm par l’ARN Pol II. En effet, les enzymes de formation de la coiffe sont recrutées via le domaine carboxy-terminal (CTD) de l’ARN Pol II, après phosphorylation au niveau de la sérine 5 du motif YSPTSPS [7]. Chez les eucaryotes supérieurs, les nucléotides adjacents à la coiffe peuvent également être méthylés en position 2’O de leur groupement ribose par des 2’O-méthyltransférases (2’O-MTases), hMTr1 (human methyltransferase 1) et hMTr2 chez l’humain, générant respectivement des structures coiffe-1 (7MeGpppN2’OMepN) ou coiffe-2 (7MeGpppN2’OMepN2’OMe). Il semble que ces méthylations en position 2’O favorisent la reconnaissance des ARNm par les ribosomes dans des conditions limitantes en eIF4E [8] et empêchent la reconnaissance des ARNm par la protéine MDA5, qui induit les mécanismes de l’immunité innée [6].
Mécanismes d’acquisition de la coiffe chez les virus
Les virus utilisent la machinerie de la cellule infectée afin de traduire leur ARNm en protéines. Bien que certains virus possèdent des structures IRES (internal ribosome entry site) permettant la traduction des ARNm en absence de structure coiffe [9], la plupart des virus coiffent l’extrémité 5’ de leurs ARNm afin de permettre leur traduction par les ribosomes cellulaires. Trois procédés permettant l’acquisition d’une coiffe par les ARNm viraux ont été décrits [2]. Les virus dont les ARNm sont synthétisés par l’ARN Pol II utilisent les enzymes cellulaires pour la formation de la coiffe. C’est le cas des rétrovirus et de la plupart des virus à ADN (Hepadnaviridae et Parvoviridae) à l’exception des Poxviridae et Herpesviridae. Une stratégie alternative consiste à subtiliser la coiffe des ARNm cellulaires déjà coiffés dans un processus appelé « vol de coiffe » (cap-snatching). Cette stratégie, utilisée notamment par les Orthomyxoviridae, les Arenaviridae et les Bunyaviridae, implique une endonucléase qui clive les ARNm cellulaires à des sites situés quelques nucléotides en aval de la structure coiffe [10]. Ces petits fragments d’ARN sont ensuite utilisés comme amorces par l’ARN polymérase ARN-dépendante virale qui synthétise les ARNm viraux. Enfin, la dernière stratégie utilisée par les virus consiste à coder et synthétiser la machinerie enzymatique nécessaire à la formation de leur structure coiffe (Figure 2B et 2C). Cette stratégie est notamment utilisée par la plupart des virus à ARN dont le cycle réplicatif est cytoplasmique. Bien que les machineries enzymatiques virales impliquées dans la formation de la coiffe soient extrêmement divergentes du point de vue de l’organisation structurale, des mécanismes réactionnels et de la fonctionnalité des enzymes, les coiffes virales sont identiques aux coiffes cellulaires, et l’inhibition ou la mutation des enzymes virales a des effets délétères sur la réplication virale [2].
Les virus utilisent des stratégies originales de formation de la coiffe
Lorsque les virus synthétisent leur coiffe, deux alternatives au mécanisme canonique ont été découvertes. Chez les Vésiculovirus (Figure 2B), une polyribonucléotidyltransférase (PRNTase) (domaine de la protéine L), remplaçant la GTase, forme une liaison covalente avec l’extrémité 5’ des ARN naissants (au lieu du GMP) via une histidine catalytique. La PRNTase liée à l’ARN transfère ensuite une molécule de GDP sur l’ARN formant ainsi la coiffe qui sera méthylée successivement en position 2’O et N7 par la 2’O-MTase et le domaine MTase de la protéine L, respectivement [11]. Chez les Alphavirus par contre, la N7-MTase virale méthyle le GTP avant de former un intermédiaire covalent avec le 7MeGMP et de le transférer à l’extrémité 5’ de l’ARN (Figure 2C) [12]. Dans le cas des Alphavirus, la coiffe synthétisée ne semble pas méthylée en 2’O du premier nucléotide transcrit.
Organisation topologique des activités enzymatiques de formation de la coiffe
Chez les eucaryotes inférieurs comme la levure, les trois activités nécessaires à la formation de la coiffe sont localisées sur des protéines distinctes alors que chez les eucaryotes supérieurs, les activités RTPase et GTase sont regroupées sur une seule protéine appelée capping enzyme [1, 2]. De même, chez certains virus comme les Orbivirus de la famille des Reoviridae (représenté par le blue tongue virus), l’ensemble des activités enzymatiques nécessaires à la formation de la structure coiffe-1 est porté par quatre domaines fonctionnels regroupés sur une protéine unique, la protéine VP4 (viral protein 4) [13]. VP4 forme une chaîne d’assemblage moléculaire localisée à proximité de l’ARN polymérase qui coiffe les ARN au moment de leur synthèse.
Chez d’autres virus, comme les Poxvirus (représentés par le virus de la vaccine [VV]) ou les Mimivirus, une protéine unique regroupe les domaines RTPase, GTase et N7-MTase, permettant la synthèse coordonnée de la structure coiffe-0. Dans le cas du VV, le domaine amino-terminal (NTD) (1-545) de la protéine D1 regroupe les activités RTPase et GTase [14] alors que le domaine carboxy-terminal (CTD) de la protéine porte l’activité N7-MTase qui est activée par la formation d’un complexe avec la protéine virale D12 [15]. L’activité 2’O-MTase dépend d’une seconde protéine, la protéine VP39 [3].
Enfin, chez plusieurs virus, le génome code, d’une part pour une protéine portant l’activité RTPase et, d’autre part, pour une protéine catalysant à la fois le transfert du groupement guanyle et la méthylation de la coiffe en position N7 et/ou 2’O. C’est notamment le cas des Rotavirus [16], des Réovirus, des Alphavirus [12] et probablement des Flavivirus [17, 18]. Chez ces derniers, les activités N7-MTase et 2’O-MTase sont localisées dans le NTD bifonctionnel de la protéine NS5 [18], tandis que le CTD porte l’activité ARN polymérase ARN-dépendante. Cette organisation suggère un couplage entre l’activité polymérase et la formation de la coiffe, comme c’est le cas chez les eucaryotes.
Structures et mécanismes des ARN triphosphatases (RTPases)
Les RTPases catalysent la première étape de la réaction de formation de la coiffe en hydrolysant le phosphate γ de l’ARNm. On distingue deux familles de RTPases. D’une part, les RTPases dont l’activité dépend de cations divalents (Figure 3A, 3B, 3C) et, d’autre part, les enzymes appartenant à la superfamille des cystéines phosphatases qui sont indépendantes des ions (Figure 3D).
 |
Figure 3. Structures des quatre classes canoniques de RTPases virales. Les structures des protéines sont représentées sous forme de rubans. Le détail des sites catalytiques est présenté sous chaque structure. Les chaînes latérales des résidus du site actif sont représentées en « bâtons ». Le code couleur de la représentation des chaînes latérales est : bleu : azote ; gris : carbone ; orange : phosphate ; rouge : oxygène. A. Structure de la RTPase de Mimivirus avec une architecture TTM (PDB [protein data bank] : 2QZE), dont l’activité est dépendante de cations divalents. B. Structure quaternaire de la protéine NSP2 de Rotavirus avec un repliement de type HIT-like (PDB : 1L9V). C. Structure du domaine RTPase/ARN hélicase de la protéine NS3 du virus de la dengue (PDB : 2BHR). D. Structure de la protéine BVP de Baculovirus (PDB : 1YN9). |
La famille des RTPases dont l’activité dépend de cations divalents
Cette famille se divise en trois sous-familles structurales : les triphosphatase tunnel metalloenzymes (TTM), les repliements HIT-like (histidine triad-like) et les repliements RTPases/hélicases.
Parmi les RTPases de type TTM, l’exemple le mieux étudié est Cet1 de S. cerevisiae dont le site actif est composé de plus de 15 groupes fonctionnels qui coordonnent l’ion métallique et le phosphate γ de l’ARNm (Figure 3A). Cette enzyme dimérique est caractérisée par une architecture en tunnel constituée de huit feuillets β antiparallèles. Les RTPases des virus à ADN telles que les protéines D1 du VV, PBCV-1 (Paramecium bursania chlorella virus-1) de Chlorella virus et la RTPase de Mimivirus appartiennent à la famille des TTM [19]
Le repliement HIT-like a été identifié dans la protéine NSP2 (nonstructural protein 2) de Rotavirus. Ce repliement unique se compose d’un NTD hélical et d’un CTD de type α/β homologue à des hydrolases cellulaires. Le site de liaison du nucléotide est constitué d’une histidine et d’un ion magnésium localisé dans un sillon compris entre les deux domaines (Figure 3B) [20].
L’analyse des repliements des RTPases virales a révélé l’existence d’une troisième classe de RTPases (Figure 3C) dont l’activité est associée à l’activité hélicase. La protéine NS3 (nonstructural protein 3) des Flavivirus, la protéine nsp13 des Coronavirus, la protéine lambda1 des Réovirus et la protéine nsP2 des Alphavirus appartiennent à cette famille [21].
Les RTPases indépendantes des ions
Ces enzymes appartiennent à la superfamille des cystéines phosphatases et ont été identifiées chez les plantes et les métazoaires (Figure 3D). Ces RTPases catalysent l’hydrolyse du phosphate en formant un intermédiaire covalent avec une cystéine localisée dans la séquence consensus HCXXXXXR(S/T). Ces RTPases sont caractérisées du point de vue structural par un domaine central conservé et composé de cinq feuillets β flanqués d’hélices α [22]. On retrouve des RTPases appartenant à cette famille chez les Baculovirus (Baculovirus phosphatases ou BVP) [23].
Structures et mécanismes catalytiques des guanylyltransférases (GTases)
Parmi les enzymes participant à la formation de la structure coiffe, les GTases restent les enzymes les moins bien caractérisées. L’étude des GTases chez le Chlorella virus (Figure 4A) et chez la levure (Figure 4B et 4C) démontre des homologies structurales importantes et a révélé la présence de deux domaines structuraux distincts : un domaine NTD du type nucleotidyl transferase (NT) et un domaine CTD homologue à celui des ligases à ADN [24]. Ces GTases sont caractérisées par un motif conservé KXDGI/L localisé 17 à 18 acides aminés en aval d’un motif riche en proline. La lysine catalytique forme un intermédiaire covalent avec le GMP avant de le transférer dans une seconde étape sur l’extrémité 5’ de l’ARN diphosphate. Bien que la capacité à former un lien covalent avec le GMP semble être une propriété de nombreuses GTases virales, le motif canonique KXDGI/L n’est pas strictement conservé [2].
 |
Figure 4. Structures de GTases eucaryotes. Les structures des protéines sont représentées sous forme de rubans de couleur orange et les résidus impliqués dans la catalyse sont représentés sous forme de bâtons. Le code couleur des atomes des chaînes latérales est identique à celui de la Figure 3. Les trois premiers panneaux présentent des structures homologues. A. GTase Ceg-1 de S. cerevisiae (PDB : 3KYH) en conformation ouverte avant le recrutement du GTP. B. GTase Cgt-1 de C. Albicans en présence de GTP (PDB : 1P16) en conformation ouverte (correspondant à l’étape de recrutement du GTP). C. GTase de Chlorella virus en présence de GTP (PDB : 1CKM), en conformation fermée. D. GTase de Réovirus (PDB : 1EJ6, résidus 2 à 383), révélant un repliement alternatif aux GTases canoniques présentées en A, B et C. |
Récemment, deux nouveaux repliements de GTases ont été identifiés chez les Orbivirus [13] et les Réovirus [25] (Figure 4D). L’activité GTase de ces deux protéines implique également la formation d’un lien covalent avec une molécule de GMP, mais la lysine catalytique a été identifiée uniquement chez les Réovirus.
Chez les Vésiculovirus et les Alphavirus (voir plus haut et Figure 2), il existe probablement des mécanismes catalytiques différents, mais ces enzymes n’ont pas été encore caractérisées d’un point de vue structural.
Structures et mécanismes des N7- et 2’O-méthyltransférases (MTases)
Les N7-MTases et/ou 2’O-MTases virales finalisent le processus de formation de la coiffe en synthétisant les structures coiffe-0 et coiffe-1. Chez les eucaryotes inférieurs, il n’y a pas de 2’O-méthylation de la coiffe, et seule la structure coiffe-0 a été décrite. Toutes les MTases impliquées dans la formation de la coiffe utilisent le SAM comme donneur de méthyl générant comme produit secondaire de réaction la molécule de SAH (S-adénosyl-L-homocystéine). Bien que les séquences primaires des différentes MTases soient très variables, leurs structures indiquent que leur cœur catalytique est organisé de manière très similaire (Figure 5). Il est dans tous les cas composé de sept feuillets β encadrés par des hélices α, formant une poche accommodant le SAM alors que des éléments structuraux additionnels assurent la reconnaissance spécifique des substrats.
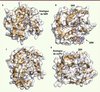 |
Figure 5. Structures des N7- et 2’O-MTases. La figure montre les repliements de N7-MTases (A et B) et 2’O-MTases (C et D). Les structures des protéines sont représentées sous forme de rubans et le code couleur est identique à celui de la Figure 3. La surface transparente permet de visualiser les cavités des sites actifs. Les molécules d’analogues de SAM sont représentées au centre du site actif. A. N7-MTase de Encephalitozoon Cuniculi (PDB : 1Z3C) en présence d’un analogue du SAM. B. 2’O-MTase VP39 (PDB : 1AV6) du virus de la vaccine en complexe avec un ARNm et en présence de SAH. C. N7-MTase (protéine D1) du virus de la vaccine (PDB : 2VDW) en présence de SAH. D. N7- et 2’O-MTase (protéine NS5) du virus de la dengue (PDB : 2P3Q) en complexe avec un analogue de la structure coiffe et de SAH. |
La structure a été déterminée pour de nombreuses MTases cellulaires et virales impliquées dans la formation de la coiffe [13, 17, 26, 27]. Leurs structures correspondent au repliement canonique et révèlent (1) la présence d’un site spécifique de fixation du SAM à proximité immédiate des résidus catalytiques, (2) une poche permettant la fixation spécifique de la coiffe, et (3) un sillon capable d’accommoder l’ARN.
Les études structurales sur les complexes entre des 2’O-MTases et des ARNm ou des analogues de coiffe ont permis de mieux comprendre les mécanismes de reconnaissance des structures coiffe-0 par les 2’O-MTases (MTase de VP39 [3], VP4 [13] et NS5 [17]). En effet, les structures obtenues en présence d’analogues de coiffe indiquent que les molécules de 7MeGpppN pénètrent dans une poche hydrophobe où elles sont maintenues par des résidus aromatiques. L’ARNm en aval de la coiffe se positionne dans une gorge chargée positivement de sorte que le groupement 2’-O du ribose du premier nucléotide de l’ARNm est localisé en face du donneur de méthyl. Le mécanisme catalytique implique une tétrade catalytique KDKE conservée [26] qui active le groupement 2’O du ribose et permet l’attaque nucléophile du groupement méthyl, et son transfert final sur la position 2’O du ribose [28].
Les bases moléculaires de la reconnaissance spécifique de la structure coiffe non méthylée en position N7 (GpppN) par les N7-MTases semblent différentes de celles déterminant la fixation du 7MeGTP par les 2’O-MTases, suggérant un mécanisme catalytique spécifique. Dans ce cas, les structures de N7-MTases en complexe avec des 7MeGpppARN [15, 29] indiquent que l’orientation optimale de la coiffe est assurée par un réseau d’interactions électrostatiques spécifiques qui positionnent le N7 du groupement guanylate à proximité immédiate du SAM.
Enfin, il est important de noter que des facteurs régulateurs de l’activité MTase (protéines virales jouant le rôle de cofacteur) ont été identifiés chez le VV et les Coronavirus [15, 30]. Le rôle physiologique de ces facteurs régulateurs, qui permettent l’activation de l’activité MTase, reste mal compris.
Conclusion et perspectives
L’acquisition de la structure coiffe par les ARN messagers viraux est essentielle à la propagation de l’infection virale. Ainsi, elle permet l’expression des protéines virales par les ribosomes et limite la reconnaissance des ARN viraux par des sentinelles de l’immunité innée. Le rôle crucial de cette structure a notamment été démontré par des études de génétique inverse sur des enzymes virales impliquées dans la formation de la coiffe [2]. L’inhibition spécifique des enzymes de formation de la coiffe devrait par conséquent bloquer la réplication virale. Leur étude structurale et fonctionnelle a révélé des mécanismes originaux ouvrant la voie au développement de nouvelles stratégies antivirales.
Conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
Références
- Bisaillon M. La structure-coiffe des ARN messagers. Med Sci (Paris) 2001 ; 17 : 312–319. [CrossRef] [Google Scholar]
- Decroly E, Ferron F, Lescar J, Canard B. Conventional and unconventional mechanisms for capping viral mRNA. Nat Rev Microbiol 2011 ; 10 : 51–65. [CrossRef] [PubMed] [Google Scholar]
- Quiocho FA, Hu G, Gershon PD. Structural basis of mRNA cap recognition by proteins. Curr Opin Struct Biol 2000 ; 10 : 78–86. [CrossRef] [PubMed] [Google Scholar]
- Parent A, Bisaillon M. Synergie entre les complexes de transcription et de maturation des ARN messagers. Med Sci (Paris) 2006 ; 22 : 626–632. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Kowalinski E, Lunardi T, McCarthy AA, et al. Structural basis for the activation of innate immune pattern-recognition receptor RIG-I by viral RNA. Cell 2011 ; 147 : 423–435. [CrossRef] [PubMed] [Google Scholar]
- Zust R, Cervantes-Barragan L, Habjan M, et al. Ribose 2’-O-methylation provides a molecular signature for the distinction of self and non-self mRNA dependent on the RNA sensor Mda5. Nat Immunol 2011 ; 12 : 137–143. [CrossRef] [PubMed] [Google Scholar]
- Ghosh A, Shuman S, Lima CD. Structural insights to how mammalian capping enzyme reads the CTD code. Mol Cell 2011 ; 43 : 299–310. [CrossRef] [PubMed] [Google Scholar]
- Kuge H, Brownlee GG, Gershon PD, Richter JD. Cap ribose methylation of c-mos mRNA stimulates translation and oocyte maturation in Xenopus laevis. Nucleic Acids Res 1998 ; 26 : 3208–3214. [CrossRef] [PubMed] [Google Scholar]
- Fitzgerald KD, Semler BL. Bridging IRES elements in mRNAs to the eukaryotic translation apparatus. Biochim Biophys Acta 2009 ; 1789 : 518–528. [PubMed] [Google Scholar]
- Dias A, Bouvier D, Crepin T, et al. The cap-snatching endonuclease of influenza virus polymerase resides in the PA subunit. Nature 2009 ; 458 : 914–918. [CrossRef] [PubMed] [Google Scholar]
- Li J, Wang JT, Whelan SP. A unique strategy for mRNA cap methylation used by vesicular stomatitis virus. Proc Natl Acad Sci USA 2006 ; 103 : 8493–8498. [CrossRef] [Google Scholar]
- Ahola T, Laakkonen P, Vihinen H, Kaariainen L. Critical residues of Semliki Forest virus RNA capping enzyme involved in methyltransferase and guanylyltransferase-like activities. J Virol 1997 ; 71 : 392–397. [PubMed] [Google Scholar]
- Sutton G, Grimes JM, Stuart DI, Roy P. Bluetongue virus VP4 is an RNA-capping assembly line. Nat Struct Mol Biol 2007 ; 14 : 449–451. [CrossRef] [PubMed] [Google Scholar]
- Shuman S. Catalytic activity of vaccinia mRNA capping enzyme subunits coexpressed in Escherichia coli. J Biol Chem 1990 ; 265 : 11960–11966. [PubMed] [Google Scholar]
- De la Pena M, Kyrieleis OJ, Cusack S. Structural insights into the mechanism and evolution of the vaccinia virus mRNA cap N7 methyl-transferase. EMBO J 2007 ; 26 : 4913–4925. [CrossRef] [PubMed] [Google Scholar]
- Zhang X, Walker SB, Chipman PR, et al. Reovirus polymerase lambda 3 localized by cryo-electron microscopy of virions at a resolution of 7.6 A. Nat Struct Biol 2003 ; 10 : 1011–1018. [CrossRef] [PubMed] [Google Scholar]
- Egloff MP, Decroly E, Malet H, et al. Structural and functional analysis of methylation and 5’-RNA sequence requirements of short capped RNAs by the methyltransferase domain of dengue virus NS5. J Mol Biol 2007 ; 372 : 723–736. [CrossRef] [PubMed] [Google Scholar]
- Ray D, Shah A, Tilgner M, et al. West Nile virus 5’-cap structure is formed by sequential guanine N-7 and ribose 2’-O methylations by nonstructural protein 5. J Virol 2006 ; 80 : 8362–8370. [CrossRef] [PubMed] [Google Scholar]
- Bisaillon M, Shuman S. Structure-function analysis of the active site tunnel of yeast RNA triphosphatase. J Biol Chem 2001 ; 276 : 17261–17266. [CrossRef] [PubMed] [Google Scholar]
- Vasquez-Del Carpio R, Gonzalez-Nilo FD, Riadi G, et al. Histidine triad-like motif of the rotavirus NSP2 octamer mediates both RTPase and NTPase activities. J Mol Biol 2006 ; 362 : 539–554. [CrossRef] [PubMed] [Google Scholar]
- Benarroch D, Selisko B, Locatelli GA, et al. The RNA helicase, nucleotide 5’-triphosphatase, and RNA 5’-triphosphatase activities of dengue virus protein NS3 are Mg2+-dependent and require a functional Walker B motif in the helicase catalytic core. Virology 2004 ; 328 : 208–218. [CrossRef] [PubMed] [Google Scholar]
- Changela A, Ho CK, Martins A, et al. Structure and mechanism of the RNA triphosphatase component of mammalian mRNA capping enzyme. EMBO J 2001 ; 20 : 2575–2586. [CrossRef] [PubMed] [Google Scholar]
- Martins A, Shuman S. Mechanism of phosphoanhydride cleavage by baculovirus phosphatase. J Biol Chem 2000 ; 275 : 35070–35076. [CrossRef] [PubMed] [Google Scholar]
- Hakansson K, Doherty AJ, Shuman S, Wigley DB. X-ray crystallography reveals a large conformational change during guanyl transfer by mRNA capping enzymes. Cell 1997 ; 89 : 545–553. [CrossRef] [PubMed] [Google Scholar]
- Reinisch KM, Nibert ML, Harrison SC. Structure of the reovirus core at 3.6 A resolution. Nature 2000 ; 404 : 960–967. [CrossRef] [PubMed] [Google Scholar]
- Hodel AE, Gershon PD, Shi X, Quiocho FA. The 1.85 A structure of vaccinia protein VP39: a bifunctional enzyme that participates in the modification of both mRNA ends. Cell 1996 ; 85 : 247–256. [CrossRef] [PubMed] [Google Scholar]
- Decroly E, Debarnot C, Ferron F, et al. Crystal structure and functional analysis of the SARS-coronavirus RNA cap 2’-O-methyltransferase nsp10/nsp16 complex. PLoS Pathog 2011 ; 7 : e1002059. [CrossRef] [PubMed] [Google Scholar]
- Li C, Xia Y, Gao X, Gershon PD. Mechanism of RNA 2’-O-methylation: evidence that the catalytic lysine acts to steer rather than deprotonate the target nucleophile. Biochemistry 2004 ; 43 : 5680–5687. [CrossRef] [PubMed] [Google Scholar]
- Fabrega C, Hausmann S, Shen V, et al. Structure and mechanism of mRNA cap (guanine-N7) methyltransferase. Mol Cell 2004 ; 13 : 77–89. [CrossRef] [PubMed] [Google Scholar]
- Bouvet M, Debarnot C, Imbert I, et al. In vitro reconstitution of SARS-coronavirus mRNA cap methylation. PLoS Pathog 2010 ; 6 : e1000863. [CrossRef] [PubMed] [Google Scholar]
- Kowalinski E, Louber J, Gerlier D, Cusack S. RIG-I : un commutateur moléculaire détecteur d’ARN viral. Med Sci (Paris) 2012 ; 28 : 136–138. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Structure de la coiffe. La structure coiffe-0 (fond bleu) est composée d’un résidu guanosine méthylé en position N7 (cercle vert à gauche) qui est lié par une liaison 5’-5’ triphosphate (fond rose) au nucléotide en aval de la coiffe. Un groupement méthyl (cercle vert à droite) peut être ajouté à la position 2’O du ribose du premier nucléotide de l’ARNm pour former la coiffe-1. |
| Dans le texte | |
 |
Figure 2. Différents mécanismes de formation de la coiffe. A. Ce panneau représente le mécanisme « conventionnel » de formation de la coiffe chez les eucaryotes. Le phosphate γ à l’extrémité 5’ de l’ARN pré-messager est hydrolysé par l’ARN triphosphatase (RTPase). L’ARN guanylyltransférase (GTase) fixe ensuite une molécule de GMP à partir du GTP (Gppp) en libérant du pyrophosphate inorganique (PPi). La GTase transfère le GMP sur l’extrémité diphosphate de l’ARN prémessager formant une liaison 5’-5’ triphosphate. La synthèse de la coiffe se termine par la méthylation du résidu guanosine en position N7 par la N7-MTase à partir du donneur de méthyl (SAM). Enfin, chez les eucaryotes supérieurs, la formation de la structure coiffe-1 fait intervenir une 2’O-MTase qui méthyle la position 2’O du ribose du premier nucléotide de l’ARN. B.Ce panneau illustre le mécanisme de formation de la coiffe chez les Vésiculovirus. La PRNTase recrute l’ARN triphosphate pour former un lien covalent avec l’ARN 5’ diphosphate et le transfère ensuite sur une molécule de GDP (Gpp). L’ordre de la séquence de méthylation est inversé par rapport au mécanisme canonique retrouvé chez les eucaryotes (A.). C. Ce panneau illustre le mécanisme de coiffe des Alphavirus où la N7-méthylation précède le transfert du GMP sur l’ARN diphosphate. |
| Dans le texte | |
 |
Figure 3. Structures des quatre classes canoniques de RTPases virales. Les structures des protéines sont représentées sous forme de rubans. Le détail des sites catalytiques est présenté sous chaque structure. Les chaînes latérales des résidus du site actif sont représentées en « bâtons ». Le code couleur de la représentation des chaînes latérales est : bleu : azote ; gris : carbone ; orange : phosphate ; rouge : oxygène. A. Structure de la RTPase de Mimivirus avec une architecture TTM (PDB [protein data bank] : 2QZE), dont l’activité est dépendante de cations divalents. B. Structure quaternaire de la protéine NSP2 de Rotavirus avec un repliement de type HIT-like (PDB : 1L9V). C. Structure du domaine RTPase/ARN hélicase de la protéine NS3 du virus de la dengue (PDB : 2BHR). D. Structure de la protéine BVP de Baculovirus (PDB : 1YN9). |
| Dans le texte | |
 |
Figure 4. Structures de GTases eucaryotes. Les structures des protéines sont représentées sous forme de rubans de couleur orange et les résidus impliqués dans la catalyse sont représentés sous forme de bâtons. Le code couleur des atomes des chaînes latérales est identique à celui de la Figure 3. Les trois premiers panneaux présentent des structures homologues. A. GTase Ceg-1 de S. cerevisiae (PDB : 3KYH) en conformation ouverte avant le recrutement du GTP. B. GTase Cgt-1 de C. Albicans en présence de GTP (PDB : 1P16) en conformation ouverte (correspondant à l’étape de recrutement du GTP). C. GTase de Chlorella virus en présence de GTP (PDB : 1CKM), en conformation fermée. D. GTase de Réovirus (PDB : 1EJ6, résidus 2 à 383), révélant un repliement alternatif aux GTases canoniques présentées en A, B et C. |
| Dans le texte | |
 |
Figure 5. Structures des N7- et 2’O-MTases. La figure montre les repliements de N7-MTases (A et B) et 2’O-MTases (C et D). Les structures des protéines sont représentées sous forme de rubans et le code couleur est identique à celui de la Figure 3. La surface transparente permet de visualiser les cavités des sites actifs. Les molécules d’analogues de SAM sont représentées au centre du site actif. A. N7-MTase de Encephalitozoon Cuniculi (PDB : 1Z3C) en présence d’un analogue du SAM. B. 2’O-MTase VP39 (PDB : 1AV6) du virus de la vaccine en complexe avec un ARNm et en présence de SAH. C. N7-MTase (protéine D1) du virus de la vaccine (PDB : 2VDW) en présence de SAH. D. N7- et 2’O-MTase (protéine NS5) du virus de la dengue (PDB : 2P3Q) en complexe avec un analogue de la structure coiffe et de SAH. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




