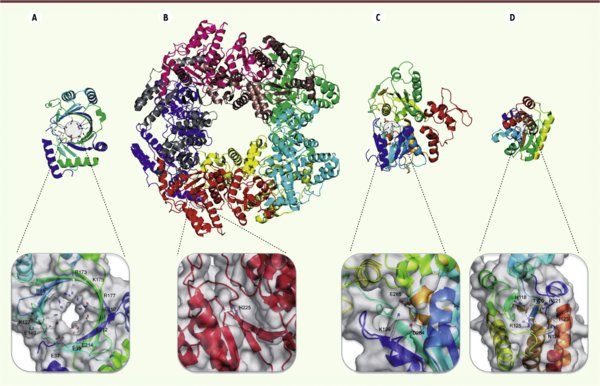
Figure 3.
Télécharger l'image originale
Structures des quatre classes canoniques de RTPases virales. Les structures des protéines sont représentées sous forme de rubans. Le détail des sites catalytiques est présenté sous chaque structure. Les chaînes latérales des résidus du site actif sont représentées en « bâtons ». Le code couleur de la représentation des chaînes latérales est : bleu : azote ; gris : carbone ; orange : phosphate ; rouge : oxygène. A. Structure de la RTPase de Mimivirus avec une architecture TTM (PDB [protein data bank] : 2QZE), dont l’activité est dépendante de cations divalents. B. Structure quaternaire de la protéine NSP2 de Rotavirus avec un repliement de type HIT-like (PDB : 1L9V). C. Structure du domaine RTPase/ARN hélicase de la protéine NS3 du virus de la dengue (PDB : 2BHR). D. Structure de la protéine BVP de Baculovirus (PDB : 1YN9).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




