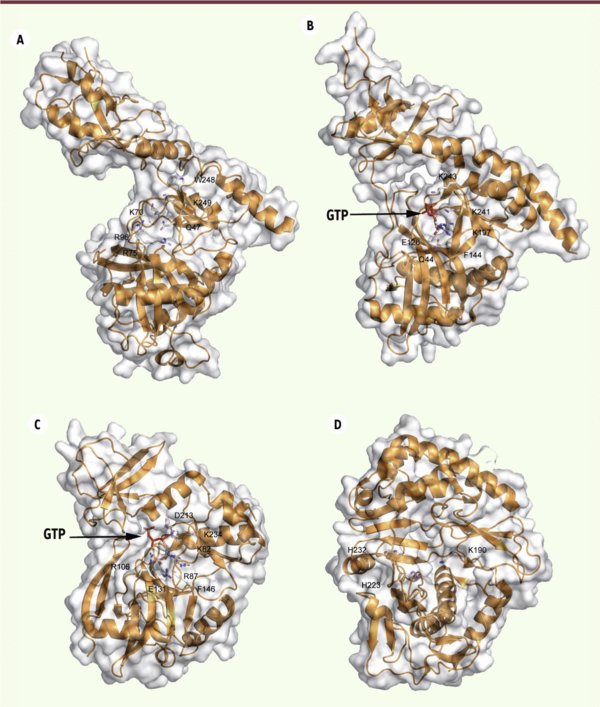
Figure 4.
Télécharger l'image originale
Structures de GTases eucaryotes. Les structures des protéines sont représentées sous forme de rubans de couleur orange et les résidus impliqués dans la catalyse sont représentés sous forme de bâtons. Le code couleur des atomes des chaînes latérales est identique à celui de la Figure 3. Les trois premiers panneaux présentent des structures homologues. A. GTase Ceg-1 de S. cerevisiae (PDB : 3KYH) en conformation ouverte avant le recrutement du GTP. B. GTase Cgt-1 de C. Albicans en présence de GTP (PDB : 1P16) en conformation ouverte (correspondant à l’étape de recrutement du GTP). C. GTase de Chlorella virus en présence de GTP (PDB : 1CKM), en conformation fermée. D. GTase de Réovirus (PDB : 1EJ6, résidus 2 à 383), révélant un repliement alternatif aux GTases canoniques présentées en A, B et C.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




