| Numéro |
Med Sci (Paris)
Volume 41, Numéro 6-7, Juin-Juillet 2025
|
|
|---|---|---|
| Page(s) | 561 - 569 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2025094 | |
| Publié en ligne | 7 juillet 2025 | |
Les protéines cellulaires de liaison à l’ARN et leur interaction avec les ARN viraux
Facteurs clés et méthodes pour l’identification de nouvelles cibles
Cellular RNA-binding proteins during viral infection: known factors and new approaches to identify novel targets
Université de Strasbourg, architecture et réactivité de l’ARN, Institut de biologie moléculaire et cellulaire, CNRS UPR9002, Strasbourg, France
*
e.girardi@ibmc-cnrs.unistra.fr
Les virus dépendent des machineries de leur hôte pour assurer la réplication de leur génome. Plusieurs virus émergents responsables des épidémies récentes ont un génome ARN simple brin de polarité positive. Dans ce cas, l’ARN viral sert de matrice pour la réplication et de messager pour la production des protéines virales. Les interactions entre ces ARN viraux et les protéines de liaison à l’ARN de l’hôte influencent le cycle viral. Les protéines de liaison à l’ARN peuvent inhiber ou favoriser l’infection en modifiant ou en interagissant directement avec les ARN cellulaires et viraux. Les techniques modernes ont permis d’approfondir la compréhension de ces interactions, laissant entrevoir de futures pistes thérapeutiques. Cette synthèse se focalise sur les protéines cellulaires de liaison à l’ARN ayant une activité antivirale et les approches actuellement mises en œuvre permettant de découvrir de nouveaux facteurs cellulaires interagissant avec l’ARN génomique des virus à ARN simple brin de polarité positive.
Abstract
Viruses are obligate intracellular parasites that rely heavily on host components and metabolism to replicate their genome. Most of the emerging viruses responsible for recent epidemic have an RNA genome. This viral RNA serves both as a template for replication and as a messenger for the production of viral proteins. Interactions between viral RNA and host RNA-binding proteins play a crucial role in viral replication and immune evasion. Some RNA-binding proteins act as antiviral factors by recognizing and restricting foreign viral RNA, while others, once hijacked by the virus, support its replication. Identification these viral RNA-protein interactions is essential for the development of new antiviral strategies. This review explores the role of cellular RNA-binding proteins in the recognition, modification, and degradation of viral RNA, focusing on pathogenic human RNA viruses and new technologies for studying these interactions.
© 2025 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Vignette (© Erika Girardi).
Les virus sont des parasites intracellulaires obligatoires qui ont besoin des composants et du métabolisme de leur cellule hôte pour répliquer leur génome et produire de nouvelles particules infectieuses [1]. Le génome viral peut être constitué d’ADN ou d’ARN. Un certain nombre de virus à l’origine de récentes épidémies, comme le coronavirus du syndrome respiratoire aigu sévère de type 2 (SARS-CoV-2), le virus de la dengue (DENV) et le virus Zika (ZIKV), possèdent un génome ARN simple brin de polarité positive [2–4]. Cet ARN viral sert à la fois de matrice pour la réplication et d’ARN messager pour la traduction des protéines virales, parmi lesquelles une ARN polymérase ARN dépendante qui va synthétiser un ARN anti-génomique (de polarité négative) permettant de générer des nouvelles molécules d’ARN génomique [1]. L’appariement entre le génome et l’antigénome conduit à la formation d’un intermédiaire de réplication d’ARN double-brin qui représente, indépendamment de sa séquence, un signal de danger reconnu par le système immunitaire inné de l’hôte. Tout comme les ARN cellulaires, les ARN viraux interagissent avec des protéines possédant des domaines de liaison à l’ARN (RBP pour RNA-binding proteins) pour former des ribonucléoprotéines fonctionnelles. Ces protéines vont notamment permettre la maturation, la stabilité, la structure, ou encore la bonne localisation des ARN. Les effets des interactions entre les RBP cellulaires et les ARN viraux sont complexes : un grand nombre de ces RBP exercent des propriétés antivirales, grâce à la reconnaissance des caractéristiques spécifiques des ARN viraux reconnus comme non-soi (absence de coiffe, ou de queue polyA, intermédiaire de réplication en ARN double brin, etc.). Cependant, d’autres RBP favorisent la réplication virale et sont exploitées par les virus [5, 6]. La capacité des virus à exploiter les RBP provirales ou à échapper à celles antivirales est essentielle pour assurer leur réplication et leur propagation. C’est pourquoi l’identification et la caractérisation des interactions ARN viraux-RBP sont déterminantes pour la mise au point de nouvelles stratégies thérapeutiques, en particulier pour combattre les virus émergents.
Dans cette revue, nous décrirons comment les RBP cellulaires agissent dès les premiers stades de l’infection pour reconnaître, dégrader et modifier les ARN viraux, influençant ainsi le devenir de l’infection. Nous nous concentrerons, en particulier, sur les virus à ARN simple brin de polarité positive, pathogènes pour l’être humain et nous explorerons les nouvelles techniques qui permettent de mieux comprendre la diversité des interactions complexes entre les RBP et les ARN viraux.
Rôles des protéines cellulaires de liaison à l’ARN dans l’activation des réponses antivirales innées
Les protéines cellulaires de liaison à l’ARN jouant un rôle de senseurs
La distinction entre les acides nucléiques du soi et du non-soi repose sur leurs caractéristiques moléculaires telles que leur structure ou leurs modifications chimiques. Chez les vertébrés, les protéines PRR (pattern recognition receptors) détectent la présence de PAMP (pathogen-associated molecular pattern) qui se trouvent dans les ARN viraux [7] tels que la structure en ARN double brin, ou les extrémités 5’ triphosphate. La stimulation des PRR déclenche une cascade de signalisation qui aboutit à l’activation des mécanismes de l’immunité innée, notamment la voie du facteur nucléaire kappa B (NF-kB) et la production d’interférons (IFN) (Figure 1). Ces voies permettent l’expression de facteurs antiviraux, et peuvent également induire la mort cellulaire afin de limiter la propagation de l’infection virale [8] (→).
(→) Voir m/s n° 3, 2020, page 206
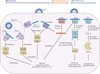 |
Figure 1 Reconnaissance des ARN exogènes. Les virus à ARN sont détectés par la cellule grâce à des protéines cellulaires de liaison aux ARN (RBPc) ayant un rôle de senseurs. TLR3/7/8 reconnaissent les ARN dans les endosomes. RIG-I et MDA5 détectent les ARN double brin (ARNdb) cytoplasmiques. Cette détection permet l’activation des réponses IFN et NF-kB. Les ARN double brin sont aussi détectés par des protéines cellulaires de liaison aux ARN effectrices telles que PKR et les OAS ce qui inhibe la traduction et conduit à une dégradation globale des ARN. Afin d’éviter un emballement de ces réponses, ADAR1 peut déstabiliser la structure des ARN double brin et éviter leur reconnaissance par les protéines cellulaires de liaison aux ARN senseurs et effectrices. |
Les Toll-like receptors (TLR)
Les TLR sont des glycoprotéines localisées au niveau de la membranes plasmique et endosomale [9]. Parmi les dix TLR identifiés chez l’homme, TLR7 et TLR8 reconnaissent les ARN simple brin et TLR3 reconnaît les ARN double brin (Figure 1) [7]. L’infection par le virus de l’encéphalomyocardite (EMCV) induit une réponse immunitaire IFN de type I (IFN-I) dépendante de TLR3, prévenant ainsi les défaillances myocardiques causées par le virus [10]. Cependant, le rôle protecteur de TLR3 reste à ce jour débattu [11]. Ce récepteur permet de limiter l’infection par le virus de la dengue (DENV) [12], mais peut avoir un rôle délétère lors de l’infection des cellules progénitrices neuronales par le virus Zika (ZIKV) [13].
Les RIG-I-like receptors (RLR)
Les protéines RIG-I (retinoic acid-inducible gene I receptor), MDA5 (melanoma differenciation-associated protein 5), et LGP2 (laboratory of genetics and physiology 2) constituent le groupe des RLR. Ces facteurs reconnaissent spécifiquement les ARN double brin grâce à leur domaine hélicase DExD/H-box [7, 14]. Contrairement à LGP2, RIG-I et MDA5 possèdent deux domaines de recrutement et d’activation des caspases (CARD) qui permettent la transduction du signal en aval. Cependant, RIG-I et MDA5 utilisent des mécanismes différents pour la reconnaissance des ARN viraux (Figure 1). RIG-I détecte des groupements triphosphate ou diphosphate aux extrémités 5’ de molécules d’ARN partiellement double brin [15] (→).
(→) Voir m/s n° 2, 2012, page 136
Il reconnaît l’ARN simple brin négatif des paramyxovirus, des rhabdovirus, des orthomyxovirus, des bunyavirus et des filovirus [16–18]. RIG-I détecte aussi l’ARN simple brin positif de quelques virus comme ceux des flavivirus DENV et ZIKV [19]. MDA5 détecte les duplex ininterrompus d’ARN, longs de plusieurs centaines de paires de bases [20]. En formant des filaments le long des ARN double brin, MDA5 initie la signalisation et l’activation de la voie conduisant à la production d’IFN-I [21]. MDA5 est requis pour l’immunité antivirale contre les picornavirus [17], les flavivirus [22], et les coronavirus [23]. Le troisième membre de la famille des RLR, LGP2, ne possède pas de domaine CARD et n’a donc pas la capacité de transduire de signaux [7, 14]. Selon certaines études LGP2 pourrait intervenir dans le déclenchement ou l’atténuation de la réponse immunitaire antivirale, en inhibant RIG-I [24], ou en activant MDA5 [25], respectivement.
Les protéines cellulaires de liaison à l’ARN jouant un rôle d’effecteurs
Les acides nucléiques d’origine virale présents dans la cellule peuvent aussi être reconnus par des RBP cellulaires qui peuvent être à la fois des senseurs et des effecteurs de la réponse antivirale. Ces RBP n’induisent pas de réponse antivirale par l’activation de la transcription de gènes du système immunitaire, mais peuvent agir soit directement sur la stabilité ou la traduction des ARN viraux, soit indirectement en activant d’autres protéines effectrices [26].
La protéine kinase R
La protéine kinase R (PKR) est une protéine kinase à sérine/thréonine qui est produite à l’état basal sous la forme d’un monomère inactif. Sa fixation aux ARN double brin (Figure 1) induit son activation par dimérisation et autophosphorylation. Une fois activée, PKR va inhiber la synthèse globale des protéines cellulaires et virales, réduisant ainsi la propagation du virus. Elle joue donc un rôle clé dans la réponse immunitaire antivirale [27], ce qui est corroboré par la capacité de certains virus à contrecarrer son action. Par exemple, le virus de la fièvre de la vallée du Rift (RVFV) est capable, via l’action de la protéine virale NSs, de cibler PKR pour induire sa dégradation [28–29] (→).
(→) Voir m/s n° 6-7, 2021, page 601
Les protéines OAS (2’-5’ oligoadénylate synthétase)
Les protéines OAS (2’-5’ oligoadénylate synthétase) humaines, OAS1, OAS2, OAS3 et OASL, sont constitutivement exprimées à de faibles niveaux dans le cytoplasme des cellules et sont activées en réponse à la présence d’ARN double brin viral [30] (Figure 1). Une fois activées, elles synthétisent le 2’,5’-oligoadénylate (2’,5’-A) [7, 26]. Ce messager secondaire active la ribonucléase L (RNase L), qui va cliver les ARN cellulaires et les ARN viraux, impactant ainsi négativement la réplication virale [31] (→).
(→) Voir m/s n° 10, 2008, page 859
De manière similaire à PKR, certains coronavirus et rotavirus expriment des protéines appartenant à la famille des phosphodiestérases qui sont capables de cliver les molécules de 2’,5’-A et d’empêcher ainsi l’activation de la RNase L et la réponse antivirale [32].
Les protéines cellulaires de liaison à l’ARN qui modifient l’ARN viral
Au cours de l’évolution, les virus ont acquis des propriétés d’échappement aux mécanismes de défense de l’hôte, souvent en détournant les fonctions primaires de RBP cellulaires. Parmi celles-ci, on retrouve des protéines de modification des ARN. Les modifications des bases ou du ribose peuvent changer les propriétés de l’ARN et affecter plusieurs étapes de son métabolisme comme son épissage, sa stabilité, sa localisation ou sa traduction [33, 34]. Il n’est donc pas surprenant que les virus utilisent ces mécanismes pour modifier leur génome et/ou leurs transcrits dans le but d’éviter leur reconnaissance par le système immunitaire antiviral de l’hôte.
L’édition de bases adénosine en inosine (A-en-I)
L’édition de bases adénosine en inosine (A-en-I) constitue l’une des modifications de l’ARN les mieux caractérisées. Depuis la fin des années 1980, l’amélioration des techniques de séquençage a permis de mettre en évidence des événements de substitution des bases adénosine en inosine dans de nombreux virus tels que le virus de la rougeole (MV), les virus influenza ou encore RVFV [35]. Ces modifications sont effectuées dans les molécules d’ARN double brin par les trois enzymes ADAR (adenosine deaminase acting on RNA), ADAR1, ADAR2 et ADAR3. ADAR1 est une enzyme qui est présente de façon constitutive dans le noyau cellulaire, mais dont une forme cytoplasmique peut apparaître après exposition des cellules à l’IFN-I. Les conséquences de l’édition A-en-I des ARN cellulaires par ADAR1 sont doubles. Lorsque l’édition se produit dans une région codante, les inosines seront mal interprétées par la machinerie de traduction et seront lues comme des guanosines (G). Par ailleurs, la conversion A-en-I déstabilise les structures des ARN double brin empêchant ainsi leur reconnaissance par les récepteurs anti-viraux cytoplasmiques, comme les RLR ou PKR (Figure 1) [36]. En éditant les ARN cellulaires, les protéines ADAR jouent donc un rôle clé dans la discrimination entre le soi et le non-soi. Cependant, la contribution de ces enzymes à la défense antivirale reste peu claire [35]. La présence de mutations A-en-G dépendantes d’ADAR1 a été décrite pour une large gamme de virus. Cela peut potentiellement mener à la synthèse de protéines virales dysfonctionnelles et déstabiliser les ARN double brin intermédiaires de réplication. Néanmoins, ADAR1 semble également jouer un rôle proviral en atténuant la réponse du système immunitaire inné. Dans le cas du virus de l’hépatite delta (HDV), le rôle proviral d’ADAR1 est plus évident car la synthèse d’une isoforme longue d’une protéine virale indispensable à la réplication du virus dépend de l’édition d’un codon STOP en codon tryptophane [37].
La modification N6-méthyladénosine (m6A)
Chez les mammifères, la N6-méthyladénosine (m6A) est la modification interne de l’ARN la plus fréquente et la plus dynamiquement modulée. En effet, cette modification est réversible et résulte de l’activité de deux classes d’enzymes [38, 39] : les méthyltransférases, qui ajoutent cette modification aux ARN, et les déméthylases qui peuvent l’effacer. Ces enzymes jouent donc un rôle crucial dans la stabilité ou l’efficacité de la traduction des ARN cellulaires en déstabilisant localement leur structure ou en altérant leur interaction avec les RBP. Des travaux récents montrent que la modification m6A constitue une marque de différenciation entre les ARN cellulaires et exogènes [40]. En effet, plusieurs études ont mis en évidence la présence de modifications m6A dans une variété d’ARN viraux, modulant leur stabilité, leur traduction et, de facto, l’expression des gènes viraux [33, 41]. La cartographie des modifications m6A dans les génomes des flavivirus suggère que cette modification est une marque conservée au sein de cette famille virale [38]. Si, dans certains cas, la modification m6A régule négativement la production des particules virales [38], elle semble aussi jouer un rôle dans la discrimination du soi et du non-soi chez d’autres virus, en permettant aux ARN viraux de mimer les ARN cellulaires, ce qui leur permet d’échapper aux mécanismes de l’immunité innée [39, 42].
Les nouvelles frontières dans la découverte des protéines cellulaires de liaison aux ARN interagissant avec les ARN viraux
Ces dernières années, les progrès techniques ont permis des avancées majeures dans l’identification et la compréhension des interactions hôte-virus. Deux stratégies ont été développées pour étudier ces interactions en fonction de la cible choisie, protéine ou ARN. Les premières visent à isoler une protéine d’intérêt spécifique et à caractériser le répertoire des molécules d’ARN avec lesquelles elle interagit. Ces approches impliquent un pontage aux rayons ultraviolets (UV), entre la protéine et l’ARN, suivi de l’immunoprécipitation de la protéine de liaison aux ARN cibles et l’identification des ARN co-précipités par séquençage [43]. Ces méthodes ont souvent été utilisées dans un contexte d’infection virale pour caractériser les espèces d’ARN se liant aux RBP cellulaires [43–46] ou virales [47, 48]. Nous n’évoquerons pas ces techniques ici, mais nous suggérons aux lecteurs de se référer à d’excellentes revues sur le sujet [49–51]. Les techniques qui ciblent l’ARN sont plus récentes et consistent à purifier un ARN d’intérêt afin d’identifier de manière exhaustive toutes les protéines associées. Ces approches ont ainsi permis d’identifier un grand nombre de RBP non canoniques.
La technique de RNA-IC
La technique de RNA-IC (RNA interactome capture) a largement étendu le répertoire des RBP connues [52]. Cette technique consiste en un pontage des protéines aux ARN in situ en irradiant des cellules vivantes aux UV, suivi de la capture des ARN polyadénylés à l’aide d’une sonde d’oligo (dT)1, puis de l’identification des protéines associées aux ARN par spectrométrie de masse [52, 53]. Cette méthode a été appliquée à l’étude de l’interactome des ARN cellulaires pendant l’infection par le virus Sindbis (SINV) et a permis d’identifier plus de 200 RBP capables d’interagir différentiellement avec les ARN de l’hôte avant et pendant l’infection virale (Figure 2) [54]. Suite à la pandémie de la COVID-19, cette approche a été adaptée en utilisant des sondes spécifiques à la séquence virale pour la caractérisation de l’interactome de l’ARN viral du virus SARS-CoV-2 (vRIC) et a permis de mettre en évidence le rôle régulateur du complexe des ARNt ligases2 [55] (Tableau I). Cette étude a également montré que des médicaments ciblant certaines RBP interagissant avec les ARN viraux pouvaient inhiber l’infection in vitro. Bien que le RNA-IC permette d’identifier des RBP qui interagissent directement avec les ARN, cette méthode permet de purifier uniquement les ARN polyadénylés et ne fait pas la distinction entre ARN viraux et ARN cellulaires. Une adaptation du protocole expérimental vRIC a été mise en place récemment en utilisant un inhibiteur des ARN polymérases cellulaires pour mieux enrichir les ARN viraux et isoler les RBP associées par spectrométrie de masse de façon plus spécifique [55, 56].
 |
Figure 2 Description des techniques centrées sur l’ARN permettant de caractériser l’interactome des génomes viraux à ARN simple brin de polarité positive. Les différentes techniques de pêche des ARN viraux sont suivies par une analyse par spectrométrie de masse. Les diagrammes de Venn indiquent le nombre de protéines identifiées communes aux différentes techniques. Seules les protéines enrichies significativement (valeur p ajustée < 0,05) dans les conditions infectées par rapport aux conditions non infectées sont représentées (log2FC > 1). Pour les RNA-IC les données utilisées sont celles correspondant à une infection de 24 h. Pour les données de VIR-CLASP, le jeu de données utilisé est celui correspondant à l’infection de 3 heures sans traitement IFN. |
Résumé de différentes techniques de mise en évidence des protéines de liaison aux ARN associées aux génomes viraux centrées sur l’ARN et leurs caractéristiques.
La technique de ChIRP-MS
La méthode de ChIRP-MS (comprehensive Identification of RNA binding proteins coupled to mass spectrometry) consiste en un pontage au formaldéhyde entre les ARN et les protéines suivi de la purification de l’ARN grâce à des sondes marquées à la biotine et complémentaires à l’ARN d’intérêt. Contrairement au pontage par UV, l’utilisation de formaldéhyde conduit à la capture de tous les partenaires de l’ARN, y compris les protéines associées de façon indirecte. Les complexes ribonucléoprotéiques pontés sont purifiés avec des billes de streptavidine, puis analysés par spectrométrie de masse [57]. Développée à l’origine pour identifier les RBP associées au longs ARN non codants (long non coding RNA [lncARN]) [58], la méthode de ChIRP-MS a récemment été adaptée à l’identification des RBP associées aux ARN viraux des virus DENV et ZIKV [44], et a permis l’identification de 464 protéines humaines (Figure 2). Un grand nombre de ces protéines proviennent du réticulum endoplasmique (RE), où ces virus répliquent. Cette méthode a également permis d’identifier 146 protéines humaines associées à l’ARN du virus SARS-CoV-2, dont HNRP-NA2B1 (heterogeneous nuclear ribonucleoprotein A2/B1), ILF3 (interleukin enhancer-binding factor 3), QKI (quaking homolog, KH domain RNA binding), et SFPQ (splicing factor proline/glutamine-rich), qui ont toutes une activité provirale [59]. Il est important de noter que la méthode de ChIRP-MS ne permet pas de définir à quelles étapes du cycle viral ont lieu les interactions entre RBP et ARN viraux.
La méthode VIR-CLASP
La méthode de VIR-CLASP (viral cross-linking and solid-phase purification) repose sur l’infection de cellules hôtes avec des virus dont les génomes ARN ont incorporé des 4-thiouridines (4SU) à la place de l’uridine. L’irradiation aux UV des cellules infectées génère un pontage covalent uniquement entre les 4SU et les protéines cellulaires ou virales fixées sur ces motifs [60]. La capture en phase solide, et en conditions dénaturantes, de ces complexes avec des billes SPRI (solid-phase reversible immobilization)3 conduit à la purification des protéines pontées au génome viral étiqueté, qui seront ensuite identifiées par spectrométrie de masse. Grâce à cette approche, une étude a identifié les interactomes précoces de différents génomes viraux (CHIKV, IAV [Influenza a virus], ZIKV, VSV [virus de la stomatite vésiculaire]) (Figure 2).
Cette méthode a permis de montrer que le génome du virus chikungunya (CHIKV) pouvait interagir à la fois avec des facteurs pro- et antiviraux. En effet, alors que l’ARN viral du virus CHIKV est capable de détourner l’acide gras synthase (FASN, fatty acid synthase) à des fins provirales, la liaison avec la protéine YTHDF1 (YTH domain family protein 1) exerce un effet inhibiteur sur la réplication de ce virus (Tableau I) [60]. Si cette technique permet de mettre en évidence les protéines liant l’ARN viral entrant, elle ne permet cependant pas d’identifier les protéines se liant à l’ARN néosynthétisé.
La méthode RapID
Toutes les méthodes préalablement citées utilisent un pontage chimique ou aux UV permettant d’identifier les interactions, suivi d’une purification de l’ARN dans des conditions dénaturantes pour éliminer les interactions non spécifiques [61]. Une approche permettant d’identifier les interactions entre ARN et protéines dans des cellules vivantes, et sans pontage préalable, repose sur l’utilisation de cellules exprimant une biotine ligase d’origine bactérienne capable de biotinyler des protéines localisées à une distance de moins de 20 nm [62]. À l’origine développée pour identifier les interactions protéine-protéine, cette approche a été récemment adaptée à l’identification des interactions ARN-protéines. La méthode RapID (RNA-protein interaction detection) consiste à étiqueter l’ARN d’intérêt avec une séquence permettant le recrutement spécifique de la biotine ligase. Cette protéine s’associe donc à l’ARN étiqueté et marque, par ajout de biotine, les protéines présentes à proximité [61, 63]. Cette technique a permis d’identifier des RBP associées aux extrémités transcrites mais non traduites du génome du virus ZIKV (Figure 2) dans les tissus neuronaux et a aussi permis de mettre en évidence le rôle proviral du facteur QKI (KH domain rna binding protein) sur la réplication du virus ZIKV, puisque sa déplétion réduit le niveau d’ARN viral de 90 % dans les cellules infectées [63]. Il est important de noter que, cette technique peut être appliquée à l’identification des RBP associées à n’importe quelle séquence d’ARN viral, en se plaçant dans un système cellulaire exprimant la biotine ligase.
La méthode DRIMS
Une autre technique pour identifier les protéines associées aux ARN double brin pendant l’infection virale repose sur l’utilisation de l’anti-corps J2 capable de reconnaitre spécifiquement de longs ARN double brin. La méthode DRIMS (dsRNA immunoprecipitation followed by mass spectrometry) (Figure 2), développée dans notre laboratoire, a permis de mettre en évidence l’importance de l’interaction entre SFPQ (splicing factor proline/glutamine-rich) et l’ARN du virus SINV dans le cytoplasme des cellules infectées [64] (Tableau I). En effet, la déplétion de SFPQ ou la mutation de son domaine de liaison à l’ARN inhibent la réplication virale. Une autre protéine cellulaire provirale mise en évidence par la méthode DRIMS est l’hélicase DDX5 (DEAD (Asp-Glu-Ala-Asp) box helicase 5) [65] (Tableau I). En particulier, DDX5 et son partenaire DDX17 sont relocalisées du noyau vers le cytoplasme pendant l’infection et interagissent avec l’ARN viral et la protéine de capside virale. La déplétion de DDX5 et de DDX17 limite la réplication du virus SINV, ce qui suggère un rôle proviral de DDX5 et DDX17. Cependant, l’inconvénient majeur de cette méthode est que l’anticorps J2 peut reconnaître aussi bien les ARN double brin viraux que ceux d’origine cellulaire.
Conclusions et perspectives
Ces quelques exemples illustrent à quel point les interactions entre les ARN viraux et RBP sont fondamentales pour le cycle de vie des virus à ARN simple brin de polarité positive sur lesquels nous nous sommes focalisés et comment les progrès techniques permettent désormais de mieux identifier ces interactions complexes, ouvrant ainsi la voie à des perspectives de traitements plus ciblés contre les infections virales émergentes.
Ces études montrent que les RBP peuvent jouer un rôle pro- ou anti-viral, en fonction du virus considéré et du contexte cellulaire. Les mécanismes mis en jeu sont le résultat d’un processus de coévolution entre ces virus et leurs hôtes. Des résultats récents mettent également en évidence que de nombreuses protéines associées aux ARN viraux n’ont pas de domaines canoniques de liaison à l’ARN et sont souvent des enzymes impliquées dans le métabolisme. Ces RBP non canoniques semblent pouvoir réguler l’ARN tout en ayant leur fonction enzymatique modulée par cette interaction [52]. Ce phénomène, encore très peu exploré, mérite d’être étudié plus en profondeur, notamment dans le contexte des infections virales.
Enfin, pour comprendre pleinement les interactions entre les ARN viraux et les RBP, il est crucial d’aller au-delà de simples caractéristiques physico-chimiques des molécules d’ARN. L’abondance, la localisation intracellulaire et les interactions moléculaires jouent également un rôle clé dans la régulation des réponses antivirales. Des structures intracellulaires dynamiques peuvent influencer la réponse antivirale en modifiant l’accessibilité et la détection des ARN viraux. Les ARN peuvent en effet être localisés dans différents compartiments cellulaires ou être associés à des structures membranaires pour échapper à leur détection, ce qui affecte également leur interaction avec les protéines cellulaires. C’est le cas des granules de stress, qui sont des structures dynamiques dans lesquelles se concentrent ARN et RBP par un processus de séparation de phase liquide-liquide, et qui sont des structures clés dans la réponse cellulaire aux virus [66].
Afin d’étudier ces aspects de manière plus détaillée, de nouvelles méthodes permettent de cartographier les modifications épitranscriptomiques des ARN [67] (→) au cours de l’infection et les interactions des ARN viraux avec des protéines à une échelle plus fine, tandis que les techniques d’imagerie avancées offrent des aperçus sur la dynamique des interactions ARN viraux-protéines cellulaires au sein des cellules infectées. Les recherches futures dans ce domaine devront continuer à explorer ces mécanismes avec des techniques de pointe afin de développer des stratégies thérapeutiques ciblées, plus efficaces, et tenant compte de la complexité et de la dynamique des interactions moléculaires.
(→) Voir m/s n° 3, 2024, page 287
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Ahlquist P, Noueiry AO, Lee W-M, et al. Host factors in positive-strand RNA virus genome replication. J Virol 2003 ; 77 : 8181–6. [CrossRef] [PubMed] [Google Scholar]
- Bhatt S, Gething PW, Brady OJ, et al. The global distribution and burden of dengue. Nature 2013 ; 496 : 504–7. [CrossRef] [PubMed] [Google Scholar]
- Lazear HM, Diamond MS. Zika virus: new clinical syndromes and its emergence in the western hemisphere. J Virol 2016 ; 90 : 4864–75. [CrossRef] [PubMed] [Google Scholar]
- Poon LLM, Peiris M. Emergence of a novel human coronavirus threatening human health. Nat Med 2020 ; 26 : 317–9. [CrossRef] [PubMed] [Google Scholar]
- Li Z, Nagy PD. Diverse roles of host RNA binding proteins in RNA virus replication. RNA Biology 2011 ; 8 : 305–15. [CrossRef] [PubMed] [Google Scholar]
- Girardi E, Pfeffer S, Baumert TF, et al. Roadblocks and fast tracks: how RNA binding proteins affect the viral RNA journey in the cell. Seminars in Cell & Developmental Biology 2021 ; 111 : 86–100. [CrossRef] [PubMed] [Google Scholar]
- Schlee M, Hartmann G. Discriminating self from non-self in nucleic acid sensing. Nat Rev Immunol 2016 ; 16 : 566–80. [CrossRef] [PubMed] [Google Scholar]
- Maarifi G, Smith N, Nisole S. La réponse interféron. Un grand pouvoir implique de grandes responsabilités. Med Sci (Paris) 2020 ; 36 : 206–9. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Iwasaki A, Medzhitov R. Toll-like receptor control of the adaptive immune responses. Nat Immunol 2004 ; 5 : 987–95. [CrossRef] [PubMed] [Google Scholar]
- Hardarson HS, Baker JS, Yang Z, et al. Toll-like receptor 3 is an essential component of the innate stress response in virus-induced cardiac injury. American Journal of Physiology-Heart and Circulatory Physiology 2007 ; 292 : H251–8. [CrossRef] [Google Scholar]
- Perales-Linares R, Navas-Martin S. Toll-like receptor 3 in viral pathogenesis: friend or foe? Immunology 2013 ; 140 : 153–67. [CrossRef] [PubMed] [Google Scholar]
- Tsai Y-T, Chang S-Y, Lee C-N, et al. Human TLR3 recognizes dengue virus and modulates viral replication in vitro. Cell Microbiol 2009 ; 11 : 604–15. [CrossRef] [Google Scholar]
- Dang J, Tiwari SK, Lichinchi G, et al. Zika virus depletes neural progenitors in human cerebral organoids through activation of the innate immune receptor TLR3. Cell Stem Cell 2016 ; 19 : 258–65. [CrossRef] [Google Scholar]
- Jensen S, Thomsen AR. Sensing of RNA viruses: a review of innate immune receptors involved in recognizing RNA virus invasion. J Virol 2012 ; 86 : 2900–10. [CrossRef] [PubMed] [Google Scholar]
- Kowalinski E, Louber J, Gerlier D, et al. RIG-I : Un commutateur moléculaire détecteur d’ARN viral. Med Sci (Paris) 2012 ; 28 : 136–8. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Habjan M, Andersson I, Klingström J, et al. Processing of genome 5′ termini as a strategy of negative-strand RNA viruses to avoid RIG-I-dependent interferon induction. PLoS ONE 2008 ; 3 : e2032. [CrossRef] [PubMed] [Google Scholar]
- Kato H, Takeuchi O, Sato S, et al. Differential roles of MDA5 and RIG-I helicases in the recognition of RNA viruses. Nature 2006 ; 441 : 101–5. [CrossRef] [PubMed] [Google Scholar]
- Kato H, Sato S, Yoneyama M, et al. Cell type-specific involvement of RIG-I in antiviral response. Immunity 2005 ; 23 : 19–28. [CrossRef] [PubMed] [Google Scholar]
- Chazal M, Beauclair G, Gracias S, et al. RIG-I recognizes the 5′ region of Dengue and Zika virus genomes. Cell Reports 2018 ; 24 : 320–8. [CrossRef] [Google Scholar]
- Dias Junior AG, Sampaio NG, Rehwinkel J. A Balancing Act: MDA5 in antiviral immunity and autoinflammation. Trends Microbiol 2019 ; 27 : 75–85. [CrossRef] [PubMed] [Google Scholar]
- Berke IC, Yu X, Modis Y, et al. MDA5 assembles into a polar helical filament on dsRNA. Proc. Natl. Acad. Sci. U.S.A. 2012 ; 109 : 18437–41. [CrossRef] [PubMed] [Google Scholar]
- Muñoz-Jordán JL, Fredericksen BL. How flaviviruses activate and suppress the interferon response. Viruses 2010 ; 2 : 676–91. [CrossRef] [PubMed] [Google Scholar]
- Zalinger ZB, Elliott R, Rose KM, et al. MDA5 is critical to host defense during Infection with murine coronavirus. J Virol 2015 ; 89 : 12330–40. [CrossRef] [PubMed] [Google Scholar]
- Rothenfusser S, Goutagny N, DiPerna G, et al. The RNA helicase Lgp2 inhibits TLR-independent sensing of viral replication by Retinoic Acid-Inducible Gene-I. J Immunol 2005 ; 175 : 5260–8. [CrossRef] [PubMed] [Google Scholar]
- Venkataraman T, Valdes M, Elsby R, et al. Loss of DExD/H Box RNA helicase LGP2 manifests disparate antiviral responses. J Immunol 2007 ; 178 : 6444–55. [CrossRef] [PubMed] [Google Scholar]
- Sadler AJ, Williams BRG. Interferon-inducible antiviral effectors. Nat Rev Immunol 2008 ; 8 : 559–68. [CrossRef] [PubMed] [Google Scholar]
- Stern-Ginossar N, Thompson SR, Mathews MB, et al. Translational control in virus-infected cells. Cold Spring Harb Perspect Biol 2019 ; 11 : a033001. [CrossRef] [Google Scholar]
- Mudhasani R, Tran JP, Retterer C, et al. Protein kinase R degradation is essential for Rift Valley Fever Virus infection and is regulated by SKP1-CUL1-F-box (SCF)FBXW11-NSs E3 ligase. PLoS Pathog 2016 ; 12 : e1005437. [CrossRef] [PubMed] [Google Scholar]
- Léger P, Lozach P-Y. Le virus de la fièvre de la vallée du Rift et son étonnante protéine NSs. Med Sci (Paris) 2021 ; 37 : 601–8. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Silverman RH. Viral Encounters with 2′, 5′-Oligoadenylate Synthetase and RNase L during the Interferon Antiviral Response. J Virol 2007 ; 81 : 12720–9. [CrossRef] [PubMed] [Google Scholar]
- Bisbal C, Salehzada T. La RNase L, un acteur essentiel de la réponse cellulaire antivirale. Med Sci (Paris) 2008 ; 24 : 859–64. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Zhang R, Jha BK, Ogden KM, et al. Homologous 2′, 5′-phosphodiesterases from disparate RNA viruses antagonize antiviral innate immunity. Proc. Natl. Acad. Sci. U.S.A. 2013 ; 110 : 13114–9. [CrossRef] [PubMed] [Google Scholar]
- Gonzales-van Horn SR, Sarnow P. Making the Mark: The role of adenosine modifications in the life cycle of RNA viruses. Cell Host & Microbe 2017 ; 21 : 661–9. [CrossRef] [Google Scholar]
- Frye M, Jaffrey SR, Pan T, et al. RNA modifications: what have we learned and where are we headed? Nat Rev Genet 2016 ; 17 : 365–72. [CrossRef] [PubMed] [Google Scholar]
- Samuel CE. Adenosine deaminases acting on RNA (ADARs) are both antiviral and proviral. Virology 2011 ; 411 : 180–93. [CrossRef] [Google Scholar]
- Lamers MM, Hoogen BG van den, Haagmans BL. ADAR1: “Editor-in-Chief” of cytoplasmic innate immunity. Front. Immunol. 2019 ; 10 : 1763. [CrossRef] [Google Scholar]
- Taylor JM. Hepatitis D Virus Replication. Cold Spring Harb Perspect Med 2015 ; 5 : a021568. [CrossRef] [Google Scholar]
- Gokhale NS, McIntyre ABR, McFadden MJ, et al. N6-methyladenosine in flaviviridae viral RNA genomes regulates infection. Cell Host & Microbe 2016 ; 20 : 654–65. [CrossRef] [Google Scholar]
- Lu M, Zhang Z, Xue M, et al. N6-methyladenosine modification enables viral RNA to escape recognition by RNA sensor RIG-I. Nat Microbiol 2020 ; 5 : 584–98. [CrossRef] [PubMed] [Google Scholar]
- Shulman Z, Stern-Ginossar N. The RNA modification N6-methyladenosine as a novel regulator of the immune system. Nat Immunol 2020 ; 21 : 501–12. [CrossRef] [PubMed] [Google Scholar]
- Dang W, Xie Y, Cao P, et al. N6-methyladenosine and viral infection. Front. Microbiol. 2019 ; 10 : 417. [CrossRef] [Google Scholar]
- Thiel V. Viral RNA in an m6A disguise. Nat Microbiol 2020 ; 5 : 531–2. [CrossRef] [PubMed] [Google Scholar]
- Ule J, Jensen KB, Ruggiu M, et al. CLIP identifies Nova-regulated RNA networks in the brain. Science 2003 ; 302 : 1212–5. [CrossRef] [PubMed] [Google Scholar]
- Ooi YS, Majzoub K, Flynn RA, et al. An RNA-centric dissection of host complexes controlling flavivirus infection. Nat Microbiol 2019 ; 4 : 2369–82. [CrossRef] [PubMed] [Google Scholar]
- Scheel TKH, Luna JM, Liniger M, et al. A broad RNA virus survey reveals both miRNA dependence and functional sequestration. Cell Host & Microbe 2016 ; 19 : 409–23. [CrossRef] [Google Scholar]
- Flynn RA, Martin L, Spitale RC, et al. Dissecting noncoding and pathogen RNA–protein interactomes. RNA 2015 ; 21 : 135–43. [CrossRef] [PubMed] [Google Scholar]
- Williams GD, Townsend D, Wylie KM, et al. Nucleotide resolution mapping of influenza A virus nucleoprotein-RNA interactions reveals RNA features required for replication. Nat Commun 2018 ; 9 : 465. [CrossRef] [PubMed] [Google Scholar]
- Sokoloski KJ, Nease LM, May NA, et al. Identification of interactions between Sindbis Virus capsid protein and cytoplasmic vRNA as novel virulence determinants. PLoS Pathog 2017 ; 13 : e1006473. [CrossRef] [PubMed] [Google Scholar]
- Bieniasz PD, Kutluay SB. CLIP-related methodologies and their application to retrovirology. Retrovirology 2018 ; 15 : 35. [CrossRef] [PubMed] [Google Scholar]
- Haddad C, Davila-Calderon J, Tolbert BS. Integrated approaches to reveal mechanisms by which RNA viruses reprogram the cellular environment. Methods 2020 ; 183 : 50–6. [CrossRef] [PubMed] [Google Scholar]
- Haecker I, Renne R. HITS-CLIP and PAR-CLIP advance viral miRNA targetome analysis. Crit Rev Eukaryot Gene Expr 2014 ; 24 : 101–16. [CrossRef] [Google Scholar]
- Hentze MW, Castello A, Schwarzl T, et al. A brave new world of RNA-binding proteins. Nat Rev Mol Cell Biol 2018 ; 19 : 327–41. [CrossRef] [PubMed] [Google Scholar]
- Castello A, Horos R, Strein C, et al. System-wide identification of RNA-binding proteins by interactome capture. Nat Protoc 2013 ; 8 : 491–500. [CrossRef] [PubMed] [Google Scholar]
- Garcia-Moreno M, Noerenberg M, Ni S, et al. System-wide profiling of RNA-binding proteins uncovers key regulators of virus infection. Molecular Cell 2019 ; 74 : 196–211.e11. [CrossRef] [PubMed] [Google Scholar]
- Kamel W, Noerenberg M, Cerikan B, et al. Global analysis of protein-RNA interactions in SARS-CoV-2-infected cells reveals key regulators of infection. Mol Cell 2021 ; 81 : 2851–2867.e7. [CrossRef] [PubMed] [Google Scholar]
- Kamel W, Ruscica V, Embarc-Buh A, et al. Alphavirus infection triggers selective cytoplasmic translocation of nuclear RBPs with moonlighting antiviral roles. Mol Cell 2024 ; 84 : 4896–911.e7. [CrossRef] [PubMed] [Google Scholar]
- Chu C, Chang HY. ChIRP-MS: RNA-directed proteomic discovery. In : Sado T, editor. X-Chromosome Inactivation. Methods in Molecular Biology. New York, NY : Springer New York, 2018 : 37–45. [CrossRef] [PubMed] [Google Scholar]
- Chu C, Zhang QC, da Rocha ST, et al. Systematic discovery of Xist RNA binding proteins. Cell 2015 ; 161 : 404–16. [CrossRef] [Google Scholar]
- Labeau A, Fery-Simonian L, Lefevre-Utile A, et al. Characterization and functional interrogation of the SARS-CoV-2 RNA interactome. Cell Reports 2022 ; 39 : 110744. [CrossRef] [Google Scholar]
- Kim B, Arcos S, Rothamel K, et al. Discovery of widespread host protein interactions with the pre-replicated genome of CHIKV using VIR-CLASP. Mol Cell 2020 ; 78 : 624–40.e7. [CrossRef] [PubMed] [Google Scholar]
- Ramanathan M, Porter DF, Khavari PA. Methods to study RNA–protein interactions. Nat Methods 2019 ; 16 : 225–34. [CrossRef] [PubMed] [Google Scholar]
- Roux KJ, Kim DI, Raida M, et al. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol 2012 ; 196 : 801–10. [CrossRef] [PubMed] [Google Scholar]
- Ramanathan M, Majzoub K, Rao DS, et al. RNA–protein interaction detection in living cells. Nat Methods 2018 ; 15 : 207–12. [CrossRef] [PubMed] [Google Scholar]
- Girardi E, Messmer M, Lopez P, et al. Proteomics-based determination of double-stranded RNA interactome reveals known and new factors involved in Sindbis virus infection. RNA 2023 ; 29 : 361–75. [CrossRef] [PubMed] [Google Scholar]
- Messmer M, Pierson L, Pasquier C, et al. DEAD box RNA helicase 5 is a new pro-viral host factor for Sindbis virus infection. Virol J 2024 ; 21 : 76. [CrossRef] [PubMed] [Google Scholar]
- Li H, Ernst C, Kolonko-Adamska M, et al. Phase separation in viral infections. Trends in Microbiol 2022 ; 30 : 1217–31. [CrossRef] [Google Scholar]
- Salvetti A. Épitranscriptome. Med Sci (Paris) 2024 ; 40 : 287–7. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des tableaux
Résumé de différentes techniques de mise en évidence des protéines de liaison aux ARN associées aux génomes viraux centrées sur l’ARN et leurs caractéristiques.
Liste des figures
 |
Figure 1 Reconnaissance des ARN exogènes. Les virus à ARN sont détectés par la cellule grâce à des protéines cellulaires de liaison aux ARN (RBPc) ayant un rôle de senseurs. TLR3/7/8 reconnaissent les ARN dans les endosomes. RIG-I et MDA5 détectent les ARN double brin (ARNdb) cytoplasmiques. Cette détection permet l’activation des réponses IFN et NF-kB. Les ARN double brin sont aussi détectés par des protéines cellulaires de liaison aux ARN effectrices telles que PKR et les OAS ce qui inhibe la traduction et conduit à une dégradation globale des ARN. Afin d’éviter un emballement de ces réponses, ADAR1 peut déstabiliser la structure des ARN double brin et éviter leur reconnaissance par les protéines cellulaires de liaison aux ARN senseurs et effectrices. |
| Dans le texte | |
 |
Figure 2 Description des techniques centrées sur l’ARN permettant de caractériser l’interactome des génomes viraux à ARN simple brin de polarité positive. Les différentes techniques de pêche des ARN viraux sont suivies par une analyse par spectrométrie de masse. Les diagrammes de Venn indiquent le nombre de protéines identifiées communes aux différentes techniques. Seules les protéines enrichies significativement (valeur p ajustée < 0,05) dans les conditions infectées par rapport aux conditions non infectées sont représentées (log2FC > 1). Pour les RNA-IC les données utilisées sont celles correspondant à une infection de 24 h. Pour les données de VIR-CLASP, le jeu de données utilisé est celui correspondant à l’infection de 3 heures sans traitement IFN. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




