| Issue |
Med Sci (Paris)
Volume 40, Number 12, Décembre 2024
Épigénétique : développement et destin cellulaire
|
|
|---|---|---|
| Page(s) | 904 - 913 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2024176 | |
| Published online | 20 December 2024 | |
Hétérochromatine et contrôle épigénétique des séquences répétées
Heterochromatin and epigenetic control of repeat sequences
Institut de génétique humaine, CNRS, Université de Montpellier, UMR 9002, Montpellier, France
*
constance.kleijwegt@igh.cnrs.fr
**
jerome.dejardin@igh.cnrs.fr
L’hétérochromatine est une structure décrite comme restrictive et répressive. On la retrouve notamment au niveau des séquences répétées qui représentent près de la moitié du génome humain. Ces séquences, dont l’origine reste incertaine, peuvent jouer un rôle structural, protecteur ou régulateur. Cependant, leur homologie de séquence ou leur capacité à transposer pour certaines, peuvent compromettre la stabilité du génome, et la formation d’hétérochromatine au niveau de ces régions permet de les réguler. Souvent imaginée comme une structure dont la composition est stable, l’hétérochromatine est en réalité bien plus hétérogène, en fonction du locus et du type cellulaire où elle est établie.
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
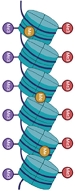
Vignette (© Biorender.com).
L’hétérochromatine est une structure nucléoprotéique fermée localisée sur des endroits spécifiques du chromosome. Cette structure limite l’accès à l’ADN pour les machineries de la transcription, de la réplication ou de la réparation, permettant un contrôle de tous ces processus. On distingue historiquement deux catégories d’hétérochromatine : l’hétérochromatine facultative, transitoire, permettant essentiellement la régulation de l’expression des gènes, et l’hétérochromatine constitutive, condensée tout au long du cycle cellulaire et généralement retrouvée dans la plupart des cellules. Cette dernière est classiquement caractérisée par des marques épigénétiques inhibitrices, telles que les tri-méthylations des lysines 9 de l’histone H3 (H3K9me3) et 20 de l’histone H4 (H4K20me3), une hypoacétylation générale des histones, et la méthylation de l’ADN (mCpG) (Figure 1).
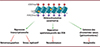 |
Figure 1 Les différents rôles de l’hétérochromatine constitutive. L’hétérochromatine est une structure permettant d’assurer la stabilité génomique. Elle est classiquement caractérisée par les marques d’histones H3K9me3 et H4K20me3 et par la méthylation de l’ADN. DSB : double strand break (cassure double brin) (adaptée de [14]) (Figure créée avec Biorender.com - agrément UG270PLMD2). |
L’hétérochromatine constitutive se forme essentiellement sur les séquences répétées du génome. Longtemps qualifiées d’ADN « poubelle », les séquences répétées représentent environ 50 % du génome humain. Elles sont généralement classées dans deux catégories principales : les répétitions en tandem et les répétitions dispersées (Tableau I). Une répétition en tandem est constituée d’une unité de séquence plus ou moins longue (de deux paires de bases à plusieurs centaines de kilopaires de bases) itérée successivement sur un locus génomique et pouvant s’étendre sur plusieurs mégabases (pour revue, voir [1]). Les répétitions dispersées sont, quant à elles, disséminées dans le génome. Elles sont principalement représentées par des séquences dérivées d’éléments transposables qui, par divers mécanismes de transposition peuvent se déplacer/se multiplier sur le génome (pour revue, voir [2]). Il existe différents types d’éléments transposables qui diffèrent par leur mode de transposition : les rétrotransposons contenant des séquences LTR (long terminal repeats) comme les ERV (endogenous retroviruses) de classes I à III ; les rétrotransposons non-LTR, tels que les LINE et les SINE (long et short interspersed nuclear elements) ; les transposons à ADN, comme les transposons de type hAT ou Tc-1. Seuls les rétrotransposons « jeunes » évolutivement sont encore capables de se mobiliser (la grande majorité ayant perdu cette capacité par dérive génétique) [3,4]. Ceci concerne quelques copies d’éléments LINE chez l’homme, étant euxmêmes capables de mobiliser en trans les éléments SVA1 et SINE. Malgré quelques avantages acquis pour certains processus physiologiques (e.g. la co-option de certaines protéines d’enveloppe codées par des ERV pour le fonctionnement du placenta [5]), les séquences répétées représentent une menace pour la stabilité du génome, du fait de leur potentiel de mobilisation dans le cas des éléments transposables, de leur homologie de séquence qui peut donner lieu à des recombinaisons illégitimes, ou de leur influence sur les gènes voisins par effets de position. En général, ces séquences sont donc réprimées et de l’hétérochromatine se forme sur ces régions.
Cette revue a pour but d’établir l’état de l’art des connaissances actuelles sur l’origine des séquences répétées et sur les mécanismes mis en place par la cellule pour les contrôler. Nous verrons notamment que l’hétérochromatine semble finalement bien plus dynamique qu’elle n’est classiquement décrite et que sa composition varie en fonction du locus génomique et du type cellulaire dans lesquels elle est établie.
Origine des séquences répétées
Il n’est pas toujours évident d’établir l’origine des séquences répétées, mais les progrès techniques en matière de séquençage du génome (notamment le séquençage de longs fragments d’ADN) et d’analyses bioinformatiques permettent de retracer leur évolution au sein de l’arbre du vivant et d’émettre des hypothèses sur leur apparition. Parmi les éléments transposables, l’origine des ERV est la plus évidente à retracer. En effet, leurs séquences suggèrent qu’ils sont les reliques d’anciennes infections rétrovirales de cellules germinales (évènements d’endogénisation). Leur insertion génomique a été par la suite maintenue à travers l’évolution par transmission verticale [6]. Les SINE semblent, quant à eux, émaner de la transcription inverse d’ARN de transfert (ARNt), ribosomiques (ARN 5SL) et d’ARN des particules de reconnaissance du signal (ARN 7SL). Cette transcription inverse implique la transcriptase inverse codée par les LINE [7]. L’origine des transposons ADN et des LINE est plus difficile à définir. Leur apparition pourrait être la résultante de transferts horizontaux (i.e. transmission d’ADN entre deux organismes non nécessairement apparentés, indépendamment de la reproduction) [8,9]. Par exemple, les rétrotransposons de type LINE-1, représentant près de 17 % du génome humain, auraient été introduits horizontalement chez l’ancêtre commun des mammifères thériens2 [9]. L’apparition et l’amplification des éléments transposables pourraient être à l’origine d’un grand nombre de répétitions en tandem [10,11]. C’est le cas pour au moins 23 % des minisatellites/satellites chez l’homme, dérivant essentiellement des rétrotransposons [10]. Plusieurs mécanismes ont été proposés pour expliquer ce lien. Lors de la réplication, un mauvais appariement des brins entre deux transposons (slipped-strand mispairing ou SSM) peut induire un « glissement » de la fourche réplicative et une duplication [10]. Un remaniement asymétrique lors de la méiose entre deux éléments transposables a le potentiel, entre autres, de créer des duplications [10,11]. Enfin, la transposition peut induire des cassures double brin et leur réparation par recombinaison homologue intrachromosomale (iHR) ou non-allélique (NAHR) peut aussi conduire à des duplications [11].
Plusieurs mécanismes peuvent être à l’origine de l’expansion ou la contraction des répétitions en tandem. La nature répétitive de la séquence peut tout d’abord induire un « glissement » de l’ADN polymérase lors de la réplication de ces régions. Les répétitions en tandem sont aussi plus enclines à former des structures secondaires de l’ADN, perturbant la progression de la polymérase et pouvant résulter en des variations de longueur. De même, comme évoqué précédemment, des remaniements lors de la méiose ou lors de la réparation par iHR ou NAHR peuvent participer à l’expansion ou la contraction [12]. Enfin, plus récemment, il a été suggéré que l’expansion et la contraction des centromères serait la résultante du processus de réplication induite par cassure (BIR), similaire au processus d’élongation des télomères dans les cellules ALT (alternative lengthening of telomeres) [13].
Rôles de l’hétérochromatine au niveau des séquences répétées
L’hétérochromatine constitutive, qui se forme généralement sur les répétitions, est une structure nécessaire à la stabilité du génome (Figure 1) [14]. Elle permet, par exemple, d’inhiber la transcription des rétrotransposons, dont la rétrotranscription en ADN complémentaire peut conduire à leur insertion, ailleurs dans le génome, et donc leur multiplication. En fonction de leur site d’insertion, ceux-ci ont le potentiel d’induire des mutations dans certains gènes [15] ou de conduire à une perturbation de leur expression [16]. La transcription des séquences d’ADN satellite peut, quant à elle, provoquer la génération de structures hybrides ADN-ARN, appelée boucles ARN ou R-loop [17]. Les structures R-loop, si non résolues, peuvent entraîner l’arrêt de la fourche de réplication et induire des dommages liés au stress réplicatif [18].
L’organisation de l’ADN en hétérochromatine rendrait celui-ci moins susceptible aux dommages. L’irradiation de cellules HeLa montre en effet une fréquence de cassures double brin (DSB) 5 à 50 fois plus basse lorsque la chromatine est condensée [19]. Plusieurs études pangénomiques ont, par la suite, montré une plus grande susceptibilité de l’euchromatine aux DSB endogènes (pour revue, voir [20]). Pour autant, le taux de mutation des régions enrichies pour H3K9me3 est plus important que celui des régions d’euchromatine dans différents types de cellules cancéreuses humaines [21]. Un défaut dans les processus de réparation des dommages pourrait expliquer cette observation. En effet, les processus de réparation de mésappariements de l’ADN (MMR) et d’excision de nucléotides (NER) semblent affectés par l’enrichissement en H3K9me3, la marque principale de l’hétérochromatine constitutive [22,23]. Ainsi, bien que l’hétérochromatine soit en général moins sujette aux dommages à l’ADN, des défauts dans la réparation de ces derniers pourraient, entre autres, être la cause d’un taux de mutation plus important. L’hétérochromatine peut également prévenir les phénomènes de recombinaison homologue (HR) entre deux répétitions situées sur des chromosomes non homologues, causant des translocations et des structures chromosomiques anormales [14]. Pour prévenir ces processus aberrants, des mécanismes de régulation spatiotemporelle ont été mis en évidence chez la drosophile et la souris [24]. Les cassures réparées lors de la HR sont relocalisées en périphérie du domaine d’hétérochromatine, et loin des séquences similaires avec lesquelles elles pourraient se mésapparier. Il est cependant important de noter que dans le cas des télomères, l’hétérochromatine semble à l’inverse promouvoir l’élongation alternative des télomères (ALT), processus fondé sur la réplication induite par une cassure (BIR), une forme particulière de recombinaison homologue [25] (voir plus loin).
Enfin, l’hétérochromatine joue également un rôle structural au niveau des régions péricentromériques où elle permet un assemblage correct des centromères voisins et la cohésion des chromatides sœurs au moment de la mitose [14,26]. En effet, la perte d’H3K9me3 et des homologues de son reader HP1 (voir plus loin) induit des défauts de ségrégation des chromosomes (aneuploïdie) au cours de la mitose.
Mécanismes moléculaires impliqués dans la formation et le maintien de l’hétérochromatine
Pour établir l’hétérochromatine au niveau des séquences répétées, plusieurs mécanismes moléculaires sont mis en place par la cellule. L’étude de différents locus génomiques dans différents types cellulaires a permis de montrer qu’il n’existe pas un type d’hétérochromatine universel et que ces régions sont bien plus dynamiques qu’initialement décrites. Dans cette partie de la revue, nous nous intéresserons aux principaux mécanismes décrits pour la régulation des régions répétées les plus étudiées : les péricentromères, les télomères et les rétrotransposons. D’autres types de répétitions peuvent être hétérochromatinisées, en particulier si leur itération devient importante (e.g. certains microsatellites, ou l’ADN ribosomique [rDNA]), mais les mécanismes, encore peu compris, ne seront pas décrits dans cette revue.
L’hétérochromatine structurale des péricentromères
Dans les cellules somatiques eucaryotes et les cellules souches embryonnaires murines (mES), l’hétérochromatine péricentromérique est généralement constituée d’un type de répétitions en tandem appelées séquences satellites (Tableau I) dont la séquence diffère entre les espèces. Ces répétitions sont caractérisées, entre autres, par un enrichissement des marques H3K9me3, H4K20me3 de leurs histones associées, et par la méthylation des cytosines au niveau des dinucléotides CpG (CpGme) (Figure 2A) [27,28]. La méthylation de l’ADN est classiquement réalisée par trois ADN méthyltransférases, DNMT (DNA methyltransferase) 1 pour la maintenance, et DNMT3A et DNMT3B pour la méthylation de novo. Comment la méthylation de l’ADN participe à la compaction de la chromatine reste encore mal compris. Il a été montré qu’elle favoriserait in vitro la stabilité des nucléosomes [29]. De plus, plusieurs protéines pouvant participer à la répression de la transcription peuvent reconnaître les CpG méthylés, en recrutant par exemple des histones déacétylases [30].
 |
Figure 2 L’hétérochromatine des péricentromères. A. L’hétérochromatine péricentromérique des cellules souches embryonnaires [ES] et différenciées est caractérisée par les marques classiques de l’hétérochromatine, H3K9me3, H4K20me3 et CpGme, apposées respectivement par les enzymes SUV39H, SUV420H et DNMTs. H3K9me3 permet le recrutement de HP1 et ATRX. ATRX permet le recrutement de DAXX, chaperon de l’histone H3.3. B. L’hétérochromatine péricentromérique sur le génome paternel des embryons précoces murins est caractérisée par les marques H3K27me3 et H2AK119Ub, apposées respectivement par PRC2 et PRC1. Enfin, la protéine BEND3 permet le recrutement du complexe déacétylase NuRD (figure créée avec Biorender.com - agrément ED279Q865G). |
L’initiation de la formation d’hétérochromatine au niveau des péricentromères est restée longtemps une question en suspens, du fait de la non-conservation de la séquence entre espèces. Cependant, très récemment, il a été démontré dans des cellules mES que la protéine ZNF512 (zinc-finger protein 512) et son paralogue ZNF512b reconnaissent une itération espacée du trinucléotide TTC. De façon intéressante, cette itération est présente sur les péricentromères de toutes les espèces mammifères. Il semble que ZNF512 soit impliquée dans le recrutement initial des histones méthyltransférases SUV39H1/2 (ou SUV39H, pour suppressor of variegation 3-9 homologs) permettant d’apposer la marque H3K9me3 [31]. Par la suite, le maintien de cette marque s’effectuerait par la présence d’ARN non codants, transcrits depuis cette région et recrutant SUV39H [31,32]. H3K9me3 peut être reconnue par diverses protéines à domaines spécialisés, dont les protéines à chromodomaine HP1 ( heterochromatin protein 1) [27,28]. Ces dernières participent à la compaction de la chromatine et permettent par la suite le recrutement des enzymes SUV420H1/2 (ou SUV420H, pour suppressor of variegation 4-20 homologs), en charge de l’établissement de la marque d’hétérochromatine H4K20me3 dont le rôle reste encore incertain [28]. Récemment, une liaison entre l’ADN méthyltransférase DNMT1 et H4K20me3 a été démontrée ; elle permettrait le maintien de l’hétérochromatine sur les rétrotransposons LINE-1 [33]. Il est donc raisonnable de penser que ce mécanisme puisse être également présent au niveau d’autres locus enrichis pour la marque H4K20me3, dont les péricentromères. Parallèlement à son rôle dans la triméthylation de H4K20, SUV420H2 se lie à la cohésine aux péricentromères et participe à la cohésion des chromatides sœurs [34].
Enfin, l’hétérochromatine des péricentromères est également caractérisée par la présence du variant d’histone H3.3, incorporé par le chaperon d’histone DAXX (death domain-associated protein 6) indépendamment de la réplication [35]. DAXX forme un dimère avec la protéine ATRX (alpha thalassemia/mental retardation syndrome x-linked) qui peut reconnaître la marque H3K9me3 [28]. Le rôle d’H3.3, initialement décrit dans des régions transcriptionnellement actives, est encore incertain, en particulier dans ces régions. H3.3 pourrait notamment être important pour la transcription des ARN non codants, essentiels pour la formation de l’hétérochromatine aux péricentromères [36].
Ce mécanisme d’hétérochromatinisation des péricentromères est différent sur le génome paternel dans les embryons précoces de souris (Figure 2B). Alors qu’on observe également la présence du variant H3.3, les marques classiques H3K9me3, H4K20me3 et CpGme ne sont pas présentes. Ces régions, dans ce cas particulier, sont caractérisées par les marques H3K27me3 et H2AK119Ub, apposées respectivement par les complexes protéiques polycomb PRC (polycomb repressive complex) 2 et PRC1 [27,28]. L’absence de H3K9me3 et de CpGme permet les recrutements consécutifs de la protéine BEND3 (BEN domain-containing protein 3) et du complexe déacétylase d’histones NuRD (nucleosome remodeling and deacetylase) [27,28].
L’hétérochromatine au niveau des télomères : une idée reçue ?
Pourquoi l’extrémité des chromosomes n’est pas reconnue comme une cassure double brin devant être réparée est une question sur laquelle de nombreux chercheurs se penchent. Les séquences télomériques, aux extrémités de chaque chromosome, doivent être protégées des processus de réparation afin d’éviter des évènements de fusion entre deux chromosomes. Le complexe shelterin (composé de telomeric repeat-binding factor 1 et 2 [TRF1,2], repressor and activator protein 1 [RAP1], TRF1-interacting nuclear protein 2 [TIN2], protection of telomeres 1 [POT1], et POT1- and TIN2-interacting protein [TPP1]) participe à cette protection en formant une structure secondaire d’ADN, la t-loop [28]. Classiquement, les télomères sont décrits comme hétérochromatinisés, postulat renforcé par la répression de gènes rapporteurs à proximité des télomères, un phénomène connu sous le nom d’effet de position télomérique (TPE pour telomeric position effect), chez la mouche, la levure et dans des cellules cancéreuses humaines [37] (→).
(→) Voir la Synthèse de A. Decottignies, m/s n° 2, février 2014, page 173
Cependant, de nombreuses études récentes viennent nuancer ce dogme. En effet, les télomères des cellules humaines (cellules ES, cellules différenciées et cellules cancéreuses non-ALT) portent plutôt des marques associées à l’euchromatine (Figure 3A), telles que les marques H3K27ac et H4K20me1, ainsi qu’une déplétion de la marque H3K9me3 [38]. De plus, des expériences de purification de la chromatine télomérique (PICh) dans plusieurs cellules somatiques murines ont montré l’absence des enzymes responsables de la triméthylation d’H3K9 (SETDB1, pour SET domain bifurcated 1, et SUV39H) et la présence du complexe G9a/GLP ainsi qu’une prévalence de la marque répressive H3K9me2 [25] (Figure 3D). La présence de DAXX et ATRX semble importante pour la structure télomérique dans les cellules somatiques. En effet, la déplétion de la protéine ATRX ou DAXX induit l’apparition de dommages à l’ADN (TIF, pour telomere dysfunctional-induced foci), potentiellement dus à l’absence du variant H3.3 [39]. Les télomères présentent en revanche des marques « classiques » d’hétérochromatine constitutive dans les cellules souches embryonnaires de souris (mES) (Figure 3C) [40]. Il a longtemps été pensé que SUV39H était responsable de l’accumulation de H3K9me3 aux télomères, mais il a été récemment montré qu’une autre enzyme entre ici en jeu : la lysine méthyltransférase SETDB1 [25]. Comme évoqué précédemment, H3K9me3 permet notamment le recrutement d’ATRX et d’HP1 ainsi que l’établissement de la marque H4K20me3 par SUV420H [40]. Comme pour les péricentromères, on observe la présence du variant d’histone H3.3, incorporé par les recrutements d’ATRX et de DAXX [41,42]. Son rôle semble être important pour l’intégrité fonctionnelle des télomères puisque sa déplétion induit l’apparition de dommages [41].
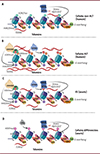 |
Figure 3 La chromatine des télomères. A. La chromatine au niveau des télomères des cellules humaines non-ALT (cellules souches embryonnaires [ES], cellules différenciées et cellules cancéreuses non-ALT) n’est étonnamment pas caractérisée par des marques d’hétérochromatine, mais par les marques d’euchromatine H4K20me1 et H3K27ac. Les télomères sont également caractérisés par la présence du complexe shelterin, empêchant notamment la reconnaissance de l’extrémité comme cassure double brin. On observe également la présence du long ARN non codant TERRA, dont la fonction reste encore mal comprise. Les nucléosomes au niveau des télomères sont intrinsèquement mobiles. B. Les télomères des cellules ALT sont caractérisés par un enrichissement de la marque H3K9me3, dont le dépôt repose sur SETDB1. H3K9me3 ainsi que l’absence d’ATRX participent au processus de recombinaison homologue (HR) illégitime observé dans ces cellules. Ces télomères sont également caractérisés par la présence de la marque H3K36me3, associée à l’élongation de la transcription, ainsi que d’une accumulation de TERRA. C. Dans les cellules ES de souris, la chromatine des télomères est plus caractéristique de l’hétérochromatine constitutive classique. On observe ainsi les marques H3K9me3 et H4K20me3, apposées respectivement par les enzymes SETDB1 et SUV420H. La marque H3K9me3 permet le recrutement de HP1 et ATRX. D. Dans les cellules différenciées de souris, telles que les MEF (murine embryonic fibroblasts), on observe une déplétion de la marque H3K9me3 et l’enrichissement de la marque répressive H3K9me2, apposée par le complexe G9a/GLP (Figure créée avec Biorender.com - agrément EV271F38OB). |
De manière a priori contre-intuitive, SETDB1 et le dépôt de la marque H3K9me3 participent au processus de recombinaison dans les cellules cancéreuses employant le mécanisme d’élongation alternative des télomères (ALT) (Figure 3B) [25]. Il semblerait que cette hétérochromatine favorise un stress replicatif local, crucial pour la mise en place de la voie ALT. En effet, la déplétion de SETDB1 dans ces cellules supprime de manière significative les phénotypes ALT caractéristiques (i.e. des corps nucléaires PML [promyelocytic leukemia protein] associés au télomères [APB] et la présence de cercles d’ADN télomérique simple brin, produits de la recombinaison homologue) [25]. Dans la même lignée, la déplétion du gène SUV39H dans des cellules mES induit une augmentation de SETDB1 et d’H3K9me3 au niveau des télomères et l’apparition de caractéristiques ALT [25]. On observe également la présence de la marque H3K36me3 au niveau des télomères des cellules ALT (Figure 3B) [25], marque associée à l’élongation de la transcription, ainsi qu’une augmentation de l’accumulation du long ARN non-codant TERRA (telomeric repeat-containing RNA) [43]. La présence de TERRA (non-exclusive aux cellules ALT), sa régulation et son rôle sont encore mal compris mais cela semblerait participer à la stabilité des télomères (pour revue, voir [44]). Enfin, plus de 90 % des tumeurs ALT présentent des mutations inactivatrices dans le gène ATRX et son absence participe à la recombinaison observée dans ces cellules [45]. Ceci pourrait être lié à l’absence d’incorporation d’H3.3, puisque la perturbation du dépôt d’H3.3 dans des cellules cancéreuses induit l’apparition de caractéristiques ALT [39].
L’hétérochromatine répressive des rétrotransposons
Comme introduit plus haut, les rétrotransposons, lorsqu’ils sont transcrits, ont la capacité de rétrotranscrire et de s’insérer ailleurs dans le génome. Des mécanismes de restriction ont évolué et peuvent agir à plusieurs étapes du cycle de rétrotransposition. En général, la régulation s’opère d’abord au niveau de la transcription des rétrotransposons par divers processus répressifs (Figure 4). L’utilisation de tel ou tel mécanisme semble dépendre de la nature de l’élément rétrotransposable, de son « âge » (état de dégénération de la séquence), mais aussi du type cellulaire dans lequel il doit être réprimé [28,46,47].
 |
Figure 4 L’hétérochromatine des rétrotransposons. A. De nombreux facteurs sont impliqués dans la répression des rétrotransposons. Les protéines KRAB-ZNF (KZF) et KAP1 permettent le recrutement de SETDB1 sur les ERV de classe I et II et le dépôt de la marque H3K9me3, le recrutement du complexe déacétylase NuRD et le recrutement de DNMT pour la méthylation des CpG. Au niveau des LINE, SUV39H est responsable de l’établissement de la marque H3K9me3. Le recrutement de SUV420H par H3K9me3 et HP1 permet d’établir la marque H4K20me3. Cette dernière peut être reconnue par DNMT1 et favoriser le maintien de la marque CpGme sur les LINE. La marque H3K9me3 peut être reconnue par le complexe HUSH qui permet le maintien de la marque H3K9me3 sur le locus en s’associant avec SETDB1. Le complexe HUSH permet aussi le recrutement du dimère de protéines MORC2 qui participe à la compaction de la chromatine. Le complexe HUSH pourrait également reconnaître des ARN et permettre la répression de novo des jeunes LINE actifs. Dans les cellules souches embryonnaires (ES) de souris, les ERV de classe III sont réprimés par la marque H3K9me2 via l’action du complexe G9a/GLP. Enfin, dans les cellules germinales primordiales (PGC) de souris, MIWI2 reconnaît les piRNA dérivés des ARN d’éléments rétrotransposables et participe à la répression du locus cible (Figure créée avec Biorender.com - agrément RG279Q98BA). B. En fonction du type cellulaire et de l’étape de développement, certains processus sont favorisés par rapport à un autre (d’après [28]) (voir le texte pour les abréviations). |
Les rétrotransposons peuvent être régulés par des modifications d’histones, telles que H3K9me3 et H4K20me3. SETDB1 est impliquée dans l’établissement de la marque H3K9me3. Son recrutement sur les rétrotransposons peut être effectué via les protéines KRAB-ZFP (Krüppelassociated box domain – zinc finger proteins) et KAP1 (KRAB-ZFP-associated protein 1) [46–49]. Au niveau des LINE, la marque H3K9me3 peut également être déposée par SETDB1 via son recrutement par le complexe HUSH (human-silencing hub, composé de TASOR [transcription activation suppressor], PPHLN1 [periphilin 1] et MPP8 [Mphospho8]) [47]. La sous-unité PPHLN1 pourrait reconnaître les transcrits de LINE actifs et médier leur répression de novo. De plus, la sous-unité MPP8 reconnaît la marque H3K9me3 afin de maintenir la marque sur la cible. Le complexe HUSH permet également le recrutement de la protéine MORC2 (MORC family CW-type zinc finger protein 2) qui reconnaît l’ADN double-brin libre et nucléosomal. La dimérisation de deux protéines MORC2 promeut la formation de boucle d’ADN et la compaction de la chromatine [47]. SUV39H est également impliquée dans l’établissement de la marque H3K9me3 au niveau des LINE, recrutée par ZNF512 et ZNF512b [28,31]. Enfin, il est important de noter que contrairement aux SVA, aux LINE et aux ERV de classe I et II enrichis en H3K9me3, les ERV de classe III chez la souris (e.g. MERVL3) sont caractérisés par la marque H3K9me2 apposée par G9a/GLP [47].
La répression des rétrotransposons peut également être médiée par l’action des DNMT et la méthylation des CpG [28,46]. Les DNMT peuvent être recrutées par le complexe KRAB-ZFP/KAP1 [46] ou la marque H4K20me3, pour DNMT1 [33]. La méthylation des CpG semble particulièrement impliquée dans la répression des rétrotransposons dans les cellules somatiques. Dans les cellules ES et les cellules primordiales germinales (PGC), la répression est principalement contrôlée par la méthylation des histones [28].
Dans les cellules mES, les ERV sont également caractérisés par leur enrichissement en histone H3.3 dont le dépôt repose sur le complexe ATRX/DAXX [50]. H3.3 facilite ainsi le recrutement de KAP1 au niveau de certains ERV et la maintenance de la marque H3K9me3. La délétion de ses gènes (H3f3a et H3f3b) induirait une augmentation modérée de l’expression des ERV de type IAP (intracisternal A particle) chez la souris. L’impact de la perte du variant H3.3 sur la transcription des IAP a cependant été remis en cause [51] et son rôle dans la répression des ERV ne semble pas prépondérant.
Enfin, dans les PGC, la répression des rétrotransposons implique également la voie PIWI (P-element induced wimpy testis) [46]. Chez la souris, MIWI2 se lie aux piRNA (PIWI-interacting RNA) dérivés du rétrotransposon sous l’action de clivage de MILI4, et cible les ARN naissants, permettant le recrutement de la machinerie de méthylation de novo de l’ADN. Plusieurs lignes de défense moléculaires ont donc évolué pour assurer la formation d’hétérochromatine au niveau des rétrotransposons. Ces différents mécanismes, parfois redondants, permettent d’assurer une répression transcriptionnelle robuste à chaque étape du développement et de prévenir les évènements de transposition potentiellement délétères.
Conclusion
L’essor et l’amélioration des techniques de protéomique et de séquençage de l’ADN ont permis de montrer que l’hétérochromatine constitutive, longtemps vue comme une structure inerte, est en fait bien plus diverse et dynamique. Finalement, cette dynamique est assez similaire à celle qui distingue l’hétérochromatine facultative régulant les gènes. Sa composition dépend du type de répétition mais aussi du type cellulaire et de l’étape de développement. Dans le cas des éléments transposables, les mécanismes sollicités sont divers et redondants ce qui permet un meilleur contrôle de leur expression et mobilisation. Décrite initialement comme une structure répressive, l’hétérochromatine constitutive semble dans certains cas permettre et même promouvoir la transcription et la recombinaison homologue (i.e. télomères des cellules ALT). L’hétérochromatine peut également jouer un rôle structural comme au niveau des péricentromères où elle participe à la cohésion des chromatides sœurs. Il ne semble donc pas y avoir de règles bien établies pour définir l’hétérochromatine, si ce n’est qu’elle varie en fonction de la nature des séquences sur laquelle elle se forme. Néanmoins, elle est essentielle pour la régulation de nombreuses séquences répétées et sa défaillance est au cœur de nombreuses maladies. On peut, par exemple, citer une certaine catégorie du syndrome ICF (immuno-déficience, instabilité centromérique et dysmorphie faciale), où des mutations du gène codant pour DNMT3B ou de protéines associées entrainent des réarrangements des péricentromères.
Il est encore difficile de comprendre précisément tous les mécanismes moléculaires impliqués dans la formation et le maintien de l’hétérochromatine ; le développement de nouvelles méthodes de protéomique permettra dans le futur d’avoir une vision plus globale des différentes formes et fonctions que l’hétérochromatine peut adopter en fonction du contexte génomique et cellulaire.
Remerciements
Constance Kleijwegt est financée par une bourse individuelle postdoctorale de l’AFM-Téléthon (contrat 24868). Le laboratoire est financé par l’ERC Consolidator Grant « METACHROM », l’ANR « EARLYTEL », la chaire d’excellence France 2030 de l’ANR pour le projet « ENDLOOP ». Le laboratoire est labellisé par la Ligue contre le cancer.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Balzano E, Pelliccia F, Giunta S. Genome (in)stability at tandem repeats. Seminars in Cell & Developmental Biology 2021 ; 113 : 97–112. [CrossRef] [PubMed] [Google Scholar]
- Wicker T, Sabot F, Hua-Van A, et al. A unified classification system for eukaryotic transposable elements. Nat Rev Genet 2007 ; 8 : 973–82. [CrossRef] [PubMed] [Google Scholar]
- Magiorkinis G, Belshaw R, Katzourakis A. ‘There and back again’: revisiting the pathophysiological roles of human endogenous retroviruses in the post-genomic era. Philos Trans R Soc Lond B Biol Sci 2013 ; 368 : 20120504. [CrossRef] [PubMed] [Google Scholar]
- Hancks DC, Kazazian HH. Active Human Retrotransposons: Variation and Disease. Curr Opin Genet Dev 2012 ; 22 : 191–203. [CrossRef] [PubMed] [Google Scholar]
- Senft AD, Macfarlan TS. Transposable elements shape the evolution of mammalian development. Nat Rev Genet 2021 ; 22 : 691–711. [CrossRef] [PubMed] [Google Scholar]
- Meyer TJ, Rosenkrantz JL, Carbone L, et al. Endogenous Retroviruses: With Us and against Us. Front Chem 2017 ; 5. [PubMed] [Google Scholar]
- Kramerov DA, Vassetzky NS. Origin and evolution of SINEs in eukaryotic genomes. Heredity 2011 ; 107 : 487–95. [CrossRef] [PubMed] [Google Scholar]
- Gilbert C, Feschotte C. Horizontal acquisition of transposable elements and viral sequences: patterns and consequences. Curr Opin Genet Develop 2018 ; 49 : 15–24. [CrossRef] [Google Scholar]
- Ivancevic AM, Kortschak RD, Bertozzi T, et al. Horizontal transfer of BovB and L1 retrotransposons in eukaryotes. Gen Biol 2018 ; 19 : 85. [CrossRef] [Google Scholar]
- Ahmed M, Liang P. Transposable Elements Are a Significant Contributor to Tandem Repeats in the Human Genome. International Journal of Genomics 2012 ; 2012 : e947089. [Google Scholar]
- Zattera ML, Bruschi DP. Transposable Elements as a Source of Novel Repetitive DNA in the Eukaryote Genome. Cells 2022 ; 11 : 3373. [CrossRef] [PubMed] [Google Scholar]
- Flynn JM, Yamashita YM. The implications of satellite DNA instability on cellular function and evolution. Seminars in Cell & Developmental Biology 2024 ; 156 : 152–9. [CrossRef] [PubMed] [Google Scholar]
- Showman S, Talbert PB, Xu Y, et al. Expansion of human centromeric arrays in cells undergoing break-induced replication. Cell Rep 2024 ; 43 : 113851. [CrossRef] [PubMed] [Google Scholar]
- Janssen A, Colmenares SU, Karpen GH. Heterochromatin: Guardian of the Genome. Annual Review of Cell and Developmental Biology 2018 ; 34 : 265–88. [CrossRef] [PubMed] [Google Scholar]
- Hancks DC, Kazazian HH. Roles for retrotransposon insertions in human disease. Mob DNA 2016 ; 7 : 9. [CrossRef] [PubMed] [Google Scholar]
- Chuong EB, Elde NC, Feschotte C. Regulatory activities of transposable elements: from conflicts to benefits. Nat Rev Genet 2017 ; 18 : 71–86. [CrossRef] [PubMed] [Google Scholar]
- Zeng C, Onoguchi M, Hamada M. Association analysis of repetitive elements and R-loop formation across species. Mobile DNA 2021 ; 12 : 3. [CrossRef] [PubMed] [Google Scholar]
- Petermann E, Lan L, Zou L. Sources, resolution and physiological relevance of R-loops and RNA-DNA hybrids. Nat Rev Mol Cell Biol 2022 ; 23 : 521–40. [CrossRef] [PubMed] [Google Scholar]
- Takata H, Hanafusa T, Mori T, et al. Chromatin Compaction Protects Genomic DNA from Radiation Damage. PLOS ONE 2013 ; 8 : e75622. [CrossRef] [PubMed] [Google Scholar]
- Fortuny A, Polo SE. The response to DNA damage in heterochromatin domains. Chromosoma 2018 ; 127 : 291–300. [CrossRef] [PubMed] [Google Scholar]
- Schuster-Böckler B, Lehner B. Chromatin organization is a major influence on regional mutation rates in human cancer cells. Nature 2012 ; 488 : 504–7. [CrossRef] [PubMed] [Google Scholar]
- Supek F, Lehner B. Differential DNA mismatch repair underlies mutation rate variation across the human genome. Nature 2015 ; 521 : 81–84. [CrossRef] [PubMed] [Google Scholar]
- Zheng CL, Wang NJ, Chung J, et al. Transcription restores DNA repair to heterochromatin, determining regional mutation rates in cancer genomes. Cell Rep 2014 ; 9 : 1228–34. [CrossRef] [PubMed] [Google Scholar]
- Caridi PC, Delabaere L, Zapotoczny G, et al. And yet, it moves: nuclear and chromatin dynamics of a heterochromatic double-strand break. Philos Trans R Soc Lond B Biol Sci 2017 ; 372 : 20160291. [CrossRef] [PubMed] [Google Scholar]
- Gauchier M, Kan S, Barral A, et al. SETDB1-dependent heterochromatin stimulates alternative lengthening of telomeres. Sci Adv 2019 ; 5 : eaav3673. [CrossRef] [PubMed] [Google Scholar]
- Gieni RS, Chan GKT, Hendzel MJ. Epigenetics regulate centromere formation and kinetochore function. J Cell Biochem 2008 ; 104 : 2027–39. [CrossRef] [PubMed] [Google Scholar]
- Déjardin J. Switching between Epigenetic States at Pericentromeric Heterochromatin. Trends Genet 2015 ; 31 : 661–72. [CrossRef] [PubMed] [Google Scholar]
- Nishibuchi G, Déjardin J. The molecular basis of the organization of repetitive DNA-containing constitutive heterochromatin in mammals. Chromosome Res 2017 ; 25 : 77–87. [CrossRef] [PubMed] [Google Scholar]
- Collings CK, Waddell PJ, Anderson JN. Effects of DNA methylation on nucleosome stability. Nucleic Acids Res 2013 ; 41 : 2918–31. [CrossRef] [PubMed] [Google Scholar]
- Fournier A, Sasai N, Nakao M, et al. The role of methyl-binding proteins in chromatin organization and epigenome maintenance. Brief Funct Genomics 2012 ; 11 : 251–64. [CrossRef] [PubMed] [Google Scholar]
- Ma R, Zhang Y, Zhang J, et al. Targeting pericentric non-consecutive motifs for heterochromatin initiation. Nature 2024 ; 631 : 678–85. [CrossRef] [PubMed] [Google Scholar]
- Iglesias N, Moazed D. Silencing repetitive DNA. eLife 2017 ; 6. [Google Scholar]
- Ren W, Fan H, Grimm SA, et al. DNMT1 reads heterochromatic H4K20me3 to reinforce LINE-1 DNA methylation. Nat Commun 2021 ; 12 : 2490. [CrossRef] [PubMed] [Google Scholar]
- Hahn M, Dambacher S, Dulev S, et al. Suv4-20h2 mediates chromatin compaction and is important for cohesin recruitment to heterochromatin. Genes Dev 2013 ; 27 : 859–72. [CrossRef] [PubMed] [Google Scholar]
- Drané P, Ouararhni K, Depaux A, et al. The death-associated protein DAXX is a novel histone chaperone involved in the replication-independent deposition of H3.3. Genes Dev 2010 ; 24 : 1253–65. [CrossRef] [PubMed] [Google Scholar]
- Morozov VM, Gavrilova EV, Ogryzko VV, et al. Dualistic function of Daxx at centromeric and pericentromeric heterochromatin in normal and stress conditions. Nucleus 2012 ; 3 : 276–85. [CrossRef] [PubMed] [Google Scholar]
- Decottignies A. L’effet de position télomérique - Silence dans le fond ! Med Sci (Paris) 2014 ; 30 : 173–8. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Cubiles MD, Barroso S, Vaquero-Sedas MI, et al. Epigenetic features of human telomeres. Nucleic Acids Res 2018 ; 46 : 2347. [CrossRef] [PubMed] [Google Scholar]
- Li F, Deng Z, Zhang L, et al. ATRX loss induces telomere dysfunction and necessitates induction of alternative lengthening of telomeres during human cell immortalization. EMBO J 2019 ; 38 : e96659. [CrossRef] [PubMed] [Google Scholar]
- Benetti R, Gonzalo S, Jaco I, et al. Suv4-20h deficiency results in telomere elongation and derepression of telomere recombination. J Cell Biol 2007 ; 178 : 925–36. [CrossRef] [PubMed] [Google Scholar]
- Wong LH, Ren H, Williams E, et al. Histone H3.3 incorporation provides a unique and functionally essential telomeric chromatin in embryonic stem cells. Genome Res 2009 ; 19 : 404–14. [CrossRef] [PubMed] [Google Scholar]
- Goldberg AD, Banaszynski LA, Noh K-M, et al. Distinct factors control histone variant H3.3 localization at specific genomic regions. Cell 2010 ; 140 : 678–91. [CrossRef] [PubMed] [Google Scholar]
- Arora R, Lee Y, Wischnewski H, et al. RNaseH1 regulates TERRA-telomeric DNA hybrids and telomere maintenance in ALT tumour cells. Nat Commun 2014 ; 5 : 5220. [CrossRef] [PubMed] [Google Scholar]
- Barral A, Déjardin J. Telomeric Chromatin and TERRA. J Mol Biol 2020 ; 432 : 4244–56. [CrossRef] [PubMed] [Google Scholar]
- Voon HPJ, Hughes JR, Rode C, et al. ATRX Plays a Key Role in Maintaining Silencing at Interstitial Heterochromatic Loci and Imprinted Genes. Cell Rep 2015 ; 11 : 405–18. [CrossRef] [PubMed] [Google Scholar]
- Almeida MV, Vernaz G, Putman ALK, et al. Taming transposable elements in vertebrates: from epigenetic silencing to domestication. Trends Genet 2022 ; 38 : 529–53. [CrossRef] [PubMed] [Google Scholar]
- Fukuda K, Shinkai Y. SETDB1-Mediated Silencing of Retroelements. Viruses 2020 ; 12 : 596. [CrossRef] [PubMed] [Google Scholar]
- Castro-Diaz N, Ecco G, Coluccio A, et al. Evolutionally dynamic L1 regulation in embryonic stem cells. Genes Dev 2014 ; 28 : 1397–409. [CrossRef] [PubMed] [Google Scholar]
- Jacobs FM, Greenberg D, Nguyen N, et al. An evolutionary arms race between KRAB zinc finger genes 91/93 and SVA/L1 retrotransposons. Nature 2014 ; 516 : 242–5. [CrossRef] [PubMed] [Google Scholar]
- Elsässer SJ, Noh K-M, Diaz N, et al. Histone H3.3 is required for endogenous retroviral element silencing in embryonic stem cells. Nature 2015 ; 522 : 240–4. [CrossRef] [PubMed] [Google Scholar]
- Wolf G, Rebollo R, Karimi MM, et al. On the role of H3.3 in retroviral silencing. Nature 2017 ; 548 : E1–3. [CrossRef] [PubMed] [Google Scholar]
- Warburton PE, Hasson D, Guillem F, et al. Analysis of the largest tandemly repeated DNA families in the human genome. BMC Genomics 2008 ; 9 : 533. [CrossRef] [PubMed] [Google Scholar]
- Nurk S, Koren S, Rhie A, et al. The complete sequence of a human genome. Science 2022 ; 376 : 44–53. [CrossRef] [PubMed] [Google Scholar]
- Hoyt SJ, Storer JM, Hartley GA, et al. From telomere to telomere: The transcriptional and epigenetic state of human repeat elements. Science 2022 ; 376 : eabk3112. [CrossRef] [PubMed] [Google Scholar]
- Altemose N, Glennis A, Bzikadze AV, et al. Complete genomic and epigenetic maps of human centromeres. Science 2022 ; 376 : eabl4178. [CrossRef] [PubMed] [Google Scholar]
Liste des tableaux
Les différentes séquences répétées dans le génome humain. Les séquences répétées peuvent être classées en deux catégories: les répétitions en tandem et les répétitions dispersées. Ce Tableau reprend les différentes répétitions retrouvées chez l’homme et la fraction du génome qu’elles représentent (adapté de [1,52–55]). *Les microsatellites peuvent être classés dans les deux catégories puisqu’il s’agit de répétitions en tandem présentes à de multiples endroits dans le génome.
Liste des figures
 |
Figure 1 Les différents rôles de l’hétérochromatine constitutive. L’hétérochromatine est une structure permettant d’assurer la stabilité génomique. Elle est classiquement caractérisée par les marques d’histones H3K9me3 et H4K20me3 et par la méthylation de l’ADN. DSB : double strand break (cassure double brin) (adaptée de [14]) (Figure créée avec Biorender.com - agrément UG270PLMD2). |
| Dans le texte | |
 |
Figure 2 L’hétérochromatine des péricentromères. A. L’hétérochromatine péricentromérique des cellules souches embryonnaires [ES] et différenciées est caractérisée par les marques classiques de l’hétérochromatine, H3K9me3, H4K20me3 et CpGme, apposées respectivement par les enzymes SUV39H, SUV420H et DNMTs. H3K9me3 permet le recrutement de HP1 et ATRX. ATRX permet le recrutement de DAXX, chaperon de l’histone H3.3. B. L’hétérochromatine péricentromérique sur le génome paternel des embryons précoces murins est caractérisée par les marques H3K27me3 et H2AK119Ub, apposées respectivement par PRC2 et PRC1. Enfin, la protéine BEND3 permet le recrutement du complexe déacétylase NuRD (figure créée avec Biorender.com - agrément ED279Q865G). |
| Dans le texte | |
 |
Figure 3 La chromatine des télomères. A. La chromatine au niveau des télomères des cellules humaines non-ALT (cellules souches embryonnaires [ES], cellules différenciées et cellules cancéreuses non-ALT) n’est étonnamment pas caractérisée par des marques d’hétérochromatine, mais par les marques d’euchromatine H4K20me1 et H3K27ac. Les télomères sont également caractérisés par la présence du complexe shelterin, empêchant notamment la reconnaissance de l’extrémité comme cassure double brin. On observe également la présence du long ARN non codant TERRA, dont la fonction reste encore mal comprise. Les nucléosomes au niveau des télomères sont intrinsèquement mobiles. B. Les télomères des cellules ALT sont caractérisés par un enrichissement de la marque H3K9me3, dont le dépôt repose sur SETDB1. H3K9me3 ainsi que l’absence d’ATRX participent au processus de recombinaison homologue (HR) illégitime observé dans ces cellules. Ces télomères sont également caractérisés par la présence de la marque H3K36me3, associée à l’élongation de la transcription, ainsi que d’une accumulation de TERRA. C. Dans les cellules ES de souris, la chromatine des télomères est plus caractéristique de l’hétérochromatine constitutive classique. On observe ainsi les marques H3K9me3 et H4K20me3, apposées respectivement par les enzymes SETDB1 et SUV420H. La marque H3K9me3 permet le recrutement de HP1 et ATRX. D. Dans les cellules différenciées de souris, telles que les MEF (murine embryonic fibroblasts), on observe une déplétion de la marque H3K9me3 et l’enrichissement de la marque répressive H3K9me2, apposée par le complexe G9a/GLP (Figure créée avec Biorender.com - agrément EV271F38OB). |
| Dans le texte | |
 |
Figure 4 L’hétérochromatine des rétrotransposons. A. De nombreux facteurs sont impliqués dans la répression des rétrotransposons. Les protéines KRAB-ZNF (KZF) et KAP1 permettent le recrutement de SETDB1 sur les ERV de classe I et II et le dépôt de la marque H3K9me3, le recrutement du complexe déacétylase NuRD et le recrutement de DNMT pour la méthylation des CpG. Au niveau des LINE, SUV39H est responsable de l’établissement de la marque H3K9me3. Le recrutement de SUV420H par H3K9me3 et HP1 permet d’établir la marque H4K20me3. Cette dernière peut être reconnue par DNMT1 et favoriser le maintien de la marque CpGme sur les LINE. La marque H3K9me3 peut être reconnue par le complexe HUSH qui permet le maintien de la marque H3K9me3 sur le locus en s’associant avec SETDB1. Le complexe HUSH permet aussi le recrutement du dimère de protéines MORC2 qui participe à la compaction de la chromatine. Le complexe HUSH pourrait également reconnaître des ARN et permettre la répression de novo des jeunes LINE actifs. Dans les cellules souches embryonnaires (ES) de souris, les ERV de classe III sont réprimés par la marque H3K9me2 via l’action du complexe G9a/GLP. Enfin, dans les cellules germinales primordiales (PGC) de souris, MIWI2 reconnaît les piRNA dérivés des ARN d’éléments rétrotransposables et participe à la répression du locus cible (Figure créée avec Biorender.com - agrément RG279Q98BA). B. En fonction du type cellulaire et de l’étape de développement, certains processus sont favorisés par rapport à un autre (d’après [28]) (voir le texte pour les abréviations). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




