| Issue |
Med Sci (Paris)
Volume 40, Number 11, Novembre 2024
Chroniques génomiques
|
|
|---|---|---|
| Page(s) | 869 - 871 | |
| Section | Forum | |
| DOI | https://doi.org/10.1051/medsci/2024149 | |
| Published online | 10 December 2024 | |
L’édition de nouvelle génération
New-generation editing
Biologiste, généticien et immunologiste, Président d’Aprogène (Association pour la promotion de la Génomique), 13007 Marseille, France
Abstract
Recent work on bacterial insertion sequences reveals that some of them use an RNA sequence (called Bridge RNA or Seek RNA) to define both donor and target DNA specificity. This opens the way to easy insertion of kilobase DNA sequences at pre-defined sites in the genome, announcing a host of new possibilities. The system still needs a lot of tweaking, as it has only been demonstrated in bacteria, but it holds great promise for genome editing and engineering.
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
La découverte du système CRISPR/Cas9, il y a à peine plus de dix ans [1] (→), a décuplé nos possibilités d’intervention sur les génomes, que ce soit en recherche ou, de plus en plus, pour donner (enfin) une réalité à la thérapie génique [2] (→).
(→) Voir la Nouvelle de H. Gilgenkrantz, m/s n° 12, décembre 2014, page 1066
(→) Voir l’Éditorial de H. Chneiweiss, m/s n° 8-9, août-septembre 2023, page 589
Le système CRISPR et ses dérivés permettent maintenant d’aller assez facilement couper l’ADN en un point spécifique d’un génome complexe ; la réparation de la coupure se fait généralement de manière un peu aléatoire, avec ajout de quelques nucléotides au point de la réparation, ce qui provoque l’inactivation du gène ciblé. On peut ainsi « éteindre » un gène dont la surexpression cause une maladie, dans un but de thérapie génique, et cela peut même être réalisé in vivo [3] (→).
(→) Voir la Chronique de B. Jordan, m/s n° 8-9, août-septembre 2024, page 677
Plus récemment, ces systèmes ont été perfectionnés pour permettre une véritable édition du génome, c’est-à-dire le remplacement programmé d’un nucléotide par un autre, autorisant ainsi la correction de mutations délétères [4]. Mais l’insertion précise en un point du génome d’une séquence de quelques centaines ou milliers de nucléotides reste hors de portée, du moins si l’on souhaite qu’elle se fasse de manière efficace (et non dans une toute petite fraction des cellules traitées). C’est ce qui explique l’intérêt soulevé par la récente publication d’une nouvelle approche utilisant les propriétés d’une recombinase codée par une famille de séquences d’insertion [5, 6] qui est, de plus, entièrement programmable via la séquence d’un ARN guide bispécifique.
Le monde des séquences d’insertion, et le cas de la famille IS110
Les séquences d’insertion (IS pour insertion sequences) sont connues depuis des décennies [7, 8]. Il s’agit de petits éléments transposables, longs de une à deux kilobases environ ; on en connaît des centaines dans les bactéries et chez certains eucaryotes, regroupés en une trentaine de familles d’après leurs similitudes de séquence. Leur ADN code une transposase, une enzyme capable de diriger l’intégration ou l’excision de l’élément. Elles ne s’intègrent pas au hasard dans le génome, et leur ciblage sur certains sites repose en général sur une reconnaissance par la transposase, donc une interaction protéine/ADN qui présente une certaine spécificité mais est difficilement manipulable. Les auteurs se sont intéressés à une famille particulière de ces séquences appelée IS110, qui, lors de l’excision, prend une forme circulaire, s’insère à des sites spécifiques, et dont la transposase a en fait une activité de recombinase. L’étude d’un membre particulier de cette famille, appelé IS621 [9], montre l’existence d’un promoteur dans la région non codante de son ADN, et indique que ce dernier provoque, lorsque l’ADN est sous sa forme circulaire, la transcription d’un ARN non codant long de 177 nucléotides. Lorsqu’il est transcrit in vitro, cet ARN s’associe à la recombinase, et l’ensemble reconstitue l’activité d’insertion de IS621. Cet ARN, que les auteurs appellent bridge RNA (ARN pont), contient des séquences complémentaires d’une part à IS621 et d’autre part à son site d’insertion : il fait effectivement le pont entre l’ADN à insérer et le site d’insertion. La Figure 1 montre la géométrie assez complexe de l’interaction entre l’ARN pont, l’ADN cible (le site d’insertion) et l’ADN donneur (l’ADN IS621) qui implique une hybridation sur les deux brins des deux ADN concernés. Tout l’intérêt du système est que les deux spécificités (donneur et cible) sont portées par un ARN, donc sont programmables ad libitum : on peut ainsi envisager de l’utiliser pour insérer une séquence choisie en un point déterminé du génome, ce qui constitue une avancée considérable. Une autre équipe a obtenu des résultats comparables, publiés presque simultanément [10], et a baptisé seekRNA l’ARN transcrit à partir de la forme circulaire de la séquence d’insertion et qui lui confère sa spécificité d’insertion.
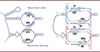 |
Figure 1. L’ARN pont seul (à gauche) et en interaction avec les ADN cible et donneur (à droite). L’ARN pont s’hybride aux deux brins de l’ADN cible (en bleu) via ses séquences LTG (left target guide) et RTG (right target guide) et aux deux brins de l’ADN donneur (en rouge) via ses séquences LDG (Left Donor Guide) et RDG (right donor guide). La recombinase (qui intervient sous forme d’un tétramère) n’est pas figurée. Les séquences indiquées ici sont celles de l’élément transposable IS621 et de son site d’insertion. Extrait partiel et modifié de la figure 2 de [5]. |
Un monde de nouvelles possibilités
Les auteurs ont poursuivi en synthétisant plusieurs éléments IS621 dans lesquels la région de l’ARN pont comporte des séquencescibles différentes, et ont observé un patron d’insertion cohérent avec les zones cibles choisies. Ils ont précisé la spécificité de l’insertion en choisissant des séquences uniques dans l’ADN d’Escherichia coli et ont constaté que l’insertion avait lieu au point choisi dans 60 à 90 % des cas. Ils ont de plus montré que cette spécificité pouvait être améliorée en augmentant la longueur de la région RTG (cf. Figure 1). Ils obtiennent des résultats analogues pour la spécificité de l’interaction avec l’ADN donneur, avec là aussi, possibilité d’amélioration en allongeant la région RDG. Ils ont enfin montré qu’ils pouvaient, dans la même séquence d’ARN pont, modifier à la fois RTG et RDG et observer les spécificités attendues. L’intervention sur la séquence codant l’ARN pont permet bien de manipuler tant la spécificité pour l’ADN donneur que le site d’insertion dans l’ADN : le système est donc programmable avec une assez bonne spécificité, et il existe des moyens pour améliorer cette dernière. Une partie de ces expériences est effectuée avec un plasmide (non répliquant) long de 4,85 kilobases : en présence de la recombinase et de l’ARN pont ad hoc transcrit in vitro, cet ADN est bien inséré dans l’ADN d’E. coli au site choisi, démontrant ainsi la possibilité d’insérer à volonté de grandes séquences d’ADN en un point choisi du génome (d’E. coli pour le moment). Les auteurs montrent aussi que ce système peut être utilisé pour exciser ou inverser des séquences d’ADN dans l’ADN bactérien. C’est donc bien le possible début d’une révolution dans nos possibilités de manipuler les génomes, une révolution sans doute comparable à ce qu’a été la découverte du système CRISPR/ Cas9.
Ce n’est encore qu’un début
L’ARN pont est une découverte importante, mais ce n’est pas encore un système « clefs en main » : la spécificité reste à améliorer, et surtout, pour le moment, l’approche est limitée à E. coli et rien ne prouve que ce système va fonctionner chez les eucaryotes et surtout dans les cellules de mammifères. Cependant, les travaux de Durrant et al. [5] et aussi ceux de Siddiquee et al. [10] suggèrent également que les systèmes d’insertion faisant appel à une reconnaissance ARN/ADN (et non protéine/ ADN) se retrouvent dans beaucoup d’autres éléments transposables, offrant ainsi un large choix pour les développements ultérieurs. Et la rapidité avec laquelle on a pu passer, pour CRISPR/Cas9, de la bactérie à l’homme (deux ou trois ans seulement) incite à l’optimisme. L’édition du génome pourrait ainsi faire un nouveau bond en avant, et ouvrir, à moyen terme, bien de nouvelles possibilités tant en ingénierie génétique que pour la thérapie génique.
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Gilgenkrantz H. La révolution des CRISPR est en marche. Med Sci (Paris) 2014 ; 30 : 1066–9. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Chneiweiss H. 2023 : premiers succès et nouveaux enjeux de l’édition du génome en thérapeutique humaine. Med Sci (Paris) 2023 ; 39 : 589–90. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Jordan B. Thérapie génique in vivo : une approche modulaire. Med Sci (Paris) 2024 ; 40 : 677–679. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Porto EM, Komor AC, Slaymaker IM, Yeo GW. Base editing: advances and therapeutic opportunities. Nat Rev Drug Discov 2020 ; 19 : 839–59. [CrossRef] [PubMed] [Google Scholar]
- Durrant MG, Perry NT, Pai JJ, et al. Bridge RNAs direct programmable recombination of target and donor DNA. Nature 2024 ; 630 : 984–93. [CrossRef] [PubMed] [Google Scholar]
- Hiraizumi M, Perry NT, Durrant MG, et al. Structural mechanism of bridge RNA-guided recombination. Nature 2024 ; 630 : 994–1002. [CrossRef] [PubMed] [Google Scholar]
- Mahillon J, Chandler M. Insertion sequences. Microbiol Mol Biol Rev 1998 ; 62 : 725–74. [CrossRef] [PubMed] [Google Scholar]
- Wells JN, Feschotte C. A Field Guide to Eukaryotic Transposable Elements. Annu Rev Genet 2020 ; 54 : 539–61. [CrossRef] [PubMed] [Google Scholar]
- Choi S, Ohta S, Ohtsubo E. A novel IS element, IS621, of the IS110/IS492 family transposes to a specific site in repetitive extragenic palindromic sequences in Escherichia coli. J Bacteriol 2003 ; 185 : 4891–900. [CrossRef] [PubMed] [Google Scholar]
- Siddiquee R, Pong CH, Hall RM, Ataide SF. A programmable seekRNA guides target selection by IS1111 and IS110 type insertion sequences. Nat Commun 2024 ; 15 : 5235. [Google Scholar]
Liste des figures
 |
Figure 1. L’ARN pont seul (à gauche) et en interaction avec les ADN cible et donneur (à droite). L’ARN pont s’hybride aux deux brins de l’ADN cible (en bleu) via ses séquences LTG (left target guide) et RTG (right target guide) et aux deux brins de l’ADN donneur (en rouge) via ses séquences LDG (Left Donor Guide) et RDG (right donor guide). La recombinase (qui intervient sous forme d’un tétramère) n’est pas figurée. Les séquences indiquées ici sont celles de l’élément transposable IS621 et de son site d’insertion. Extrait partiel et modifié de la figure 2 de [5]. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




