| Issue |
Med Sci (Paris)
Volume 40, Number 5, Mai 2024
|
|
|---|---|---|
| Page(s) | 408 - 411 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2024053 | |
| Published online | 31 May 2024 | |
Le génome d’un bunyavirus ne peut être défini qu’à l’échelle de la population de ses virions
The genome of a bunyavirus can only be defined at the scale of the viral population
1
Plant health institute of Montpellier (PHIM), Université de Montpellier, INRAE, CIRAD, IRD, Institut Agro, Montpellier, France
2
Maladies infectieuses et vecteurs : écologie, génétique, évolution et contrôle (MIVEGEC), Université de Montpellier, CNRS, IRD, Montpellier, France
a michel.yvon@inrae.fr
b yannis.michalakis@ird.fr
c stephane.blanc@inrae.fr
L’ordre des Bunyavirales est un groupe de virus à ARN répandus à travers le monde, et dont la grande diversité est illustrée à la fois par la vaste gamme d’hôtes (plantes ou animaux, dont les êtres humains) pour l’ensemble des espèces virales qui le composent, et par la variété des arthropodes (moustiques, tiques, phlébotomes, culicoïdes, pucerons, thrips1) qui les transmettent, auxquels il faut même ajouter certains rongeurs pour une des familles de ce groupe [1].
Plus spécifiquement, les membres des familles Arenaviridae, Hantaviridae, Nairoviridae, Peribunyaviridae et Phenuiviridae sont responsables de maladies graves du bétail ou de l’espèce humaine, à l’image des fièvres de la Vallée du Rift et de Crimée-Congo [1]. Les espèces membres de la famille Tospoviridae et du genre Tenuivirus constituent également un risque majeur pour les cultures agricoles, comme par exemple le tomato spotted wilt virus (TSWV), le rice stripe virus (RSV), ou encore le maize stipe virus (MSV), respectivement parasites de la tomate, du riz et du maïs [2]. En raison de l’impact considérable sur la santé des plantes, des animaux et des êtres humains, les épidémies de bunyavirus ont des répercussions socio-économiques dans le monde entier.
Le génome des bunyavirus est divisé en plusieurs segments d’ARN simple brin de polarité dite négative car c’est le brin complémentaire qui code les protéines virales. Bien que chez certaines espèces, un ou plusieurs segments d’ARN puisse être ambisens – i.e., les deux polarités de l’ARN peuvent chacune coder une protéine –, les bunyavirus sont généralement désignés comme des virus à ARN simple brin négatif, ce qui signifie que c’est le brin non-codant qui est encapsidé et qui est donc dénommé le « brin viral ». Historiquement, la représentation du génome des bunyavirus s’est appuyée sur un modèle simple : chaque particule virale (virion) incorporerait une copie de chacun des segments d’ARN (négatif ou ambisens) composant le génome viral (Figure 1). Selon ce modèle, une unique particule virale serait capable de provoquer une infection autonome et complète. Cependant, bien que cette représentation persiste dans tous les sites internet de virologie, incluant le site officiel du Comité international pour la taxonomie des virus2 et le site ViralZone3, ainsi que dans les articles de synthèse récemment publiés sur ce groupe de virus [1, 3], cette vision a progressivement été remise en question par une série d’observations reflétant une réalité plus nuancée. Au fil du temps, de nombreuses études ont en effet signalé des situations où les virions ne semblaient pas respecter ce schéma d’emballage génomique rigoureux, et semblaient au contraire renfermer une quantité et une nature de matériel génétique variables ([4], et références citées dans cet article). Les divers indices d’hétérogénéité du matériel génétique contenu dans les virions sont les suivants : des proportions inégales des différents segments d’ARN mesurées dans une population virale, l’emballage des brins d’ARN de polarité complémentaire aux brins viraux, la présence de plusieurs copies d’un même segment au sein d’un virion et, le plus souvent, des virions ne contenant pas tous les segments génomiques. Ces informations qui émanent de plusieurs études portant sur des espèces différentes de bunyavirus et utilisant des méthodes non-quantitatives ou semi-quantitatives, bien que fragmentaires, ont suscité une interrogation fondamentale sur la définition et la constitution du génome des bunyavirus. L’idée que la majorité des particules virales puissent contenir un génome incomplet remet en cause le principe du déclenchement de l’infection par un seul virion, et suggère plutôt un mécanisme de complémentation au sein d’une population de virions.
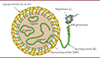 |
Figure 1. Représentation classique d’une particule virale du bunyavirus tomato spotted wilt. Le schéma montre un virion en coupe transversale. Les protéines de surface (en jaune et en orange) comprennent les glycoprotéines Gn et Gc, enchâssées dans une enveloppe membranaire (en bleu clair). Les segments génomiques sont des ARN de polarité négative ou ambisens (brin viral: vRNA) enrobés par de nombreuses copies de la nucléoprotéine N (en vert), organisée en hélicoïdes dont la longueur varie en fonction de la nature du segment. Chaque segment protégé dans la particule virale se présente donc sous la forme d’une ribonucléoprotéine. Une ou quelques copies de la polymérase virale, la protéine L, sont associées à chacun des trois segments. Il convient de noter que dans ce type de représentation, une particule virale semble contenir spécifiquement une copie de chacun des trois segments génomiques: L (grand segment), M (moyen segment), et S (petit segment) (image téléchargée depuis le site internet ViralZone: https://viralzone.expasy.org/9764). |
Avec l’objectif de confirmer et de quantifier cette hétérogénéité génomique des virions, qui soulève de nombreuses interrogations biologiques et évolutives [5], nous avons concentré nos efforts sur le bunyavirus tomato spotted wilt [4]. Une analyse des images de cryo-microscopie électronique a révélé la grande variabilité de la taille des virions (Figure 2A), dont le diamètre peut varier de 65 à 135 nanomètres. Grâce à l’utilisation complémentaire de la tomographie, nous avons constaté que le contenu ribonucléoprotéique de ces virions est également très variable, même entre particules de même taille, la quantité d’ARN associé à la protéine N variant d’un facteur 1 à 4.
 |
Figure 2. Illustration de la composition génétique hétérogène des virions individuels et de celle, homogène, de la population de tomato spotted wilt virus. A. Une population de tomato spotted wilt virus purifiée est présentée sous la forme d’un cliché de cryo-microscopie électronique (cliché Joséphine Lai-Kee-Him), où l’hétérogénéité de taille est visible entre certaines particules de petit (flèches noires) ou grand (flèches blanches) diamètre. B. Notre interprétation des observations tomographiques et des reconstructions en trois dimensions (3D) de l’intérieur des particules est ici illustrée par l’absence de certains segments dans certaines particules virales, ou par la présence de plusieurs copies d’un même segment dans d’autres. Les segments L, M et S sont respectivement représentés en vert, bleu et orange. Il est à noter que la quantité de ribonucléoprotéines contenue dans une particule virale n’est pas corrélée à la taille de la particule. C. Fréquences relatives de chacun des segments S, M et L, dans leur polarité virale (v) ou complémentaire (vc), mesurées dans des populations virales purifiées à partir de 24 plantes infectées en parallèle. La distribution des valeurs dans ces 24 plantes est représentée sous forme de diagramme «en boîte». |
La reconstruction en trois dimensions des volumes internes des virions correspondant aux segments génomiques, bien qu’avec une faible résolution, a fait apparaître un ou plusieurs volumes distincts dans ces virions. Chacun de ces volumes internes pouvant être interprété comme un segment génomique, ces reconstructions tomographiques semblent indiquer que chaque virion contient un nombre très variable de segments différents, ou de copies d’un même segment (Figure 2B).
La diversité du matériel génétique contenu dans les virions s’est avérée encore plus grande qu’on ne le soupçonnait, puisque nous avons montré que les deux polarités de chaque segment (brin viral et brin complémentaire) peuvent être encapsidées dans des proportions importantes au sein des populations de virions. Cette observation a pu être confirmée en utilisant deux techniques différentes : séquençage par nanopore direct de l’ARN et RT-PCR (reverse transcriptase-polymerase chain reaction) quantitative spécifique des brins viral et complémentaire pour les différents segments génomiques. Les résultats obtenus avec cette dernière technique indiquent que le brin complémentaire représente approximativement 10 %, 30 %, et plus de 50 % du total du segment (i.e., brin viral + brin complémentaire) respectivement pour les segments L, S, et M. Nous avons également mis en évidence une forte corrélation entre les proportions du brin viral et du brin complémentaire pour chaque segment, mesurées dans les tissus infectés d’une part, et dans les particules virales purifiées à partir de ces tissus d’autre part. Ce résultat suggère qu’un segment d’ARN génomique produit dans la cellule infectée, quelle que soit son identité et sa polarité, a la même probabilité d’être encapsidé dans la population de virions issue de cette cellule.
Après avoir constaté que les particules virales diffèrent grandement quant au nombre, à l’identité, et à la polarité des segments qu’elles renferment, nous avons conclu que le génome de ce bunyavirus ne peut pas être appréhendé au niveau du virion individuel mais uniquement à un niveau supérieur, celui de la population de virions. Nous avons donc évalué la fréquence relative de chacune des polarités de chaque segment génomique au sein de 24 populations de tomato spotted wilt virus, chacune de ces populations de virions correspondant à une plante Nicotiana rustica infectée. Nous avons ainsi établi la première « formule génomique » d’un bunyavirus, définie comme la distribution des fréquences relatives des segments et de chaque polarité de ces segments dans une population virale. Les résultats illustrés dans la Figure 2C montrent que, contrairement aux compositions génomiques des virions individuels, celles des populations de virions des 24 plantes infectées (analysées en parallèle) sont similaires.
Nous avions précédemment suggéré, à propos d’un virus multipartite4, que des variations de la formule génomique influent sur la régulation de l’expression des gènes, en ajustant leur nombre de copies dans divers environnements [6]. La possibilité d’une modification de la formule génomique des bunyavirus en fonction de l’environnement, par exemple selon l’espèce hôte, et la possibilité de modifier rapidement l’expression des gènes viraux par l’intermédiaire des modifications de cette formule, sont désormais ouvertes à l’investigation.
En révélant un niveau supérieur d’organisation des bunyavirus, la population des virions, comme un possible niveau de régulation clé de leur cycle de vie, nos travaux de recherche contribuent à redéfinir le cadre conceptuel de ces virus : ils doivent désormais être considérés comme des systèmes génétiques complexes dans lesquels les particules virales sont interdépendantes, rappelant le fonctionnement des virus multipartites qui encapsident séparément chacun de leurs segments génomiques [6–8]. La reconnaissance de l’hétérogénéité génomique des bunyavirus au niveau des virions, opposée à l’homogénéité génomique au niveau populationnel, ouvre de nouvelles perspectives dans la compréhension des mécanismes de propagation, de pathogénicité et d’évolution virale.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Boshra H. An overview of the infectious cycle of Bunyaviruses. Viruses 2022; 14 : 2139. [CrossRef] [PubMed] [Google Scholar]
- Kormelink R, Verchot J, Tao X, Desbiez C. The Bunyavirales: The plant-infecting counterparts. Viruses 2021; 13 : 842. [CrossRef] [PubMed] [Google Scholar]
- Elliott RM. Orthobunyaviruses: recent genetic and structural insights. Nat Rev Microbiol 2014 ; 12 : 673–685. [CrossRef] [PubMed] [Google Scholar]
- Yvon M, German TL, Ullman DE, et al. The genome of a bunyavirus cannot be defined at the level of the viral particle but only at the scale of the viral population. Proc Natl Acad Sci USA 2023 ; 120 : e2309412120. [CrossRef] [PubMed] [Google Scholar]
- Michalakis Y, Blanc S. Editorial overview: Multicomponent viral systems. Curr Opin Virol 2018 ; 33 : VI–IX. [CrossRef] [PubMed] [Google Scholar]
- Gallet R, Di Mattia J, Ravel S, et al. Gene copy number variations at the within-host population level modulate gene expression in a multipartite virus. Virus Evol 2022; 8 : veac058. [CrossRef] [PubMed] [Google Scholar]
- Michalakis Y, Blanc S. The curious strategy of multipartite viruses. Annu Rev Virol 2020; 7 : 203–18. [CrossRef] [PubMed] [Google Scholar]
- Bermúdez-Méndez E, Bronsvoort KF, Zwart MP, et al. Incomplete bunyavirus particles can cooperatively support virus infection and spread. PLoS Biol 2022; 20 : e3001870. [CrossRef] [PubMed] [Google Scholar]
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Représentation classique d’une particule virale du bunyavirus tomato spotted wilt. Le schéma montre un virion en coupe transversale. Les protéines de surface (en jaune et en orange) comprennent les glycoprotéines Gn et Gc, enchâssées dans une enveloppe membranaire (en bleu clair). Les segments génomiques sont des ARN de polarité négative ou ambisens (brin viral: vRNA) enrobés par de nombreuses copies de la nucléoprotéine N (en vert), organisée en hélicoïdes dont la longueur varie en fonction de la nature du segment. Chaque segment protégé dans la particule virale se présente donc sous la forme d’une ribonucléoprotéine. Une ou quelques copies de la polymérase virale, la protéine L, sont associées à chacun des trois segments. Il convient de noter que dans ce type de représentation, une particule virale semble contenir spécifiquement une copie de chacun des trois segments génomiques: L (grand segment), M (moyen segment), et S (petit segment) (image téléchargée depuis le site internet ViralZone: https://viralzone.expasy.org/9764). |
| Dans le texte | |
 |
Figure 2. Illustration de la composition génétique hétérogène des virions individuels et de celle, homogène, de la population de tomato spotted wilt virus. A. Une population de tomato spotted wilt virus purifiée est présentée sous la forme d’un cliché de cryo-microscopie électronique (cliché Joséphine Lai-Kee-Him), où l’hétérogénéité de taille est visible entre certaines particules de petit (flèches noires) ou grand (flèches blanches) diamètre. B. Notre interprétation des observations tomographiques et des reconstructions en trois dimensions (3D) de l’intérieur des particules est ici illustrée par l’absence de certains segments dans certaines particules virales, ou par la présence de plusieurs copies d’un même segment dans d’autres. Les segments L, M et S sont respectivement représentés en vert, bleu et orange. Il est à noter que la quantité de ribonucléoprotéines contenue dans une particule virale n’est pas corrélée à la taille de la particule. C. Fréquences relatives de chacun des segments S, M et L, dans leur polarité virale (v) ou complémentaire (vc), mesurées dans des populations virales purifiées à partir de 24 plantes infectées en parallèle. La distribution des valeurs dans ces 24 plantes est représentée sous forme de diagramme «en boîte». |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




