| Issue |
Med Sci (Paris)
Volume 40, Number 5, Mai 2024
|
|
|---|---|---|
| Page(s) | 412 - 413 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2024057 | |
| Published online | 31 May 2024 | |
La transcription, clé de voûte de l’architecture du chromosome bactérien
Transcription: The backbone of bacterial chromosome architecture
Institut Pasteur, CNRS UMR3525, Université Paris Cité, Unité Régulation spatiale des génomes, Paris, France
Chez les bactéries, l’information génétique est typiquement portée par une, parfois deux, rarement plus, molécules d’ADN circulaire d’environ 1 à 10 Mb, comportant une origine de réplication. Ce chromosome bactérien est stocké dans le nucléoïde, un compartiment qui, contrairement au noyau des cellules eucaryotes, n’est pas délimité par une membrane. La double hélice d’ADN est un très long polymère, surenroulé et structuré en différents domaines [1, 2]. Cette organisation chromosomique permet d’empaqueter une molécule de quelques mm dans un volume contraint de l’ordre du μm3, tout en exploitant l’information génétique qu’elle contient afin de diriger la croissance et la division cellulaire, ou de réagir à des changements de l’environnement. La structure tridimensionnelle du chromosome peut ainsi jouer un rôle important pour la régulation de l’expression génique, ainsi que pour la réparation et la réplication de la molécule d’ADN. Cette organisation 3D peut également varier en fonction de l’état métabolique de la cellule [3].
Chez la bactérie Escherichia coli, dont le génome a été très étudié, des expériences ont mis en évidence une subdivision du chromosome en macrodomaines correspondant notamment à l’origine et à la terminaison de la réplication de la molécule d’ADN, et aux bras chromosomiques entre ces deux régions [4]. À l’intérieur de ces macrodomaines, deux segments d’ADN interagissent plus fréquemment que s’ils étaient situés à la même distance dans des macrodomaines différents. Ces macrodomaines ont pu être subdivisés eux-mêmes en sous-domaines délimités par des gènes fortement exprimés [3]. La réplication et la transcription de l’ADN jouent ainsi un rôle structurant dans l’organisation du chromosome bactérien. Cependant, l’impact local d’une plage de transcription sur l’organisation du chromosome reste mal caractérisé, bien que l’on sache que les processus biologiques liés à l’ADN modifient localement sa structure. Par exemple, lors de la réplication ou de la transcription, la double hélice d’ADN doit être ouverte, ce qui produit des contraintes physiques sur le polymère et un surenroulement de la molécule [5, 6]. Les progrès récents en imagerie ainsi que la technique de capture de conformation chromosomique (Hi-C) pour analyser la structure tridimensionnelle moyenne d’un génome ont permis d’étudier l’impact de la transcription sur l’organisation du nucléoïde avec un degré de résolution sans précédent [7]. En particulier, il devenait possible de déterminer comment une unité de transcription influe localement sur la structure du chromosome bactérien, et dans quelle mesure la somme de ces effets locaux peut rendre compte de propriétés organisationnelles du génome à plus grande échelle.
Des microdomaines induits par la transcription
L’analyse du repliement tridimensionnel du chromosome de la bactérie E. coli a ainsi révélé la présence de dizaines de microdomaines couvrant de quelques centaines à quelques milliers de paires de bases, dont nous avons montré qu’ils correspondent aux régions transcrites du génome [8]. Ces microdomaines (transcription-induced domains, TID) isolent les unes des autres les régions avoisinantes. De plus, des TID situés à proximité les uns des autres tendent à se regrouper entre eux. L’ajout de rifampicine, un antibiotique inhibant l’ARN polymérase bactérienne, dans le milieu de culture des bactéries a conduit à la disparition des microdomaines [8]. Pour caractériser plus finement ces microdomaines, nous avons utilisé une approche expérimentale permettant d’activer une seule unité de transcription dans le génome bactérien. Cette approche exploite deux propriétés de l’ARN polymérase du bactériophage T7 : sa résistance à la rifampicine et sa spécificité de transcrire à partir d’une séquence promotrice de l’ADN de ce bactériophage préalablement introduite dans le génome de E. coli. Après avoir intégré dans le génome bactérien un promoteur du phage T7, et placé l’ARN polymérase de ce bactériophage sous le contrôle d’un promoteur inductible, nous avons vérifié qu’en présence de rifampicine ajoutée au milieu de culture, l’ARN polymérase du phage T7 reste capable de transcription à partir du seul promoteur phagique du chromosome bactérien. Cela conduit à l’apparition d’une unique unité de transcription active au milieu d’un génome par ailleurs « éteint ». L’induction contrôlée de cette unité de transcription a ainsi permis de confirmer que la transcription induit la formation d’un microdomaine TID, isolé du reste du génome [8]. En combinant des paires d’unités de transcription activées spécifiquement grâce au système du phage T7 sus-décrit, nous avons également confirmé et directement visualisé par la technique Hi-C l’effet de surenroulement de la transcription sur la molécule d’ADN, avec une accumulation de supertours « négatifs » en amont de l’unité de transcription, et de supertours « positifs » en aval [5, 6, 9]. Nos résultats indiquent que le chromosome est structuré en une succession de TID induits par la transcription (Figure 1). Nous avons ensuite cherché à déterminer dans quelle mesure ces unités influent localement sur la dynamique de l’organisation de l’ADN.
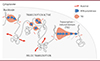 |
Figure 1. Impact de la transcription sur le repliement d’un chromosome bactérien. L’ARN polymérase est représentée en bleu. En rouge sont représentés les domaines induits par la transcription (transcription-induced domains, TID), qui sont isolés des régions adjacentes. Les TID sont en périphérie du nucléoïde à la suite de leur transcription, et sont moins mobiles que les régions non transcrites (flèches rouges). |
Les microdomaines transcrits sont localisés en périphérie de la cellule
Pour analyser par microscopie la localisation des unités transcrites dans le nucléoïde, nous avons étiqueté une région transcrite et une région non-transcrite par des marqueurs fluorescents (collaboration avec Olivier Espeli au Collège de France, Paris). Nous avons constaté une localisation plus fréquente du marqueur de la région transcrite en périphérie du nucléoïde. L’inactivation de la transcription a entraîné la relocalisation de la région auparavant transcrite vers le centre du nucléoïde, tandis que l’activation transcriptionnelle de la région auparavant non transcrite, à l’aide du système du bactériophage T7 décrit précédemment, l’a déplacée vers la périphérie du nucléoïde. De plus, en observant la mobilité des étiquettes de marquage en fonction de l’état transcriptionnel de la région marquée, nous avons constaté que lorsque la région était transcrite, elle était moins mobile au sein du nucléoïde. Nous en avons conclu que les régions transcrites du génome bactérien sont localisées vers la périphérie du nucléoïde, et que leur mobilité est plus contrainte que celle des régions non transcrites (Figure 1).
La transcription génique joue ainsi un rôle crucial dans l’architecture du nucléoïde bactérien. Les TID apparaissent comme des unités fondamentales de l’organisation des domaines chromosomiques, modulant les interactions entre les régions génomiques, leur localisation dans le nucléoïde, et contraignant leur mobilité. La transcription est à l’origine d’une multitude de contraintes mécaniques locales, dont la résultante a pour effet de compacter et organiser le chromosome bactérien. Voilà pourquoi, en présence de rifampicine inhibant la transcription, le nucléoïde se décondense et occupe un volume bien plus grand qu’à l’état basal.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Kleckner N, Fisher JK, Stouf M, et al. The bacterial nucleoid: nature, dynamics and sister segregation. Curr Opin Microbiol 2014 ; 22 : 127–137. [CrossRef] [PubMed] [Google Scholar]
- Lioy VS, Junier I, Boccard F. Multiscale dynamic structuring of bacterial chromosomes. Annu Rev. Microbiol 2021; 75 : 541–61. [CrossRef] [PubMed] [Google Scholar]
- Marbouty M, Le Gall A, Cattoni DI, et al. Condensin- and replication-mediated bacterial chromosome folding and origin condensation revealed by Hi-C and super-resolution imaging. Mol Cell 2015 ; 59 : 588–602. [CrossRef] [PubMed] [Google Scholar]
- Valens M, Penaud S, Rossignol M, et al. Macrodomain organization of the Escherichia coli chromosome. EMBO J 2004 ; 23 : 4330–4341. [CrossRef] [PubMed] [Google Scholar]
- Liu LF, Wang JC. Supercoiling of the DNA template during transcription. Proc Natl Acad Sci 1987 ; 84 : 7024–7027. [CrossRef] [PubMed] [Google Scholar]
- Visser BJ, Sharma S, Chen PJ, et al. Psoralen mapping reveals a bacterial genome supercoiling landscape dominated by transcription. Nucleic Acids Res 2022; 50 : 4436–49. [Google Scholar]
- Cockram C, Thierry A, Gorlas A, et al. Euryarchaeal genomes are folded into SMC-dependent loops and domains, but lack transcription-mediated compartmentalization. Mol Cell 2021; 81 : 459–72.e10. [CrossRef] [PubMed] [Google Scholar]
- Bignaud A, Cockram C, Borde C, et al. Transcription-induced domains form the elementary constraining building blocks of bacterial chromosomes. Nat Struct Mol Biol 2024 ; 31 : 489–97. [CrossRef] [PubMed] [Google Scholar]
- Guo MS, Kawamura R, Littlehale ML, et al. High-resolution, genome-wide mapping of positive supercoiling in chromosomes. eLife 2021 ; 10 : e67236. [CrossRef] [PubMed] [Google Scholar]
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Impact de la transcription sur le repliement d’un chromosome bactérien. L’ARN polymérase est représentée en bleu. En rouge sont représentés les domaines induits par la transcription (transcription-induced domains, TID), qui sont isolés des régions adjacentes. Les TID sont en périphérie du nucléoïde à la suite de leur transcription, et sont moins mobiles que les régions non transcrites (flèches rouges). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




