| Issue |
Med Sci (Paris)
Volume 39, Number 8-9, Août–Septembre 2023
Nos jeunes pousses ont du talent !
|
|
|---|---|---|
| Page(s) | 669 - 675 | |
| Section | Partenariat médecine/sciences - Écoles doctorales - Masters | |
| DOI | https://doi.org/10.1051/medsci/2023103 | |
| Published online | 11 September 2023 | |
Les bactériophages, nouveaux auteurs du génome
Bacteriophages, new authors of the genome
1
Master 2 Microbiologie intégrative et fondamentale, Aix Marseille université, Marseille, France
2
Aix Marseille université, CNRS, LCB UMR7283, IMM, Marseille, France
a amelie.astezan@etu.univ-amu.fr
b eliot.david@etu.univ-amu.fr
c jade.chargelegue@etu.univ-amu.fr
d julien.giraud@etu.univ-amu.fr
e clementine.saunier@etu.univ-amu.fr
f aussel@imm.cnrs.fr
Dans le cadre de l’unité d’enseignement « Rédiger en sciences » proposée par Aix-Marseille université, les étudiants du Master 2 Microbiologie intégrative et fondamentale (MIF) - en partenariat avec l’institut de microbiologie, bioénergies et biotechnologie (IM2B) - ont été confrontés aux exigences de l’écriture scientifique. Trois thématiques leur ont été proposées en relation étroite avec les laboratoires de recherche du tissu local : l’homéostasie des métaux chez les pathogènes, la synthèse de la paroi bactérienne et les systèmes anti-CRISPR des bactériophages. Les étudiants ont ainsi rédigé une nouvelle soulignant les résultats majeurs et l’originalité des articles étudiés. Complété par un entretien avec leurs auteurs, l’ensemble offre un éclairage original sur la compréhension du vivant dans le domaine de la microbiologie et de la santé.
© 2023 médecine/sciences – Inserm
L’actualité scientifique vue par les étudiants du Master 2 Microbiologie d’Aix-Marseille université (AMU), parcours microbiologie intégrative et fondamentale (MIF), unité d’enseignement « Rédiger en sciences »

Responsable de l’Unité d’Enseignement
Laurent Aussel
Équipe pédagogique
Laurent Aussel - Professeur, Aix-Marseille Université - aussel@imm.cnrs.fr
Amel Latifi - Professeur, Aix-Marseille Université - latifi@imm.cnrs.fr
Site web https://sciences.univ-amu.fr/fr/formation/masters/mastermicrobiologie
Série coordonnée par Sophie Sibéril.
Présents dans l’ensemble des écosystèmes, les bactériophages (appelés communément phages) sont l’objet d’une exceptionnelle diversité et sont aujourd’hui au cœur de la recherche fondamentale. Cette famille de virus a la particularité d’infecter spécifiquement les bactéries, cette propriété les rendant inertes à l’égard des cellules eucaryotes. Les phages virulents, qui peuvent provoquer la lyse des bactéries cibles, réalisent un cycle viral productif qui permet la formation de nouveaux virions. Après l’infection des bactéries hôtes, les phages prolifèrent avant de provoquer leur lyse et d’infecter d’autres bactéries. Dans le cas des phages tempérés, le génome viral intègre celui de la bactérie cible par des mécanismes de recombinaison. Les phages tempérés ne provoquent pas la lyse des bactéries et restent en dormance sous la forme de prophages. La dépendance des phages aux bactéries reflète une relation étroite entre l’hôte et son parasite obligatoire. La co-évolution de ces deux derniers, leur co-adaptation suite à des mécanismes d’attaque et de défense, ont favorisé l’apparition chez les bactéries de systèmes moléculaires de résistance aux phages. En parallèle, les phages se sont adaptés en développant différents mécanismes visant à surmonter cette résistance. Ainsi, les bactéries ont acquis un système de résistance aux phages, généralement comparé au système immunitaire des vertébrés, le système CRISPR-Cas (clustered regularly interspaced short palindromic repeat - CRISPR associated proteins). Lors d’une « primo-infection » d’une bactérie par un phage, un fragment de l’ADN phagique intègre le génome bactérien. Sa transcription en ARN induit la création d’un ARN guide du complexe CRISPR-Cas9 qui, lors d’une nouvelle infection par le même bactériophage, permet la reconnaissance de cet ADN phagique et son clivage. Ce système, généralement assimilé à une paire de ciseaux moléculaires, assure la dégradation spécifique du génome du phage. Les phages se sont adaptés en acquérant, au cours de l’évolution, des protéines anti-CRISPR (Acr) leur permettant de ne pas être sensibles à l’action de la Cas9.
Le système CRISPR-Cas9 a été adapté pour permettre la mise au point d’un système universel d’édition de l’ADN. Cette technique ouvre notamment des perspectives pour l’édition du génome humain, qui, bien que soulevant des questions éthiques, représente une piste thérapeutique prometteuse dans le cadre de la thérapie génique. La découverte des Acr apporte de nouveaux outils pour le contrôle de la technique CRISPR, notamment pour bloquer ce système quand cela est nécessaire. Cette nouvelle détaille le fonctionnement des Acr, et plus particulièrement la découverte d’AcrIIA6, ainsi que leurs intérêts pour l’amélioration de l’outil d’édition CRISPR.
CRISPR-Cas9, un système immunitaire bactérien et un outil d’édition du génome
Chez les bactéries, le système CRISPR-Cas9 est un mécanisme de défense qui cible les molécules d’ADN exogènes. Il assure la défense contre les bactériophages, mais peut aussi avoir un effet sur certains plasmides. Ce mécanisme s’apparente à un système immunitaire adaptatif puisqu’il permet à la cellule de garder en « mémoire » dans son génome la séquence d’un fragment d’ADN exogène pour s’en défendre plus efficacement lors d’une rencontre ultérieure. Pour acquérir cette « mémoire », la cellule intègre le fragment d’ADN appelé espaceur (spacer en anglais) dans son génome au niveau du locus CRISPR (Figure 1.1.a). Cette intégration s’opère grâce à l’action de deux protéines, Cas1 et Cas2, dont les modes d’action ne sont pas totalement élucidés. La transcription de l’espaceur phagique ayant intégré le génome bactérien va permettre la formation d’un ARN guide (ARNg) qui va se lier à l’endonucléase Cas9 pour former un complexe de surveillance (Figure 1.1.b). Le complexe, présent dans le cytosol, va être capable de reconnaître de façon spécifique le même ADN exogène lors d’une nouvelle infection (Figure 1.1.c). Cette spécificité est en partie liée à la reconnaissance par le complexe CRISPR-Cas9 d’une courte séquence d’ADN, appelée PAM (protospacer adjacent motif), adjacente à la région ciblée par la Cas9. Cette séquence PAM permet ainsi la discrimination entre l’ADN bactérien et l’ADN phagique.
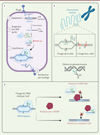 |
Figure 1. Le « système immunitaire » bactérien CRISPR-Cas9 et l’édition du génome humain. 1. Schématisation du fonctionnement du système CRISPR-Cas9 bactérien : (a) Incorporation de l’ADN viral au sein du chromosome bactérien ; (b) L’enzyme Cas9 forme un complexe avec l’ARN issu de la transcription de l’espaceur (crARN) et un ARN transactivateur (tracrARN) ; (c) L’enzyme Cas9 clive l’ADN viral, bloquant alors sa réplication. (d) Le même phage infecte à nouveau une bactérie immunisée. 2. Utilisation de CRISPRCas9 pour la construction d’un outil d’édition du génome humain. Un clivage hors cible constitue une limitation à l’application thérapeutique de la technique d’édition. 3. Les protéines anti- CRISPR (Acr), protéines phagiques induisant l’inhibition du système immunitaire CRISPRCas9, peuvent agir par différents mécanismes qui empêchent le complexe de se lier à l’ADN cible (figure réalisée avec BioRender). |
En 2020, le prix Nobel de chimie a été décerné à Emmanuelle Charpentier et Jennifer Doudna pour avoir adapté le système CRISPR-Cas9 en un outil d’édition de l’ADN, simple d’utilisation et d’une grande précision [1]. La synthèse en laboratoire d’un ARN guide, pris en charge par Cas9, permet de cibler un fragment d’ADN spécifique à cliver. L’outil d’édition du génome CRISPR-Cas9 possède cependant une limite majeure : l’apparition de coupures aspécifiques dites hors cible (Figure 1.2). L’étude de la coévolution entre les phages et les bactéries, notamment des systèmes phagiques mis en place pour répondre à l’action du système CRISPR-Cas9, a permis la découverte des protéines anti-CRISPR (Acr). Ces protéines sont capables d’interférer avec le complexe de surveillance. Des études récentes [3, 4, 9] fondées sur l’analyse structurale et fonctionnelle d’Acr récemment découvertes, ont permis de mieux comprendre les mécanismes d’actions de ces Acr.
Les protéines anti-CRISPR, une puissante réponse adaptative des phages
Les protéines Acr ont été décrites pour la première fois en 2013 [2]. Cinq gènes codant des protéines Acr ont été identifiés dans le génome d’un phage infectant Pseudomonas aeruginosa. À ce jour, une centaine d’Acr ont été identifiées et de nouvelles sont régulièrement découvertes. De façon inattendue, ces protéines ne présentent aucune similarité de séquences avec des protéines de fonction connue. Généralement de petite taille, elles ne partagent pas de caractéristiques communes, ce qui rend leur identification et leur caractérisation complexes. L’emploi d’outils bio-informatiques comme la recherche d’homologies de séquences protéiques et structurales au sein de banques de données de protéines déjà connues n’est alors pas suffisant. Dans le but d’identifier de nouvelles Acr, le génome d’un phage (D4276) contenant le gène codant la protéine AcrIIA5 a été comparé à un autre génome de phage (D1811) infectant la même souche bactérienne, Streptococcus thermophilus. Cette comparaison, réalisée par l’équipe de Sylvain Moineau, a permis d’identifier chez S. thermophilus une séquence codant une possible protéine Acr, appelée AcrIIA6 [3].
Des expériences « d’immunisation » de S. thermophilus avec le phage D1811 ont permis l’intégration d’un espaceur spécifique du phage au sein du chromosome bactérien. En infectant avec ce même phage des bactéries portant différents Acr (AcrIIA2, AcrIIA4 et AcrIIA6) et en comparant la viabilité phagique à l’issue de l’infection, les auteurs de l’étude ont démontré que seule AcrIIA6 assurait la protection du phage contre le système CRISPR-Cas de la bactérie [3]. S. thermophilus, immunisée par le système CRISPR-Cas9, peut ainsi être rendue sensible à un phage grâce à la production d’AcrIIA6 qui inhibe spécifiquement le système CRISPR-Cas9 de la bactérie [3]. Dans une autre étude menée dans le cadre d’une collaboration entre les équipes de Sylvain Moineau et d'Adeline Goulet, les interactions entre les protéines AcrIIA6 et la protéine Cas9 ont été analysées par des expériences de BioLayer interferometry. Cette technique, qui permet d’analyser les interactions (associations et dissociations) entre deux molécules, a pu démontrer une forte interaction entre AcrIIA6 et Cas9 et un très faible taux de dissociation [4]. L’étude structurale d’AcrIIA6 par cristallographie a également mis en évidence sa capacité à former un dimère. Cette propriété lui permet de se lier à deux protéines Cas9, lui conférant un important pouvoir d’inhibition [4]. Afin de mieux comprendre le mécanisme d’inhibition par AcrIIA6, la structure tridimensionnelle du complexe AcrIIA6-Cas9 a été déterminée par des expériences de cryomicroscopie électronique (CryoEM). Jusqu’alors, une étude similaire avait déjà été réalisée avec AcrIIA4, une Acr inhibant également la reconnaissance de l’ADN cible, et avait montré qu’AcrIIA4 se fixait au niveau du domaine d’interaction Cas9-ADN. Cette dernière rentre alors en compétition avec l’ADN double brin cible en interagissant avec le domaine de liaison à la séquence PAM, empêchant ainsi l’enzyme de se fixer au matériel génétique exogène et de le cliver (Figure 2a). En revanche, les analyses structurales menées sur AcrIIA6 ont montré que cette dernière ne se liait pas sur le site de reconnaissance du PAM mais sur la face opposée de l’enzyme, agissant ainsi par inhibition allostérique et en empêchant les modifications de conformation de Cas9 nécessaires à sa liaison au domaine PAM (Figure 2b) [4]. Cette nouvelle découverte sur l’inhibition de Cas9 par AcrIIA6 ouvre ainsi la voie à une meilleure compréhension et donc un meilleur contrôle de l’outil CRISPR-Cas9 [5]. La protéine AcrIIA6, comme AcrIIA4, empêche le clivage de l’ADN exogène par Cas9, avec l’avantage non négligeable pour la protéine AcrIIA6 d’inhiber simultanément deux enzymes Cas9 [4].
 |
Figure 2. Comparaison des modes d’action d’AcrIIA4 et d’AcrIIA6. (A) Mode d’action d’AcrIIA4. (1) Formation du complexe de surveillance avec Cas9 (gris) et l’ARN guide (violet). (2) AcrIIA4 (vert) se fixe sur le site de liaison au PAM (rose) responsable de la reconnaissance de l’ADN cible. (3) La séquence PAM de l’ADN cible ne peut être reconnue par le complexe de surveillance lié à AcrIIA4. (4) Le clivage de l’ADN cible est donc inhibé. (B) Mode d’action d’AcrIIA6. (1’) AcrIIA6 est capable de se dimériser (rouge et bleu) et (2’) de se fixer à deux complexes de surveillance (Cas9 et son ARN guide). (3’) La fixation du dimère d’AcrIIA6 sur la face opposée (orange) du site de liaison à l’ADN (rose) empêche leur dynamique conformationnelle et la reconnaissance de la séquence PAM. (4’) Le complexe de surveillance ne s’étant pas lié à l’ADN cible, celui-ci n’est pas clivé. La structure d’AcrIIA4 a été publiée en 2017 par Shin et al. [5], et celle d’AcrIIA6 par Fuchsbauer et al. en 2017 [4] (figure réalisée avec BioRender). |
Entretien avec Adeline Goulet

Adeline Goulet est chargée de recherche au sein de l’Institut de microbiologie de la Méditerranée (IMM) dans le groupe Interaction Hôte Pathogène. À l’issue de son doctorat en biologie structurale, obtenu en 2009 à l’Université d’Aix-Marseille, elle réalise un stage postdoctoral au Birbeck College de Londres. Elle se spécialise alors en cryomicroscopie électronique. Elle intègre le CNRS en 2015, et se concentre depuis 2017 sur l’étude de l’interaction entre bactéries lactiques et phages, notamment via l’étude de protéines virales inhibant l’immunité bactérienne CRISPR-Cas9. Elle a obtenu la médaille de bronze du CNRS en 2022 pour ses travaux sur l’étude fonctionnelle et structurale des mécanismes mis en place par les phages lors de l’infection de bactéries.
Comment avez-vous découvert la thématique de l’interaction phages-bactéries et celle des protéines anti-CRISPR (Acr) et pour quelles raisons avez-vous décidé de travailler sur ces sujets ?
Pendant ma thèse, j’ai travaillé sur des protéines de virus d’archées extrêmophiles. Il s’agissait de protéines dont on ne connaissait pas les fonctions. J’ai donc un passif interaction phage-bactéries ou phage-archées. C’est quelque chose qui me plaît, mais c’est difficile de définir pourquoi on est attiré vers une thématique plutôt que vers une autre. En ce qui concerne les anti-CRISPR, c’est très fortuit. Un collaborateur, Sylvain Moineau, qui travaille sur les bactériophages depuis très longtemps, nous a contactés en nous disant : “On a des anti-CRISPR, c’est très chaud, est-ce que vous voulez nous rejoindre dans l’aventure ?” C’est ce que j’ai fait.
Votre équipe a découvert la protéine AcrIIA6 en 2018, pouvez-vous nous dire quelles sont les difficultés que vous avez rencontrées ?
C’est en fait Sylvain Moineau qui a découvert ces protéines-là, et nous avons poursuivi l’étude pour caractériser leurs mécanismes d’action au niveau moléculaire. Il y a d’abord une difficulté due à l’ambiance, c’est un milieu très compétitif. À partir du moment où on se lance, c’est beaucoup de pression à porter sur ses épaules. D’un point de vue technique, cette protéine a été très bien produite, il n’y a pas eu de problèmes. Cependant, pour pouvoir étudier sa fonction, il fallait également son partenaire, Cas9, qui s’associe à une molécule d’ARN. Nous avons eu plus de difficultés pour obtenir cette molécule. L’étude s’est divisée en deux temps, d’abord la protéine seule, puis avec les partenaires quand on a pu les obtenir. Pour, AcrIIA5, c’est plus compliqué et, dans la communauté, l’enjeu est important car elle inhibe la majorité des Cas9 utilisés pour l’édition du génome.
La découverte d’AcrIIA6 pourrait fournir de nouvelles pistes pour l’optimisation de l’outil d’édition CRISPR-Cas9, qu’est-ce que vous en pensez ?
D’une manière générale, les protéines anti-CRISPR inhibent l’activité des Cas9 et pourraient être utilisées comme des éléments de régulation des ciseaux moléculaires. Cette thématique a créé un engouement très important et a orienté de nombreuses études sur les molécules anti-interférences qui bloquent Cas9. Quant à AcrIIA6, elle n’est pas la candidate idéale pour réguler les ciseaux moléculaires, car elle est spécifique d’une seule Cas9 au lieu d’avoir une action large spectre comme AcrIIA5. AcrIIA5 cible toutes les Cas9 utilisées dans l’édition du génome et grâce à son renouvellement important, elle représente la candidate favorite. Une autre piste est AcrIIA4, dont il a été montré qu’elle était capable de réguler l’édition du génome par Cas9 et de limiter les clivages non spécifiques.
L’existence d’une forme tronquée d’AcrIIA6 a été mise en avant ainsi que sa potentielle utilité pour limiter l’utilisation d’antibiotiques. Pensez-vous que cette découverte pourrait être exploitée dans un avenir proche dans les laboratoires et seriez-vous prête à utiliser ce type de technique ?
Effectivement, l’Acr tronquée n’a plus d’activité d’interférence, elle n’est donc pas candidate pour inhiber les ciseaux moléculaires. Cependant, elle a le potentiel d’augmenter la persistance de plasmides en l’absence d’antibiotiques ce qui pourrait être intéressant pour certaines applications biotechnologiques. Il est difficile de dire si son utilisation va être développée rapidement. Personnellement, je serais prête à l’utiliser, par contre, il faut travailler avec une souche ayant un système CRISPR-Cas actif.
Selon vous, est-il important de continuer à étudier les bactériophages ? Que pourraient-ils encore nous apprendre ?
Je pense qu’il est important de continuer à étudier les bactériophages puisque leur découverte est assez récente (un tout petit siècle !). Ils ont été à la base de découvertes majeures en biologie, comme la découverte du matériel génétique. On pourrait penser qu’on en a fait le tour, mais ce n’est pas le cas, parce qu’un des challenges actuels de la recherche sur les bactériophages est de comprendre la fonction de ce qu’on appelle la « viral dark matter ». Les génomes des bactériophages sont très compacts ; tout ce qui s’y trouve est utile, sinon le phage s’en débarrasse. Pour certains des phages, il peut y avoir jusqu’à 75 % de gènes codant des protéines dont la fonction est inconnue. Cependant elles sont là, donc elles sont utiles. C’est donc un réservoir très important de nouvelles découvertes possibles et d’applications biotechnologiques éventuelles.
L’édition du génome humain soulève des questions éthiques et reste interdite dans la plupart des pays. Avez-vous des réserves sur l’utilisation de l’outil CRISPR-Cas9 ?
Je ne suis pas vraiment spécialiste du domaine, mais ce qui est sûr c’est qu’il y a ce qu’on appelle les effets hors-cibles. La Cas9 est très spécifique au début de l’activité des ciseaux moléculaires, qui vont aller cliver la cible d’intérêt. Mais du fait de la persistance au cours du temps de Cas9-ARN dans la cellule ou dans le tissu, in fine, inévitablement de l’ADN non ciblé pourrait être potentiellement clivé, d’où l’intérêt des Acr. Mais, pour l’instant, ce problème n’est pas résolu, ce qui limite leur utilisation en santé humaine.
Quels sont vos projets pour l’avenir à moyen terme dans vos recherches scientifiques ? Et est-ce que les Acr suscitent toujours votre intérêt ?
Les Acr suscitent toujours mon intérêt. Il y a d’abord eu la caractérisation d’AcrIIA6 puis de la forme tronquée d’AcrIIA6. Aujourd’hui nous travaillons activement pour essayer de comprendre son mécanisme d’action. Nous savons qu’elle inhibe la première étape d’adaptation, ce qui est très intéressant car cette étape est également l’étape de l’immunité CRISPR-Cas qui est la moins connue. Ainsi, comprendre comment fonctionne AcrIIA6 tronquée permettra aussi de comprendre comment fonctionne l’étape d’adaptation. C’est donc quelque chose qui m’intéresse actuellement et plusieurs demandes de financement sont en cours. À moyen terme, c’est cette Acr qui retient mon attention, mais après, qui sait ? Dans le monde des Acr, il peut encore apparaitre d’autres choses qui m’intéresseront à l’avenir. Je prévois aussi d’essayer de développer un peu plus activement l’analyse structurale et fonctionnelle de la « viral dark matter ».
Que pensez-vous de la place des femmes dans le milieu de la recherche et plus particulièrement dans le milieu très masculin qu’est la biologie structurale ?
La place de la femme est assez compliquée ; disons que je ne pensais pas vraiment le vivre un jour, mais il est vrai que quand on est une femme, notre parole n’a pas le même poids qu’un homme à responsabilités égales. Il m’est déjà arrivé qu’on ne réponde pas directement à mes mails, mais qu’on s’adresse à quelqu’un qui est à la retraite, mais qui est masculin ; il faut savoir gérer. Au début c’est très bizarre, il faut encaisser ! Mais, ensuite, il faut vivre avec et avancer. Mais c’est vrai, ça existe, et je pense que ça existe dans n’importe quel milieu professionnel.
Est-ce qu’un résultat ou un moment de votre carrière vous a particulièrement marquée ou rendue fière ?
Il n’y a pas vraiment un moment en particulier. Je pense que la recherche scientifique est tellement personnelle, on y met tellement de soi que les joies comme les peines se vivent à cent pour cent. Je n’ai donc pas un moment particulier. C’est vraiment plusieurs moments au cours de ma carrière. Après, il y a juste une petite histoire que je trouve amusante : comme je vous le disais, pendant ma thèse, je travaillais sur des protéines de fonctions inconnues. On a résolu la structure de ces protéines, et, pour certaines d’entre elles, on ne savait pas trop à quoi elles servaient. On a fait des hypothèses, on a publié nos résultats. Et quand j’ai commencé à travailler sur les Acr, la première Acr inhibant CRISPR-Cas9 chez les archées a été découverte. Il se trouve que cette Acr était la protéine sur laquelle j’avais travaillé, c’était donc une jolie coïncidence.
L’optimisation de l’outil d’édition CRISPR, un enjeu thérapeutique
L’utilisation de la technologie CRISPR est aujourd’hui très répandue dans le domaine de la recherche. En effet, cette technique d’édition du génome très efficace a permis à des équipes de recherche travaillant sur des microorganismes à croissance lente (Mycobactéries, Myxococcus), sur des cellules eucaryotes ou encore sur des organismes entiers, de réaliser des mutations rapidement. En effet, des modifications génétiques chez les organismes à croissance lente peuvent désormais se faire en quelques jours, voire semaines, alors qu’il fallait compter plusieurs mois avant cette découverte. Quant aux organismes entiers, tels que les souris, la suppression d’un gène pouvait prendre plusieurs années pour que la mutation soit stabilisée et transmise à la descendance. Cette technique peut également être appliquée à des stratégies de thérapie génique. Dans ce contexte, des résultats pré-cliniques prometteurs ont déjà été obtenus pour le traitement de la tyrosinémie héréditaire dans des modèles de souris [6], ou pour modifier in vitro les récepteurs de certains virus et bloquer leur infectiosité [7, 8]. Récemment, cette technique a été utilisée chez l’homme dans un essai clinique visant à traiter la drépanocytose et la béta thalassémie (des maladies du sang). Un autre essai clinique prometteur, en cours de phase I, évalue la mise au point d’une stratégie thérapeutique pour lutter contre l’amaurose congénitale de Leber. Cette maladie est caractérisée par de graves malformations cardiaques résultant de l’accumulation de fibrilles amyloïdes. La mutation du gène TTR (transthyretin) qui code la transthyrétine, une protéine plasmatique, est à l’origine de l’accumulation de ces fibrilles. L’objectif de cet essai clinique fondé sur l’utilisation du système CRISPR-Cas9 est donc d’inactiver le gène muté, dans ce cas TTR, empêchant ainsi la formation de fibrilles amyloïdes [9] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 10, octobre 2021, page 933
Néanmoins, lorsqu’il s’agit de modifier des cellules germinales, l’utilisation de cette technique ne peut s’effectuer que dans le cadre des laboratoires de recherche fondamentale. Un autre frein à l’utilisation de cette technique en médecine est le clivage non spécifique du génome par Cas9. Lorsque le système CRISPR est intégré dans une cellule, soit directement dans la cellule soit via un plasmide codant Cas9 et l’ARN guide, il peut se produire un clivage de l’ADN chromosomique de la cellule hôte en dehors des séquences cibles de l’ARN guide. La découverte d’AcrIIA6 ouvre de nouvelles pistes pour solutionner ce problème. Sa capacité de dimérisation lui permettant, à la différence d’AcrIIA4, d’inhiber deux Cas9 bactériennes la rend très efficace. L’utilisation d’AcrIIA6, une fois l’édition du génome par l’outil CRISPR-Cas9 terminée, pourrait permettre d’éviter la dégradation de l’ADN hôte en inactivant la Cas9, empêchant ainsi les clivages hors cibles. Par ailleurs, une forme tronquée d’AcrIIA6 a été récemment découverte [10]. Cette dernière empêche les bactéries d’acquérir un espaceur, mais n’est plus capable d’inactiver la protéine Cas9. Une étude récente a montré que la présence du gène codant cette forme tronquée d’AcrIIA6 sur un plasmide, favoriserait son maintien dans une bactérie, sans avoir recours à l’utilisation de cassettes de résistance à un antibiotique. Ces derniers exemples sont l’une des multiples facettes des applications des technologies CRISPR et démontrent une fois encore la diversité et la richesse des phages. De nombreuses protéines Acr continuent d’être découvertes et ouvrent la voie vers le développement et l’amélioration de nouveaux outils dédiés à l’édition du génome.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Jinek M, Chylinski K, Fonfara I, et al. A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science 2012 ; 337 : 816–821. [CrossRef] [PubMed] [Google Scholar]
- Bondy-Denomy J, Pawluk A, Maxwell KL, Davidson AR. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system. Nature 2013 ; 493 : 429–432. [CrossRef] [PubMed] [Google Scholar]
- Hynes AP, Rousseau GM, Agudelo D, et al. Widespread anti-CRISPR proteins in virulent bacteriophages inhibit a range of Cas9 proteins. Nat Commun 2018 ; 9 : 2919. [CrossRef] [PubMed] [Google Scholar]
- Fuchsbauer O, Swuec P, Zimberger C, et al. Cas9 Allosteric Inhibition by the Anti-CRISPR Protein AcrIIA6. Molecular Cell 2019 ; 76 : 922–37 e7. [CrossRef] [PubMed] [Google Scholar]
- Shin J, Jiang F, Liu JJ, et al. Disabling Cas9 by an anti-CRISPR DNA mimic. Sci Adv 2017 ; 3 : e1701620. [CrossRef] [PubMed] [Google Scholar]
- Yin H, Xue W, Chen S, et al. Genome editing with Cas9 in aduLT mice corrects a disease mutation and phenotype. Nat Biotechnol 2014 ; 32 : 551–553. [CrossRef] [PubMed] [Google Scholar]
- Zhen S, Hua L, Liu YH, et al. Harnessing the clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated Cas9 system to disrupt the hepatitis B virus. Gene Ther 2015 ; 22 : 404–412. [CrossRef] [PubMed] [Google Scholar]
- Ye L, Wang J, Beyer AI, et al. Seamless modification of wild-type induced pluripotent stem cells to the natural CCR5Δ32 mutation confers resistance to HIV infection. Proc Natl Acad Sci USA 2014 ; 111 : 9591–9596. [CrossRef] [PubMed] [Google Scholar]
- Jordan, B. Édition de gènes in vivo et thérapie génique. Chroniques génomiques. Med Sci (Paris) 2021; 37 : 933–5. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Philippe C, Morency C, Plante PL, et al. A truncated anti-CRISPR protein prevents spacer acquisition but not interference. Nat Commun 2022; 13 : 2802. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Le « système immunitaire » bactérien CRISPR-Cas9 et l’édition du génome humain. 1. Schématisation du fonctionnement du système CRISPR-Cas9 bactérien : (a) Incorporation de l’ADN viral au sein du chromosome bactérien ; (b) L’enzyme Cas9 forme un complexe avec l’ARN issu de la transcription de l’espaceur (crARN) et un ARN transactivateur (tracrARN) ; (c) L’enzyme Cas9 clive l’ADN viral, bloquant alors sa réplication. (d) Le même phage infecte à nouveau une bactérie immunisée. 2. Utilisation de CRISPRCas9 pour la construction d’un outil d’édition du génome humain. Un clivage hors cible constitue une limitation à l’application thérapeutique de la technique d’édition. 3. Les protéines anti- CRISPR (Acr), protéines phagiques induisant l’inhibition du système immunitaire CRISPRCas9, peuvent agir par différents mécanismes qui empêchent le complexe de se lier à l’ADN cible (figure réalisée avec BioRender). |
| Dans le texte | |
 |
Figure 2. Comparaison des modes d’action d’AcrIIA4 et d’AcrIIA6. (A) Mode d’action d’AcrIIA4. (1) Formation du complexe de surveillance avec Cas9 (gris) et l’ARN guide (violet). (2) AcrIIA4 (vert) se fixe sur le site de liaison au PAM (rose) responsable de la reconnaissance de l’ADN cible. (3) La séquence PAM de l’ADN cible ne peut être reconnue par le complexe de surveillance lié à AcrIIA4. (4) Le clivage de l’ADN cible est donc inhibé. (B) Mode d’action d’AcrIIA6. (1’) AcrIIA6 est capable de se dimériser (rouge et bleu) et (2’) de se fixer à deux complexes de surveillance (Cas9 et son ARN guide). (3’) La fixation du dimère d’AcrIIA6 sur la face opposée (orange) du site de liaison à l’ADN (rose) empêche leur dynamique conformationnelle et la reconnaissance de la séquence PAM. (4’) Le complexe de surveillance ne s’étant pas lié à l’ADN cible, celui-ci n’est pas clivé. La structure d’AcrIIA4 a été publiée en 2017 par Shin et al. [5], et celle d’AcrIIA6 par Fuchsbauer et al. en 2017 [4] (figure réalisée avec BioRender). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




