Figure 2.
Télécharger l'image originale
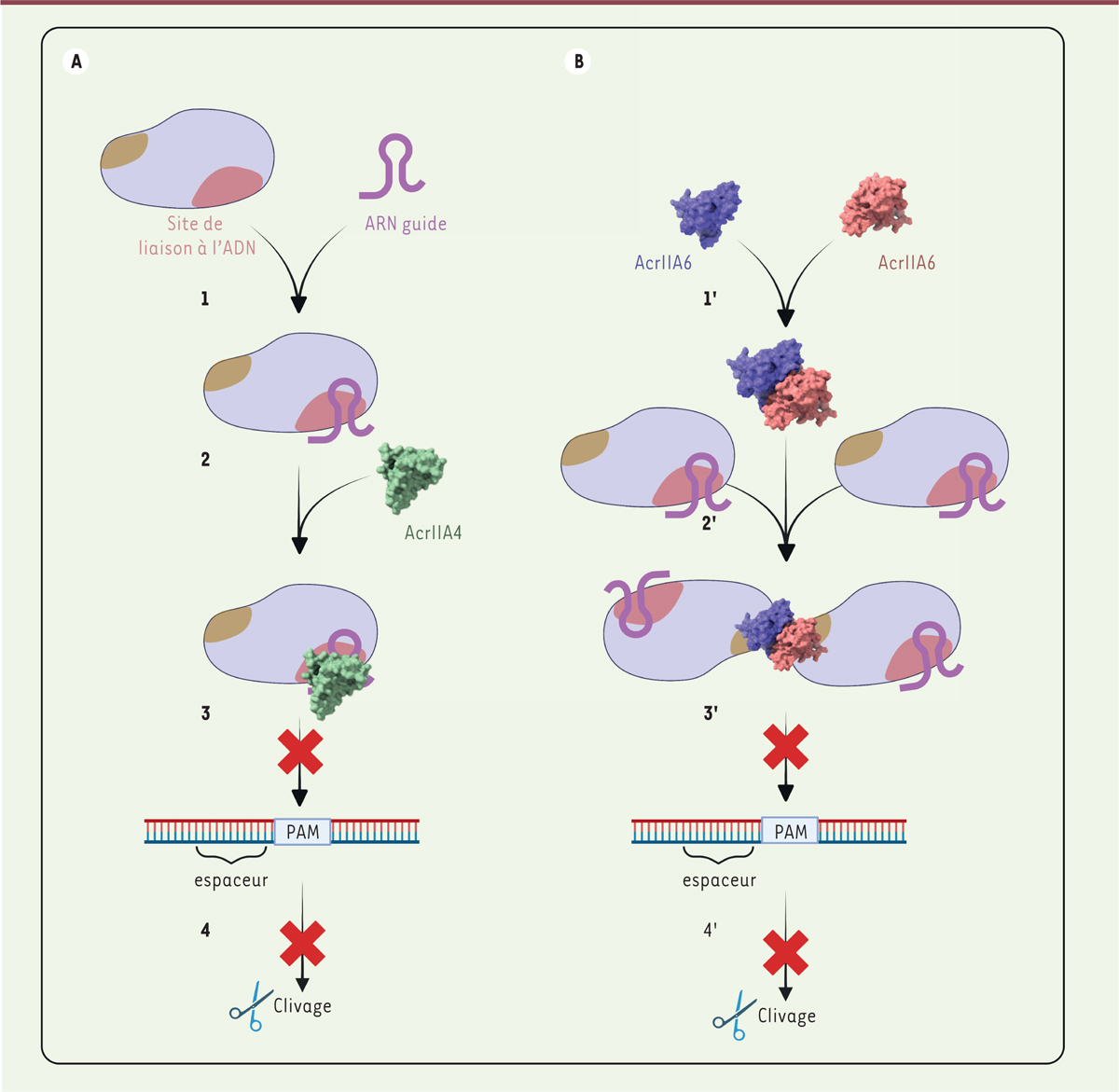
Comparaison des modes d’action d’AcrIIA4 et d’AcrIIA6. (A) Mode d’action d’AcrIIA4. (1) Formation du complexe de surveillance avec Cas9 (gris) et l’ARN guide (violet). (2) AcrIIA4 (vert) se fixe sur le site de liaison au PAM (rose) responsable de la reconnaissance de l’ADN cible. (3) La séquence PAM de l’ADN cible ne peut être reconnue par le complexe de surveillance lié à AcrIIA4. (4) Le clivage de l’ADN cible est donc inhibé. (B) Mode d’action d’AcrIIA6. (1’) AcrIIA6 est capable de se dimériser (rouge et bleu) et (2’) de se fixer à deux complexes de surveillance (Cas9 et son ARN guide). (3’) La fixation du dimère d’AcrIIA6 sur la face opposée (orange) du site de liaison à l’ADN (rose) empêche leur dynamique conformationnelle et la reconnaissance de la séquence PAM. (4’) Le complexe de surveillance ne s’étant pas lié à l’ADN cible, celui-ci n’est pas clivé. La structure d’AcrIIA4 a été publiée en 2017 par Shin et al. [5], et celle d’AcrIIA6 par Fuchsbauer et al. en 2017 [4] (figure réalisée avec BioRender).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




