| Issue |
Med Sci (Paris)
Volume 39, Number 8-9, Août–Septembre 2023
Nos jeunes pousses ont du talent !
|
|
|---|---|---|
| Page(s) | 676 - 680 | |
| Section | Partenariat médecine/sciences - Écoles doctorales - Masters | |
| DOI | https://doi.org/10.1051/medsci/2023104 | |
| Published online | 11 September 2023 | |
Les métallophores
La ruée vers les métaux
Metallophores: The metal rush
1
Master 2 Microbiologie intégrative et fondamentale, Aix Marseille université, Marseille, France
2
Aix Marseille université, CNRS, LCR UMR7283, IMM, Marseille, France
a laura.bonhomme@etu.univ-amu.fr
b ludovic.dubard@etu.univ-amu.fr
c melvin.riha@etu.univ-amu.fr
d anthony.rodrigues@etu.univ-amu.fr
e aussel@imm.cnrs.fr
Les métaux sont des éléments essentiels au métabolisme et à la survie de tous les êtres vivants. Ces éléments jouent un rôle structural et catalytique en tant que cofacteurs dans des processus biologiques, tels que la signalisation ou la biosynthèse de précurseurs. De plus, de nombreuses protéines possèdent des cofacteurs métalliques indispensables à leur fonction biologique. Comprendre les voies de signalisation et connaître les acteurs impliqués dans l’homéostasie des métaux sont donc un enjeu majeur.
Certains organismes ont acquis, au cours de l’évolution, des mécanismes moléculaires permettant de piéger et d’importer des métaux, leur conférant ainsi un avantage sélectif dans les milieux carencés. C’est le cas des plantes, chez qui des transporteurs spécifiques (les métallophores) ont été identifiés. À l’instar des mineurs cherchant des métaux rares, les métallophores permettent la récupération de ces éléments essentiels. Chez les plantes comme chez les bactéries, une famille d’enzymes permet la biosynthèse de ces métallophores : les nicotianamine synthases.
Dans les écosystèmes où différentes espèces cohabitent, certaines bactéries vont sécréter ces chélateurs, puis les importer complexés à des métaux [1]. D’autres bactéries, qualifiées d’opportunistes [2], vont pouvoir capter dans l’environnement des métallophores sans avoir eu besoin de les synthétiser au préalable. Le rôle de ces métallophores est crucial lors du processus d’infection, au cours duquel les cellules hôtes de l’organisme infecté vont mettre en place une « immunité nutritionnelle » visant à séquestrer les métaux, les rendant indisponibles aux bactéries et freinant leur prolifération [3]. Pour survivre et continuer à se développer au sein de l’organisme infecté, certains pathogènes ont acquis la capacité de sécréter et d’importer des métallophores : la staphylopine chez Staphyloccocus aureus [4, 5], la pseudopaline chez Pseudomonas aeruginosa [5, 6], et la yersinopine chez Yersinia pestis [7]. Cette identification a été possible grâce à la présence d’un ensemble de gènes (opérons) conservés chez ces espèces. La présente nouvelle synthétise des travaux récents portant sur l’identification de différentes voies de biosynthèse de métallophores chez les bactéries. Elle décrit également une méthode innovante menant à l’identification de ces molécules.
La biosynthèse des métallophores
La technique d’ICP-MS (spectrométrie de masse à plasma à couplage inductif) a permis, dans plusieurs études, de mieux comprendre comment étaient synthétisés les métallophores [4, 6]. Cette méthode, qui consiste à ioniser un échantillon biologique au moyen d’une torche à plasma, puis à analyser le plasma par spectrométrie de masse, permet de séparer et de quantifier de manière extrêmement précise les ions métalliques contenus dans le cytoplasme bactérien. En appliquant cette technique sur des souches sauvages et des souches mutantes comportant des délétions, il a été possible d’identifier plusieurs gènes impliqués dans la voie de biosynthèse de certains métallophores [4,6]. Ainsi, la voie de biosynthèse de la staphylopine, premier métallophore identifié chez S. aureus, est dépendante d’un ensemble de trois gènes (cntK, cntL, cntM) organisés en structure opéronique1 [4]. Le gène cntK code la racémase CntK, une enzyme permettant l’inversion de la structure spatiale d’un acide aminé. Le substrat de cette enzyme est l’histidine, un chélateur connu du nickel grâce à son cycle imidazole. La protéine CntL est une enzyme permettant la formation d’un intermédiaire réactionnel à partir de la S-adénosylméthionine et de la D-histidine. Enfin, CntM est une opine déshydrogénase terminant la voie de biosynthèse du métallophore en réduisant l’intermédiaire réactionnel associé à du pyruvate avec un cosubstrat : le NADPH (Figure 1) [4]. La pseudopaline est un autre métallophore [6]. Produite par P. aeruginosa, elle est spécialisée dans l’import du nickel ainsi que dans celui du zinc. Sa biosynthèse diffère de celle de la staphylopine par l’utilisation de la L-histidine, ne nécessitant donc pas d’inversion stéréochimique catalysée par CntK (dont la séquence codante est absente dans l’opéron cnt chez cette bactérie). De plus, CntM ne synthétise pas de pseudopaline à partir de pyruvate et de NADPH mais à partir d’a-cétoglutarate et de NADH (Figure 1) [6]. Le large spectre de métaux chélatés par la staphylopine et la pseudopaline confère un avantage sélectif aux bactéries qui les produisent. En effet, la capacité des micro-organismes à importer ces éléments est proportionnelle à leur aptitude à proliférer dans des milieux carencés.
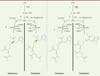 |
Figure 1. Voies de biosynthèse des métallophores chez différents microorganismes. 1. Conversion de la L-histidine en D-histidine par la racémase CntK chez Staphylococcus aureus et Paenibacillus mucilaginosus. 2. Catalyse de la réaction par CntL du S-adénosylméthionine (SAM) sur la D-histidine ou L-histidine pour produire un intermédiaire réactionnel : xNA ou yNA respectivement. 3. Condensation de xNA/yNA avec soit le pyruvate ou l’α-cétoglutarate en utilisant le NAD(P)H et un H+ pour former le métallophore. Cette réaction se fait par l’intermédiaire de CntM, une opine déshydrogénase (figure réalisée avec BioRender). |
Régulation et transport des métallophores
La teneur en métaux pouvant fortement varier d’un environnement à l’autre, la régulation de la production et le transport des métallophores vont avoir un rôle clé dans le contrôle de l’homéostasie cellulaire. En effet, le fer et le zinc figurent parmi les ions métalliques les plus importants pour la cellule. De nombreuses réactions au sein de celle-ci impliquent des cofacteurs métalliques comme les centres fer-soufre (pour le transfert d’électrons lors de réactions d’oxydo-réduction) ou encore les motifs en doigts de zinc (impliqués dans les interactions avec l’ADN).
Comme la biosynthèse de métallophores est un mécanisme nécessitant une importante dépense d’énergie pour la bactérie, leur production est activée seulement lors d’une carence en métaux. Les gènes codant la staphylopine et la pseudopaline sont tous deux sous le contrôle de Zur [8], un répresseur dépendant de la concentration intracellulaire en zinc. Lorsque la quantité de zinc dans l’environnement est élevée, Zur réprime l’expression des gènes codant la biosynthèse et le transport de ces métallophores. À l’inverse, dans des conditions limitantes en zinc, ce répresseur n’exerce plus son activité, ce qui permet de maintenir l’homéostasie du métal. P. aeruginosa possède également un régulateur spécifique du fer ayant un fonctionnement similaire : Fur [8]. Par ailleurs, la sécrétion des métallophores requiert la présence de transporteurs spécifiques tels que CntE ou CntI exportant respectivement la staphylopine et la pseudopaline [9]. Ces derniers « véhiculent » le métallophore en une ou deux étapes à travers la membrane. Une fois le complexe métal-métallophore formé, celui-ci réintègre la cellule en passant par différents transporteurs spécifiques qui sont codés par d’autres gènes de l’opéron comme cntABCDF chez S. aureus ou cntO chez P. aeruginosa (Figure 2). Une fois dans le compartiment intracellulaire bactérien, le métal séquestré va pouvoir participer à divers processus cellulaires. Les bactéries sécrètent des métallophores dont les affinités varient selon les métaux. Chez S. aureus la hiérarchie de ces constantes d’affinité est la même que celle retrouvée chez les plantes (Cu2+ > Ni2+ > Co2+ > Zn2+ > Fe2+), indiquant un possible lien évolutif entre les deux organismes [4]. La relation entre la structure des métallophores et leur affinité pour les différents métaux n’est, en revanche, pas encore élucidée (Figure 2) [1].
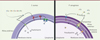 |
Figure 2. Trafic des métallophores chez Staphyloccocus aureus et Pseudomonas aeruginosa. Chez S. aureus, CntE (notée E) permet l’export de la staphylopine dans le milieu extracellulaire. Ce dernier peut se complexer à un métal, puis être importé de nouveau dans la cellule par un ABC transporteur formé par les protéines CntA, B, C, D et F (notées A, B, C, D et F). Chez P. aeruginosa, la protéine CntI (notée I) permet l’export de la pseudopaline dans le périplasme où elle va être prise en charge par un transporteur jusqu’au milieu extracellulaire. Cette dernière peut se complexer à un métal, puis être importée de nouveau dans le périplasme par la protéine CntO (notée O). Le devenir du complexe pseudopaline-métal n’est pas encore connu (figure réalisée avec BioRender). |
De la prédiction vers l’identification de nouveaux métallophores
L’équipe du Dr Pascal Arnoux a récemment développé une méthode bioinformatique afin d’identifier de nouvelles molécules de type nicotianamine. Cette approche repose sur la recherche, au sein des génomes bactériens, de gènes codant les enzymes impliquées dans ces voies de biosynthèse. Ainsi, deux règles régissent la production des métallophores [7] :
1. La présence ou non du gène cntK dans l’opéron cnt (impactant l’orientation dans l’espace de l’histidine du métallophore, Figure 1).
2. La présence d’un acide aspartique ou d’une alanine à la position 150 dans la séquence protéique de l’enzyme CntM. Le choix de l’un ou de l’autre de ces deux acides aminés va orienter la voie de biosynthèse vers l’utilisation de pyruvate (acide aspartique) ou d’a-cétoglutarate (alanine) dans la réaction catalysée par CntM (Figure 1). Ce biais dans l’utilisation des substrats semble in fine orienter l’affinité du métallophore pour les différents métaux.
Sur les quatre métallophores de type nicotianamine produits par la voie de biosynthèse de l’opéron cnt, trois ont été identifiés et caractérisés par des approches biochimiques : la pseudopaline, la staphylopine et la yersinopine (Figure 1). Grâce à l’approche bioinformatique précédemment décrite, il a été possible de prédire la biosynthèse d’un quatrième métallophore : la bacilopaline [7], retrouvé chez la bactérie Paenibacillus mucilaginosus. D’ordinaire, l’étude d’une molécule découle de sa découverte. Dans le cas présent, l’équipe du Dr Pascal Arnoux nous montre que le cheminement inverse est possible : en recherchant les gènes codant les enzymes requises pour sa biosynthèse, l’existence d’un métallophore peut être prédite avant que celui-ci ne soit découvert.
Cette nouvelle met en lumière les réactions enzymatiques menant à la biosynthèse des métallophores de type nicotianamine chez plusieurs bactéries pathogènes ainsi que la conservation inter-espèces de ces voies anaboliques. Elle recense également certaines variations, notamment au niveau de l’opéron cnt au sein duquel figurent des gènes codant des enzymes impliquées dans la formation, la régulation et le transport de ces métallophores. La combinaison de l’ensemble de ces résultats est une étape vers la compréhension d’un mécanisme adaptatif chez certaines bactéries pathogènes dans des milieux carencés en métaux. Ainsi, des recherches plus approfondies sur l’affinité des métallophores pour leurs substrats pourraient apporter de nouvelles perspectives quant à leur rôle dans l’homéostasie métallique des bactéries. Des molécules chélatrices de métaux comme l’holomycine [10] ou d’autres visant des enzymes de la voie de biosynthèse des métallophores pourraient ainsi constituer de nouvelles cibles thérapeutiques dans le futur.
Entretien avec Pascal Arnoux

Pascal Arnoux est directeur de recherche au Commissariat à l’Énergie Atomique de Cadarache. Il est responsable de l’équipe MEM (Microbiologie Environnementale et Moléculaire) à l’institut de Recherche en biosciences et biotechnologies d’Aix-Marseille (BIAM).
Il étudie la microbiologie ainsi que la biologie structurale d’enzymes et de métalloenzymes. Au fil des années, il est devenu expert dans l’étude des mécanismes de transport des métaux, un processus essentiel chez les bactéries.
Au-delà des bactéries pathogènes et des plantes, quelles autres espèces sont pourvues de métallophores de type nicotianamine ?
Principalement des eucaryotes comme des plantes ou d’autres types de bactéries, mais il y a également des champignons.
Les variations de la structure des métallophores sont-elles dues aux différentes concentrations en métaux des niches écologiques ?
Je ne dirais pas cela. En revanche, il y a une compétition entre les micro-organismes pour l’accès à des ressources métalliques. Chaque bactérie va avoir un intérêt à développer son propre métallophore car cela lui confère un avantage sélectif vis-à-vis des autres.
Quel est le lien entre la structure d’un métallophore et son affinité pour un métal ?
Ce n’est pas une question simple. L’affinité va dépendre de beaucoup de paramètres de stabilisation du complexe avec le métal. Il vaut mieux prendre l’approche expérimentale pour déterminer l’affinité.
Est-ce une bonne idée de faire le même travail que vous avez fait avec CntM mais sur CntL ?
Pour CntL se pose une question qui n’a toujours pas de réponse : quelle est la stéréospécificité de cette enzyme ? L’approche structurale est une très bonne approche pour essayer de comprendre pourquoi certaines CntL sont spécifiques de la L-histidine et d’autres de la D-histidine. À l’heure actuelle ce n’est toujours pas connu.
Pensez-vous qu’il existe un lien direct entre l’import des métaux et la virulence ?
Dans certains cas, oui, il existe des métalloprotéases qui sont des facteurs de virulence et qui sont dépendantes de certains métaux. L’accès à ces ressources métalliques est en lien avec la virulence. Une bactérie ne pouvant pas y accéder ne va pas pouvoir survivre dans son environnement comme chez l’hôte et ne sera donc plus virulente.
Au vu des différents résultats présentés dans la nouvelle, que verriez-vous comme application à l’étude des métallophores ?
Il y a une application qui est dans l’air du temps : la stratégie du Cheval de Troie. Elle vise à attacher un antibiotique à ce type de métallophore et notamment les sidérophores, spécifiques de l’import du fer. Comme ces métallophores sont importés de manière active, l’antibiotique va être également importé de la même manière, s’accumuler dans la bactérie et augmenter sa toxicité.
Qu’est-ce qui vous a amené à l’étude des métalloprotéines ?
Initialement, c’est un facteur chance. J’ai commencé en thèse à travailler sur un hémophore : HasA de Serratia marcescens. Il se trouve que par la suite j’ai toujours gardé un intérêt pour cette thématique et j’ai la chance de pouvoir la poursuivre.
Votre équipe regroupe de nombreuses compétences scientifiques. Quels ont été leurs apports dans votre projet de recherche ?
C’est une chance de pouvoir travailler avec des gens qui proviennent de différents horizons. Il est vrai qu’il est parfois compliqué de discuter avec des chimistes ou des physiciens mais c’est une réelle opportunité qu’il faut savoir cultiver.
Vous avez une expertise poussée dans le domaine de la biologie structurale. Que pensez-vous de la proportion de plus en plus importante de la bioinformatique dans votre domaine à l’image d’Alphafold ?
C’est une bonne question mais qui est compliquée et sur laquelle il faut se garder d’avoir des idées figées car on n’a pas encore le recul nécessaire pour y répondre. Il est sûr que ces approches de bioinformatique prennent de plus en plus d’importance.
Mis à part les métallophores, travaillez-vous sur d’autres projets ?
J’ai notamment contribué à l’étude d’une enzyme qui marche à la lumière et qui produit des alcanes à partir d’acides gras. J’ai d’autres sujets un peu annexes mais qui restent axés sur la biologie structurale.
Si vous aviez un conseil à donner à un jeune chercheur découvrant le milieu, quel serait-il ?
C’est toujours compliqué pour moi de donner des conseils. Je dirais peut-être : poursuivez ce qui vous fait plaisir et qui fait briller vos yeux.
Quel est le meilleur souvenir de votre carrière ?
En thèse, j’ai eu la chance de résoudre une structure qui ressemblait à un poisson et que l’on a comparé à une rascasse. Il y a un côté que j’imagine très plaisant dans la recherche qui est de découvrir en avant-première des choses que personne ne sait.
Références
- Andreini C, Bertini I, Cavallaro G, et al. Metal ions in biological catalysis: from enzyme databases to general principles. J Biol Inorg Chem 2008 ; 13 : 1205–1218. [CrossRef] [PubMed] [Google Scholar]
- Butaite˙ E, Baumgartner M, Wyder S, et al. Siderophore cheating and cheating resistance shape competition for iron in soil and freshwater Pseudomonas communities. Nat Commun 2017; 8 : 414. [CrossRef] [PubMed] [Google Scholar]
- Grim KP, San Francisco B, Radin JN, et al. The metallophore staphylopine enables Staphylococcus aureus to compete with the host for zinc and overcome nutritional immunity. mBio 2017; 8 : e01281–17. [PubMed] [Google Scholar]
- Ghssein G, Brutesco C, Ouerdane L, et al. Biosynthesis of a broad-spectrum nicotianamine-like metallophore in Staphylococcus aureus. Science 2016 ; 352 : 1105–1109. [CrossRef] [PubMed] [Google Scholar]
- Laffont C, Arnoux P. The ancient roots of nicotianamine: diversity, role, regulation and evolution of nicotianamine-like metallophores. Metallomics 2020; 12 : 1480–93. [CrossRef] [PubMed] [Google Scholar]
- Lhospice S, Gomez NO, Ouerdane L, et al. Pseudomonas aeruginosa zinc uptake in chelating environment is primarily mediated by the metallophore pseudopaline. Sci Rep 2017 ; 7 : 17132. [CrossRef] [PubMed] [Google Scholar]
- Laffont C, Brutesco C, Hajjar C, et al. Simple rules govern the diversity of bacterial nicotianamine-like metallophores. Biochem J 2019 ; 476 : 2221–2233. [CrossRef] [PubMed] [Google Scholar]
- Fojcik C, Arnoux P, Ouerdane L, et al. Independent and cooperative regulation of staphylopine biosynthesis and trafficking by Fur and Zur. Mol Microbiol 2018 ; 108 : 159–177. [CrossRef] [PubMed] [Google Scholar]
- Remy L, Carrière M, Derré-Bobillot A, et al. The Staphylococcus aureus Opp1 ABC transporter imports nickel and cobaLT in zinc-depleted conditions and contributes to virulence. Mol Microbiol 2013 ; 87 : 730–743. [CrossRef] [PubMed] [Google Scholar]
- Chan AN, Shiver AL, Wever WJ, et al. Role for dithiolopyrrolones in disrupting bacterial metal homeostasis. Proc Natl Acad Sci USA 2017 ; 114 : 2717–2722. [CrossRef] [PubMed] [Google Scholar]
© 2023 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Voies de biosynthèse des métallophores chez différents microorganismes. 1. Conversion de la L-histidine en D-histidine par la racémase CntK chez Staphylococcus aureus et Paenibacillus mucilaginosus. 2. Catalyse de la réaction par CntL du S-adénosylméthionine (SAM) sur la D-histidine ou L-histidine pour produire un intermédiaire réactionnel : xNA ou yNA respectivement. 3. Condensation de xNA/yNA avec soit le pyruvate ou l’α-cétoglutarate en utilisant le NAD(P)H et un H+ pour former le métallophore. Cette réaction se fait par l’intermédiaire de CntM, une opine déshydrogénase (figure réalisée avec BioRender). |
| Dans le texte | |
 |
Figure 2. Trafic des métallophores chez Staphyloccocus aureus et Pseudomonas aeruginosa. Chez S. aureus, CntE (notée E) permet l’export de la staphylopine dans le milieu extracellulaire. Ce dernier peut se complexer à un métal, puis être importé de nouveau dans la cellule par un ABC transporteur formé par les protéines CntA, B, C, D et F (notées A, B, C, D et F). Chez P. aeruginosa, la protéine CntI (notée I) permet l’export de la pseudopaline dans le périplasme où elle va être prise en charge par un transporteur jusqu’au milieu extracellulaire. Cette dernière peut se complexer à un métal, puis être importée de nouveau dans le périplasme par la protéine CntO (notée O). Le devenir du complexe pseudopaline-métal n’est pas encore connu (figure réalisée avec BioRender). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




