| Issue |
Med Sci (Paris)
Volume 32, Number 12, Décembre 2016
|
|
|---|---|---|
| Page(s) | 1079 - 1086 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20163212011 | |
| Published online | 03 January 2017 | |
Immunité innée et maladies chez l’homme
De l’introgression archaïque à la sélection naturelle
Innate immunity and human diseases: from archaic introgression to natural selection
Institut Pasteur, Unité de Génétique Évolutive humaine, Département Génomes et Génétique, 25, rue du Docteur Roux, 75015 Paris, France ; Centre national de la recherche scientifique, URA3012, 75015 Paris, France ; Université Pierre et Marie Curie, cellule Pasteur UPMC, 25, rue du Docteur Roux, 75015 Paris, France
Les pressions de sélection exercées par les pathogènes ont participé à façonner la variabilité du génome humain au cours de notre évolution. Les approches de génétique évolutive et de génétique des populations permettent d’étudier la forme et l’intensité de ces forces évolutives. Ces études ont mis en évidence l’importance fonctionnelle de certains gènes, l’implication d’autres dans l’adaptation de l’homme à son environnement pathogénique, ainsi que le maintien d’allèles archaïques supposés avantageux pour notre espèce. Cependant, avec l’avènement de traitements préventifs et curatifs, la sélection exercée par les pathogènes s’est récemment relâchée et des allèles ayant conféré un avantage à nos ancêtres se révèlent parfois être impliqués dans des troubles du système immunitaire, comme l’auto-immunité et l’inflammation.
Abstract
Throughout evolution, humans have had to face strong variation in environmental conditions, with pathogens being among the strongest threats that our species has encountered. The use of population genetic approaches provides novel insights into how natural selection imposed by pathogen pressures, in its different forms and intensities, has shaped the patterns of diversity of the human genome at the population level. These studies help to distinguish genes playing essential, non-redundant functions in host defence from genes variation in which has conferred selective advantages to specific human populations and/or has been acquired through admixture with archaic hominins, such as Neandertals. However, with the improvements in hygiene and the advent of antibiotics and vaccination, pressures imposed by pathogens have recently been relaxed. Accumulating evidence suggests that alleles having conferred an advantage against infection in the past may nowadays be associated with increased risk to develop immune-related disorders, such as autoimmunity and inflammation.
© 2016 médecine/sciences – Inserm
Diversité génétique de l’espèce humaine
L’étude de la diversité génétique humaine permet de retracer l’histoire évolutive de notre espèce et de mieux comprendre la variabilité phénotypique des populations modernes. L’avènement des technologies à haut débit et leur utilisation pan-génomique ont permis de mettre en évidence le niveau de variabilité génétique dans de nombreuses populations humaines. La version la plus récente du 1000 Genomes Project recense ainsi plus de 88 millions de variants, dont la plupart ne portent que sur un seul nucléotide [1]. Les forces génomiques (mutations et événements de recombinaison), l’histoire démographique de notre espèce et la sélection naturelle sont à l’origine de cette diversité qui est aujourd’hui observée dans l’espèce humaine.
Au cours de notre évolution, de nombreuses migrations ont participé à façonner la variabilité génétique humaine (Figure 1). En effet, le déplacement d’une population peut s’accompagner d’une réduction importante de sa taille efficace1, laissant libre cours à l’action de la dérive génétique, c’est-à-dire à la variation stochastique des fréquences alléliques au cours des générations. L’histoire démographique de l’espèce humaine inclut aussi de nombreux mélanges entre populations qui sont à l’origine d’une partie de la diversité génétique humaine. En effet, certaines populations migrantes se sont mêlées aux individus précédemment installés, augmentant la diversité génétique de la population receveuse. Cet échange a aussi pu se faire entre notre espèce et une autre suffisamment proche génétiquement pour qu’il y ait interfécondation. On parle alors de flux de gènes ou d’introgression.
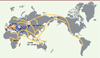 |
Figure 1. Modèle démographique des migrations humaines et territoires occupés par l’homme de Néandertal. L’espèce humaine est apparue il y a environ 200 000 ans en Afrique. Les populations non africaines modernes sont issues de la sortie côtière d’une population d’Afrique sub-saharienne vers l’Asie et le reste du monde, il y a entre 60 000 et 100 000 ans. Lors de leur arrivée en Eurasie, ces hommes modernes se sont retrouvés au contact de populations archaïques, dont les hommes de Néandertal, qui occupaient certains territoires jusqu’à – 40 000 ans (hachurés en bleu). En rouge sont indiqués les lieux où ont été retrouvés des ossements d’hommes archaïques qui ont été utilisés afin d’établir les séquences des génomes des Néandertaliens et des Dénisoviens (S : El Sidron, 49 000 ans ; V : Vindija, > 38 000 ans ; M : Mezmaiskaya, 60 000 – 70 000 ans ; A/D : Altaï et Denisova, > 50 000 ans). |
L’ensemble de cette diversité génétique a été également modelé par la sélection naturelle. Ce concept s’applique aux variations génétiques ayant un effet sur le phénotype et inclut l’adaptation génétique aux différents facteurs environnementaux. Ainsi, la fréquence des mutations ayant un effet fortement délétère sur l’organisme diminue rapidement jusqu’à ce que ces variants soient éliminés de la population par l’action de la sélection purificatrice 2. Dans le cas d’une mutation défavorable mais non délétère, le variant peut persister à faible fréquence dans la population. La sélection positive, ou d’autres formes d’adaptation génétique comme la sélection balancée 3, agissent quant à elles sur les variations génétiques conférant un avantage aux individus qui les portent. Par exemple, la sélection positive induit une augmentation rapide de la fréquence de ces mutations dans la population et traduit une adaptation à l’environnement dans lequel les individus évoluent, qu’il soit climatique, nutritionnel ou pathogénique [2–5]. La sélection naturelle influence ainsi le devenir des variations génétiques apparues chez l’homme ou issues d’un apport de diversité par mélange de populations.
Introgression d’allèles archaïques chez Homo sapiens
Nous savons aujourd’hui, grâce à la paléoanthropologie et la génétique, que notre espèce est apparue il y a moins de 200 000 ans en Afrique avant de coloniser le reste du monde dans les 60 000 dernières années. D’autres travaux ont révélé que des hommes archaïques, les hommes de Néandertal, ont vécu en Europe et en Asie de l’Ouest pendant près de 230 000 ans avant de s’éteindre il y a environ 40 000 ans [6, 7]. Des ossements vieux de 30 000 à 50 000 ans retrouvés dans la caverne de Denisova dans les monts Altaï4 ont montré que d’autres hommes archaïques, les Dénisoviens, vivaient, eux aussi, à cette période. Ainsi, Homo sapiens a pu cohabiter avec ces deux populations éteintes d’hominidés pendant de larges périodes [43] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 12, décembre 2012, page 1129
L’amélioration des techniques de séquençage d’ADN ancien a permis d’obtenir les séquences de l’intégralité du génome de plusieurs représentants de ces groupes d’hommes archaïques [8–12]. Dès lors, des comparaisons entre les génomes néandertaliens, dénisoviens et ceux des hommes modernes ont été réalisées et ont établi que les hommes de Néandertal ont contribué au génome des populations eurasiennes modernes à hauteur de 1,5 à 4 % et que les Dénisoviens ont contribué au génome des populations mélanésiennes modernes à un taux de 6 % [9, 11, 12]. Pour terminer, il semble que certaines populations africaines portent en elles des traces de mélanges génétiques avec des groupes d’hommes archaïques encore indéterminés [13]. Une partie de la diversité génétique de notre espèce a donc été acquise par croisements avec d’autres représentants du genre Homo aujourd’hui éteints.
Afin d’estimer l’intensité du flux de gènes archaïques dans le génome des populations humaines, diverses méthodes ont été développées. Leur application a révélé que l’introgression n’est pas homogène le long du génome des populations européennes et asiatiques, suggérant que l’ADN archaïque introgressé a été soumis à différentes pressions de sélection [14]. Ainsi, certaines régions telles que l’ensemble du chromosome X, le bras q du chromosome 8 ou les séquences codantes prises dans leur ensemble, sont pauvres en allèles néandertaliens. Si la pauvreté du chromosome X en allèles introgressés semble liée à une baisse de la fertilité des mâles issus des croisements entre Homo sapiens et hommes de Néandertal, il existe plus généralement une corrélation significative entre la forte densité en éléments fonctionnels présents dans le génome et la faible probabilité d’ascendance néandertalienne des allèles retrouvés dans ces régions [14]. Une partie de la diversité génétique acquise par introgression a donc eu des effets délétères sur les hommes modernes et a, par conséquent, été éliminée par la sélection purificatrice [14, 15].
À l’inverse, des haplotypes introgressés ont été maintenus dans des régions géniques spécifiques chez certaines populations modernes (Figure 2). Ainsi, les gènes codant la kératine présentent un fort taux d’introgression [14], le mélange des populations tibétaines avec des hommes archaïques semble avoir participé à leur adaptation à l’altitude [16] et certains gènes impliqués dans les réponses immunitaires mises en jeu à la suite d’une infection contiennent des allèles néandertaliens et dénisoviens dans différentes populations d’hommes modernes [17–22]. Ainsi, malgré l’effet globalement délétère des allèles hérités des populations archaïques, une partie de la diversité génétique apportée par ces individus aujourd’hui éteints s’est révélée avantageuse pour certaines populations d’hommes modernes et a participé à leur adaptation à leur environnement.
 |
Figure 2. Modèle d’introgression d’allèles archaïques dans les populations européennes modernes. Des haplotypes néandertaliens sont retrouvés dans les génomes des populations européennes et asiatiques modernes, témoignant d’un flux de gènes qui s’est effectué entre les hommes archaïques et les hommes modernes non-africains lors de la cohabitation de ces populations. Les datations estimées des ancêtres communs les plus récents entre l’homme et le chimpanzé (6-6,5 millions d’années) ainsi que l’homme moderne et les hommes archaïques (550 000-760 000 ans) sont indiquées (Ka : milliers d’années ; Ma : millions d’années). |
Les pathogènes, principale contrainte sélective
Notre espèce a été confrontée à une diversité considérable de microorganismes, qu’ils soient pathogéniques ou non. Avant l’amélioration des conditions d’hygiène et l’avènement de thérapies préventives et curatives, l’espérance de vie à la naissance n’excédait pas 25 ans à travers le monde, de nombreux enfants mourant en bas âge de maladies infectieuses (Figure 3) [23]. Des études de génétique évolutive ont permis d’apprécier l’intensité de la pression de sélection exercée par les microorganismes sur notre évolution. Une comparaison du génome humain avec celui du chimpanzé a ainsi montré que certains des gènes présentant des évidences moléculaires d’une évolution accélérée depuis la divergence entre ces deux espèces étaient enrichis en fonctions immunitaires [24]. Les études de plusieurs populations humaines ont aussi révélé que de nombreux gènes impliqués dans les réponses immunes font partie des régions génomiques ayant le plus participé à l’adaptation récente de ces groupes d’individus à leurs environnements respectifs [1, 25–27]. De plus, la variabilité génétique touchant des gènes de l’immunité peut être corrélée à la richesse pathogénique de l’environnement. La diversité locale des pathogènes se révèle donc être le principal facteur extérieur guidant l’adaptation des populations [28–30].
 |
Figure 3. Représentation schématique de la pression de sélection exercée par les pathogènes au cours de l’histoire évolutive humaine. Les pathogènes ont exercé une pression de sélection particulièrement forte tout au long de l’évolution de l’espèce humaine. Jusqu’au début du XXe siècle, l’espérance de vie à la naissance n’excédait ainsi pas 25 ans. L’amélioration des conditions d’hygiène et l’avènement des traitements préventifs et curatifs ont permis une réduction de l’incidence des maladies infectieuses (courbe pointillée bleue) et une augmentation de l’espérance de vie à la naissance (courbe orange). En parallèle, l’incidence des troubles immunologiques tels que les maladies auto-immunes et les allergies a augmenté (courbe violette), suggérant que certaines variations génétiques touchant la réponse immunitaire favorisées au cours de l’évolution de l’homme sont aujourd’hui impliquées dans des défaillances de la régulation de la réponse immune, du fait de la baisse de la charge pathogénique à laquelle sont soumis les organismes. |
Les populations actuelles sont constituées des descendants des individus qui ont survécu aux agressions incessantes des pathogènes. La génétique évolutive appliquée aux gènes de l’immunité s’avère complémentaire des études cliniques et épidémiologiques afin de mettre en évidence les gènes de la réponse immune dont la fonction est indispensable à la survie de l’organisme (c’est-à-dire évoluant sous sélection purificatrice) ainsi que ceux qui ont participé à l’adaptation d’une population à son environnement pathogénique précis (c’est-à-dire soumis à l’action de la sélection positive locale) [44] (→).
(→) Voir la Synthèse de J. Manry et L. Quintana-Murci, m/s n° 12, décembre 2012, page 1095
De nombreuses études en génétique évolutive se sont intéressées à l’évolution des gènes dont les produits sont impliqués dans l’immunité innée, première ligne de défense de l’hôte contre les pathogènes [31]. Les agents infectieux sont reconnus par un ensemble de récepteurs de l’immunité innée de l’hôte, tels que les récepteurs Toll-like (TLR), RIG1-like (RLR), NOD-like (NLR), les lectines de type C (CLR), ainsi que d’autres senseurs cytosoliques des acides nucléiques. Ces études ont révélé que certains membres des familles TLR, NLR et RLR ont été soumis à divers degrés de sélection négative (de faible à purificatrice), soulignant leur rôle essentiel dans la survie de l’hôte, alors que d’autres présentent des évidences moléculaires d’une participation à l’adaptation spécifique de populations humaines à leur environnement [22, 26, 32, 33].
Une fois activés par leurs ligands microbiens, les récepteurs recrutent des molécules dans le but de transmettre le signal au reste de la machinerie de signalisation de la cellule. Parmi les adaptateurs utilisés par les TLR, MYD88 (myeloid differentiation primary response gene 88) et TRIF (Toll/IL-1R[interleukin-1 receptor] domain-containing adaptor-inducing IFN-β [interferon beta]) présentent de très faibles taux de mutations non-synonymes5 au regard des variations qui ne modifient pas la séquence protéique. Cela traduit la nécessité de la conservation de leur séquence et souligne le rôle non redondant qu’ont ces molécules dans la transmission du signal de reconnaissance des agents infectieux [34].
En aval de la transduction du signal d’activation de la réponse immunitaire innée, les interférons (IFN), par exemple, font partie des effecteurs qui sont produits. Un gradient de forces sélectives a façonné ces gènes. Aucune mutation faux-sens6 n’a par exemple été identifiée dans IFNG (le gène codant l’IFN-γ, seul membre de la famille des interférons de type II), traduisant l’action d’une sélection purificatrice extrême sur cette séquence. Les IFN de type I sont, eux, soumis à des différences de contraintes sélectives particulièrement importantes. Alors que IFNA6 (gène codant l’IFN-α6), IFNA8, IFNA13 et IFNA14 semblent évoluer sous sélection purificatrice, ce qui souligne leur rôle essentiel, des mutations non synonymes sont retrouvées à fréquences élevées dans IFNA1, IFNA4, IFNA7, IFNA10, IFNA16 et IFNA17, ce qui témoigne d’un relâchement des pressions sélectives et laisse supposer une certaine redondance immunologique. Enfin, certaines variations non sens sont retrouvées dans IFNA10 et IFNE (gène codant l’IFN-ε) à l’état homozygote, suggérant que ces gènes pourraient être en voie de pseudogénisation. Pour terminer, certains membres du groupe des IFN de type III (les IFN-λ, principalement impliqués dans les réponses antivirales) présentent des signatures de sélection positive restreintes à certaines populations, ce qui laisse supposer qu’ils auraient conféré un avantage sélectif particulier, probablement relié aux défenses antivirales, à ces groupes d’individus [35].
L’évolution des gènes impliqués dans la réponse immunitaire innée semble donc complexe. Elle résulte en effet de l’action de différentes pressions de sélection avec une intensité dépendant de la fonction biologique des protéines [31]. Cependant, l’ensemble des études de génétique évolutive mentionnées ici ne s’est porté que sur un nombre restreint de gènes de l’immunité.
Une vue globale de la sélection naturelle sur les gènes de l’immunité innée
Nous avons récemment réalisé une étude large visant à mieux comprendre l’action de la sélection naturelle sur les gènes de l’immunité innée, dans leur ensemble, chez l’homme [22]. À cet effet, nous avons établi une liste de plus de 1 500 gènes impliqués dans ce processus cellulaire – en les distinguant par rapport à leur principale fonction – et estimé la diversité génétique présente dans ces séquences à travers l’étude des données de séquençage du 1000 Genomes Project [36]. Nous avons ensuite estimé l’étendue et l’intensité des forces sélectives ayant agit sur ces gènes à l’aide de différentes approches statistiques. Finalement, nous avons utilisé des estimations de l’ascendance néandertalienne des variations génétiques présentes dans les populations non africaines pour estimer la part de la diversité génétique qui est aujourd’hui observée chez les hommes modernes qui provient d’événements d’introgression. Cette analyse nous a permis de mettre en évidence l’étendue de l’action de la sélection naturelle, depuis la différenciation entre l’homme et le chimpanzé jusqu’aux adaptations récentes des populations à leur environnement, et de caractériser le rôle des allèles archaïques introgressés dans le génome des hommes modernes.
Estimation de l’importance fonctionnelle des gènes de l’immunité innée
Dans l’ensemble, les gènes de l’immunité innée ont accumulé moins de mutations non synonymes que le reste des séquences codantes du génome depuis la divergence entre l’homme et le chimpanzé. Cela suppose que la fonction de ces gènes est essentielle et qu’une perturbation de ces séquences protéiques est délétère pour l’organisme. Cette notion est particulièrement bien illustrée par les exemples de TRAF3 (tumor necrosis factor receptor associated factor 3) et STAT2 (signal transducer and activator of transcription 2), deux gènes codant des protéines de signalisation de l’immunité innée faisant partie des séquences codantes les plus contraintes du génome. Ces gènes ont été précédemment rapportés comme étant impliqués dans des phénotypes cliniques et immunologiques particulièrement critiques comme la susceptibilité mendélienne aux infections mycobactériennes, l’auto-immunité ou l’encéphalite induite par le virus de l’herpès de type 1 [37, 38].
Malgré tout, l’intensité des contraintes sélectives agissant sur les gènes de l’immunité innée n’est pas homogène tant entre l’ensemble des gènes qu’au sein des groupes de molécules ayant une même fonction au cours de la réponse immunitaire (Figure 4). Ainsi, les senseurs cytosoliques des acides nucléiques (CNAS pour cytosolic nucleic acid sensors) présentent une diversité génétique similaire à celle observée dans les gènes codant les TLR endosomaux (TLR3, 7, 8 et 9), qui participent eux aussi à la reconnaissance des acides nucléiques des agents infectieux. Cette diversité est moindre comparée aux autres gènes codant des senseurs microbiens, ce qui suggère que toute modification de la séquence de ces protéines est particulièrement délétère pour l’organisme. Il apparaît donc important de mieux caractériser ces molécules dont les fonctions sont encore peu décrites à ce jour dans la réponse immunitaire mise en jeu immédiatement après l’infection.
 |
Figure 4. Action de la sélection purificatrice sur les gènes de l’immunité innée chez l’homme. Nous avons établi une liste de 1 553 gènes impliqués dans la réponse immunitaire immédiate chez l’homme que nous avons classés en fonction de leur principale fonction : 274 senseurs (récepteurs et co-récepteurs), 46 adaptateurs, 245 molécules impliquées dans la transduction du signal, 93 facteurs et co-facteurs de transcription, 284 effecteurs, 70 récepteurs secondaires, 310 régulateurs de la réponse immunitaire innée, 68 molécules accessoires et 163 molécules dont la fonction ne permet pas de les classer dans les familles décrites précédemment. Nous avons par la suite estimé l’intensité de l’action de la sélection purificatrice agissant sur l’ensemble des séquences codantes du génome, qu’elles soient impliquées ou non dans l’immunité innée (A) à l’aide de la valeur f [22]. Nous avons constaté que, bien que l’action de la sélection négative sur l’ensemble des séquences codantes du génome est fortement hétérogène, la valeur f moyenne des gènes impliqués dans l’immunité innée est significativement inférieure à celle des autres gènes. Nous avons ensuite comparé les distributions des valeurs f des gènes impliqués dans l’immunité innée en nous basant sur leur principale fonction (B) . Plus la valeur f est faible (proche de 0), plus la séquence codante est contrainte, laissant supposer un rôle fonctionnel particulièrement important. |
Immunité innée et adaptation à l’environnement
Comme nous l’avons précédemment mentionné, de nombreux gènes de l’immunité ont pu être identifiés au cours des études visant à mettre en évidence les régions génomiques ayant participé à l’adaptation des populations humaines à leur environnement. Nous avons identifié avec notre analyse 57 gènes de l’immunité innée présentant des signaux de sélection positive très forts dans certaines populations humaines spécifiques7 [22]. La réalisation d’une cartographie fine de ces signaux nous a permis de proposer un ensemble de mutations faux-sens et non-sens6 comme étant les cibles de cette sélection positive. Nous avons également estimé que la plupart de ces mutations « adaptatives » étaient apparues il y a 6 000 à 13 000 ans, ce qui correspond à une période de changements de style de vie et d’environnement importants pour l’homme : le passage d’un mode de vie nomade chasseur-cueilleur à un mode de vie sédentaire agriculteur. Cette transition s’est accompagnée d’une augmentation de la densité de population, de crises alimentaires ainsi que de contacts prolongés avec le bétail et les déchets organiques. Cela a provoqué une modification de l’exposition aux pathogènes qui semble expliquer l’adaptation génétique importante qui a eu lieu à cette époque.
Introgression d’allèles néandertaliens dans les gènes de l’immunité innée
Des travaux récents ont étudié les génomes de l’homme de Néandertal et des hommes modernes (non-)Africains. En se basant sur diverses propriétés telles que les longueurs d’haplotypes, la présence ou non d’allèles dérivés ainsi que les divergences génétiques, cette étude a déterminé la probabilité d’ascendance néandertalienne de chacune des positions variables du génome dans les populations européennes et asiatiques du 1000 Genomes Project [14]. Tirant profit de ces calculs, nous avons montré que la probabilité de cette ascendance était plus importante pour les gènes de l’immunité innée que pour l’ensemble des autres séquences codantes du génome [22]. Elle reste cependant faible, ce qui est en accord avec une forte sélection négative ayant agi sur les allèles archaïques introgressés dans les séquences codantes. Les hypothèses d’une sélection négative faible ou d’un effet avantageux modéré des allèles introgressés semblent pouvoir concilier cette observation avec celle d’une contrainte sélective forte ayant globalement agi sur les gènes de l’immunité innée. Malgré tout, parmi les gènes présentant le plus fort taux d’introgression, nous avons identifié respectivement 76 et 78 gènes dans les populations européennes et asiatiques, qui sont impliqués dans la réponse immunitaire innée [22]. Si certains locus comme le cluster OAS (oligoadenylate synthetase genes) avaient été préalablement décrits grâce à des études « gène candidat » [21], nous en rapportons d’autres tels que IFITM3 (interferon-induced transmembrane protein 3) pour lequel des variations génétiques ont été décrites pour leur implication dans des différences de morbidité et de mortalité à la suite d’une infection par le virus de la grippe [39].
Plus spécifiquement, nous avons remarqué que pour deux de ces locus, SIRT1 (NAD-dependent deacetylase sirtuin-1) chez les Asiatiques et TLR1-6-10 chez les Européens, des signaux de sélection positive récente étaient également détectés [22]. Une étude indépendante a montré que deux haplotypes néandertaliens et un haplotype dénisovien sont retrouvés dans les génomes d’hommes modernes pour ce dernier cluster, à des fréquences laissant supposer qu’ils ont conféré un avantage sélectif aux populations dans lesquelles ils ont été introgressés (Figure 5) [17]. Ces résultats, ainsi que d’autres études utilisant des approches gène-candidat pour les gènes HLA (human leukocyte antigen, ou complexe majeur d’histocompatibilité) de classe I, le cluster des OAS ou STAT2, suggèrent donc qu’une part de notre immunité a été héritée des hommes archaïques et qu’une fraction de cet héritage a conféré un avantage sélectif aux hommes modernes [17, 18, 20–22].
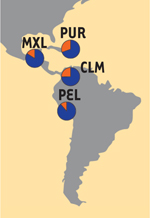
 |
Figure 5. Répartition géographique des haplotypes archaïques retrouvés au locus TLR1-6-10. Sont représentées en orange les fréquences des haplotypes « archaïques » (Néandertaliens et/ou Dénisoviens) et en bleu celles des haplotypes non-archaïques dans les populations du projet 1000 Genomes. YRI : Yoruba d’Ibadan, Nigéria ; LWK : Luhya de Webuye, Kenya ; GWD : Gambiens de la division de West Coast, Gambie ; MSL : Mende, Sierra Leone ; ESN : Esan, Nigéria ; ASW : Afro-Américains du Sud-Ouest des États-Unis ; ACB : Afro-Caribéens de la Barbade ; CEU : résidents de l’Utah d’origines d’Europe du Nord et de l’Ouest ; TSI : Toscans, Italie ; FIN : Finlandais, Finlande ; GBR : Britanniques, Angleterre et Écosse ; IBS : Ibériques, Espagne ; CHB : Chinois Han de Pékin, Chine ; JPT : Japonais de Tokyo, Japon ; CHS : Chinois Han du Sud, Chine ; CDX : Chinois Dai de Xishuangbanna, Chine ; KHV : Kinh de Hô-Chi-Minh-Ville, Vietnam ; GIH : Indiens Gujarati de Houston, États-Unis ; PJL : Pendjabis de Lahore, Pakistan ; BEB : Bengalis, Bengladesh ; STU : Tamil du Sri Lanka, Royaume-Uni ; ITU : Indiens Télugus, Royaume-Uni ; MXL : résidents de Los Angeles d’origines Mexicaines, États-Unis ; PUR : Portoricains, Porto Rico ; CLM : Colombiens de Medellin, Colombie ; PEL : Péruviens de Lima, Pérou. |
Adaptation aux pathogènes et risque de dérèglement du système immunitaire
Plusieurs gènes que nous avons identifiés au cours de notre étude comme soumis à la sélection positive dans une population donnée ont également été associés à des phénotypes infectieux, auto-immuns ou inflammatoires [22]. C’est par exemple le cas de FER ([Fps/Fes related] tyrosine kinase), IL(interleukin)4 et IRGM (immunity-related GTPase family M, ou interferon-inducible protein 1, IFI1) qui ont tous trois été décrits dans les maladies inflammatoires chroniques de l’intestin. Nombre d’autres régions ayant participé à l’adaptation des populations humaines à leur environnement contiennent des variations génétiques qui ont été rapportées, au cours d’autres travaux, pour leur implication dans des maladies inflammatoires [3, 29, 40]. Ainsi, certains allèles ayant conféré un avantage sélectif au cours de l’évolution de notre espèce s’avèrent être, aujourd’hui, des facteurs de risques. Cette dichotomie apparente semble trouver explication au sein de l’hypothèse hygiéniste. Les allèles soumis à la sélection positive ont, pendant des milliers d’années, permis aux individus de se défendre efficacement contre les pathogènes. Cet avantage sélectif explique que leurs fréquences alléliques soient aujourd’hui relativement élevées dans la population. Cependant, l’amélioration des conditions d’hygiène, le développement des vaccins et l’avènement des traitements curatifs, qui se sont faits récemment à l’échelle évolutive, ont contribué à diminuer l’exposition des êtres humains aux pathogènes dans les pays où l’accès aux soins est aisé (Figure 3). Notre système immunitaire, qui s’est amélioré au fil de notre évolution dans le but de nous permettre de nous défendre efficacement face aux agressions extérieures, se retrouve aujourd’hui face à une charge pathogénique plus faible sans pour autant avoir perdu sa capacité d’activation. Il participe donc à l’augmentation de notre susceptibilité à diverses pathologies au cours desquelles il est déréglé.
Cette hypothèse est particulièrement bien illustrée par le cas du cluster TLR1-6-10. Comme mentionné plus haut, des haplotypes néandertaliens et dénisoviens sont retrouvés dans le génome de populations européennes et asiatiques à des fréquences qui suggèrent qu’ils résultent d’un événement d’introgression adaptative [17]. Une analyse fonctionnelle a montré que ces haplotypes sont associés à une plus forte expression des trois récepteurs dans des lignées lymphoblastoïdes. De plus, les allèles archaïques introgressés sont associés à une augmentation du risque de développer des allergies alors qu’ils semblent protéger des infections par Helicobacter pylori, la séroprévalence étant significativement diminuée chez les individus qui les portent [41, 42]. Ainsi, la diversité génétique apportée par les hommes archaïques au niveau de ces trois gènes a permis d’améliorer notre immunité innée mais elle a, en contrepartie, augmenté le risque que nous voyions notre système immunitaire reconnaître des molécules non pathogéniques et induire ainsi une réponse inappropriée.
Conclusion
Les approches de génétique évolutive et de génétique des populations appliquées au système immunitaire inné sont complémentaires des analyses cliniques et épidémiologiques. Elles permettent de mettre en évidence l’importance fonctionnelle et universelle de certains gènes, la participation d’autres à des événements d’adaptation de certaines populations humaines à leur environnement spécifique et le maintien d’allèles archaïques qui ont pu conférer un avantage sélectif à un groupe d’individus. L’ensemble des variations génétiques ainsi identifiées, résultat de notre passé démographique et sélectif, permettent non seulement de proposer de nouveaux candidats à des études fonctionnelles mais aussi de souligner l’implication de certains gènes dans les différences de susceptibilité aux maladies, qu’elles soient infectieuses ou non, qui font intervenir le système immunitaire.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Cette étude a été financée par l’Institut Pasteur, le CNRS, et l’Agence nationale de la recherche.
La sélection naturelle peut prendre de nombreuses formes et agir avec des intensités différentes. Les séquences codantes sont principalement soumises à l’action de la sélection dite « négative » ou, dans sa forme la plus extrême, « purificatrice ». Cette sélection diminue la fréquence des mutations qui s’avèrent défavorables à leurs porteurs dans un environnement donné.
Une substitution dans un codon peut se traduire par (1) le même acide aminé : on dit qu’elle est synonyme. Il n’y aura pas de modification de l’acide aminé traduit ; (2) un acide aminé différent : on dit qu’elle est faux-sens. La modification de la séquence protéique est dans la plupart des cas délétère mais elle peut aussi être neutre ou bénéfique pour l’individu qui la porte, en fonction des conséquences fonctionnelles du changement d’acide aminé ; (3) un codon de terminaison : on dit qu’elle est non-sens. La protéine traduite sera tronquée à cet endroit.
Différentes stratégies permettent de mettre en évidence les signaux de sélection positive dans le génome. Dans notre étude, nous avons utilisé une combinaison de six statistiques reposant sur les longueurs haplotypiques et les signaux de différenciation entre populations, que nous avons regroupées suivant la méthode de Fisher. Ce score a été par la suite calculé pour chacune des positions variables du génome rapportées dans le 1000 Genomes Project chez les Yorubas (Afrique), les résidents de l’Utah d’origines de l’Europe du Nord et de l’Ouest (Europe) et les Hans de Pékin (Asie) séparément.
Références
- 1000 Genomes Project Consortium. A global reference for human genetic variation. Nature 2015 ; 526 : 68–74. [CrossRef] [PubMed] [Google Scholar]
- Vitti JJ, Grossman SR, Sabeti PC. Detecting natural selection in genomic data. Annu Rev Genet 2013 ; 47 : 97–120. [CrossRef] [PubMed] [Google Scholar]
- Barreiro LB, Quintana-Murci L. From evolutionary genetics to human immunology : how selection shapes host defence genes. Nat Rev Genet 2010 ; 11 : 17–30. [CrossRef] [PubMed] [Google Scholar]
- Karlsson EK, Kwiatkowski DP, Sabeti PC. Natural selection and infectious disease in human populations. Nat Rev Genet 2014 ; 15 : 379–393. [CrossRef] [PubMed] [Google Scholar]
- Nielsen R, Hellmann I, Hubisz M, et al. Recent and ongoing selection in the human genome. Nat Rev Genet 2007 ; 8 : 857–868. [CrossRef] [PubMed] [Google Scholar]
- Hublin JJ. Out of Africa : modern human origins special feature. The origin of Neandertals. Proc Natl Acad Sci USA 2009 ; 106 : 16022–16027. [CrossRef] [Google Scholar]
- Higham T, Douka K, Wood R, et al. The timing and spatiotemporal patterning of Neanderthal disappearance. Nature 2014 ; 512 : 306–309. [CrossRef] [PubMed] [Google Scholar]
- Castellano S, Parra G, Sanchez-Quinto FA, et al. Patterns of coding variation in the complete exomes of three Neandertals. Proc Natl Acad Sci USA 2014 ; 111 : 6666–6671. [CrossRef] [Google Scholar]
- Green RE, Krause J, Briggs AW, et al. A draft sequence of the Neandertal genome. Science 2010 ; 328 : 710–722. [CrossRef] [PubMed] [Google Scholar]
- Meyer M, Kircher M, Gansauge MT, et al. A high-coverage genome sequence from an archaic Denisovan individual. Science 2012 ; 338 : 222–226. [CrossRef] [PubMed] [Google Scholar]
- Prufer K, Racimo F, Patterson N, et al. The complete genome sequence of a Neanderthal from the Altai Mountains. Nature 2014 ; 505 : 43–49. [CrossRef] [PubMed] [Google Scholar]
- Reich D, Green RE, Kircher M, et al. Genetic history of an archaic hominin group from Denisova Cave in Siberia. Nature 2010 ; 468 : 1053–1060. [CrossRef] [PubMed] [Google Scholar]
- Lachance J, Vernot B, Elbers CC, et al. Evolutionary history and adaptation from high-coverage whole-genome sequences of diverse African hunter-gatherers. Cell 2012 ; 150 : 457–469. [CrossRef] [PubMed] [Google Scholar]
- Sankararaman S, Mallick S, Dannemann M, et al. The genomic landscape of Neanderthal ancestry in present-day humans. Nature 2014 ; 507 : 354–357. [CrossRef] [PubMed] [Google Scholar]
- Vernot B, Akey JM. Resurrecting surviving Neandertal lineages from modern human genomes. Science 2014 ; 343 : 1017–1021. [CrossRef] [PubMed] [Google Scholar]
- Huerta-Sanchez E, Jin X, Asan, et al. Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA. Nature 2014 ; 512 : 194–197. [CrossRef] [PubMed] [Google Scholar]
- Dannemann M, Andres AM, Kelso J. Introgression of Neandertal- and Denisovan-like haplotypes contributes to adaptive variation in human Toll-like receptors. Am J Hum Genet 2016 ; 98 : 22–33. [CrossRef] [PubMed] [Google Scholar]
- Abi-Rached L, Jobin MJ, Kulkarni S, et al. The shaping of modern human immune systems by multiregional admixture with archaic humans. Science 2011 ; 334 : 89–94. [CrossRef] [PubMed] [Google Scholar]
- Mendez FL, Watkins JC, Hammer MF. Global genetic variation at OAS1 provides evidence of archaic admixture in Melanesian populations. Mol Biol Evol 2012 ; 29 : 1513–1520. [CrossRef] [PubMed] [Google Scholar]
- Mendez FL, Watkins JC, Hammer MF. A haplotype at STAT2 Introgressed from neanderthals and serves as a candidate of positive selection in Papua New Guinea. Am J Hum Genet 2012 ; 91 : 265–274. [CrossRef] [PubMed] [Google Scholar]
- Mendez FL, Watkins JC, Hammer MF. Neandertal origin of genetic variation at the cluster of OAS immunity genes. Mol Biol Evol 2013 ; 30 : 798–801. [CrossRef] [PubMed] [Google Scholar]
- Deschamps M, Laval G, Fagny M, et al. Genomic signatures of selective pressures and introgression from archaic Hominins at human innate immunity genes. Am J Hum Genet 2016 ; 98 : 5–21. [CrossRef] [PubMed] [Google Scholar]
- Casanova JL, Abel L. Inborn errors of immunity to infection : the rule rather than the exception. J Exp Med 2005 ; 202 : 197–201. [CrossRef] [PubMed] [Google Scholar]
- Bustamante CD, Fledel-Alon A, Williamson S, et al. Natural selection on protein-coding genes in the human genome. Nature 2005 ; 437 : 1153–1157. [CrossRef] [PubMed] [Google Scholar]
- Barreiro LB, Laval G, Quach H, et al. Natural selection has driven population differentiation in modern humans. Nat Genet 2008 ; 40 : 340–345. [CrossRef] [PubMed] [Google Scholar]
- Grossman SR, Andersen KG, Shlyakhter I, et al. Identifying recent adaptations in large-scale genomic data. Cell 2013 ; 152 : 703–713. [CrossRef] [PubMed] [Google Scholar]
- Pickrell JK, Coop G, Novembre J, et al. Signals of recent positive selection in a worldwide sample of human populations. Genome Res 2009 ; 19 : 826–837. [CrossRef] [PubMed] [Google Scholar]
- Fumagalli M, Cagliani R, Pozzoli U, et al. Widespread balancing selection and pathogen-driven selection at blood group antigen genes. Genome Res 2009 ; 19 : 199–212. [CrossRef] [PubMed] [Google Scholar]
- Fumagalli M, Sironi M, Pozzoli U, et al. Signatures of environmental genetic adaptation pinpoint pathogens as the main selective pressure through human evolution. PLoS Genet 2011 ; 7 : e1002355. [CrossRef] [PubMed] [Google Scholar]
- Prugnolle F, Manica A, Charpentier M, et al. Pathogen-driven selection and worldwide HLA class I diversity. Curr Biol 2005 ; 15 : 1022–1027. [CrossRef] [PubMed] [Google Scholar]
- Quintana-Murci L, Clark AG. Population genetic tools for dissecting innate immunity in humans. Nat Rev Immunol 2013 ; 13 : 280–293. [CrossRef] [PubMed] [Google Scholar]
- Barreiro LB, Ben-Ali M, Quach H, et al. Evolutionary dynamics of human Toll-like receptors and their different contributions to host defense. PLoS Genet 2009 ; 5 : e1000562. [CrossRef] [PubMed] [Google Scholar]
- Vasseur E, Boniotto M, Patin E, et al. The evolutionary landscape of cytosolic microbial sensors in humans. Am J Hum Genet 2012 ; 91 : 27–37. [CrossRef] [PubMed] [Google Scholar]
- Fornarino S, Laval G, Barreiro LB, et al. Evolution of the TIR domain-containing adaptors in humans : swinging between constraint and adaptation. Mol Biol Evol 2011 ; 28 : 3087–3097. [CrossRef] [PubMed] [Google Scholar]
- Manry J, Laval G, Patin E, et al. Evolutionary genetic dissection of human interferons. J Exp Med 2011 ; 208 : 2747–2759. [CrossRef] [PubMed] [Google Scholar]
- Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature 2012 ; 491 : 56–65. [CrossRef] [PubMed] [Google Scholar]
- Boisson-Dupuis S, Kong XF, Okada S, et al. Inborn errors of human STAT1: allelic heterogeneity governs the diversity of immunological and infectious phenotypes. Curr Opin Immunol 2012 ; 24 : 364–378. [CrossRef] [PubMed] [Google Scholar]
- Perez de Diego R, Sancho-Shimizu V, Lorenzo L, et al. Human TRAF3 adaptor molecule deficiency leads to impaired Toll-like receptor 3 response and susceptibility to herpes simplex encephalitis. Immunity 2010 ; 33 : 400–411. [CrossRef] [PubMed] [Google Scholar]
- Everitt AR, Clare S, Pertel T, et al. IFITM3 restricts the morbidity and mortality associated with influenza. Nature 2012 ; 484 : 519–523. [CrossRef] [PubMed] [Google Scholar]
- Brinkworth JF, Barreiro LB. The contribution of natural selection to present-day susceptibility to chronic inflammatory and autoimmune disease. Curr Opin Immunol 2014 ; 31 : 66–78. [CrossRef] [PubMed] [Google Scholar]
- Hinds DA, McMahon G, Kiefer AK, et al. A genome-wide association meta-analysis of self-reported allergy identifies shared and allergy-specific susceptibility loci. Nat Genet 2013 ; 45 : 907–911. [CrossRef] [PubMed] [Google Scholar]
- Mayerle J, den Hoed CM, Schurmann C, et al. Identification of genetic loci associated with Helicobacter pylori serologic status. JAMA 2013 ; 309 : 1912–1920. [CrossRef] [PubMed] [Google Scholar]
- Jordan B. Chroniques génomiques. Néandertal et Homo sapiens : To meet, or not to meet? Med Sci (Paris) 2012 ; 28 : 1129–1132. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Manry J, Quintana-Murci L. Génétique des populations et immunité chez l’homme. Med Sci (Paris) 2012 ; 28 : 1095–1101. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Modèle démographique des migrations humaines et territoires occupés par l’homme de Néandertal. L’espèce humaine est apparue il y a environ 200 000 ans en Afrique. Les populations non africaines modernes sont issues de la sortie côtière d’une population d’Afrique sub-saharienne vers l’Asie et le reste du monde, il y a entre 60 000 et 100 000 ans. Lors de leur arrivée en Eurasie, ces hommes modernes se sont retrouvés au contact de populations archaïques, dont les hommes de Néandertal, qui occupaient certains territoires jusqu’à – 40 000 ans (hachurés en bleu). En rouge sont indiqués les lieux où ont été retrouvés des ossements d’hommes archaïques qui ont été utilisés afin d’établir les séquences des génomes des Néandertaliens et des Dénisoviens (S : El Sidron, 49 000 ans ; V : Vindija, > 38 000 ans ; M : Mezmaiskaya, 60 000 – 70 000 ans ; A/D : Altaï et Denisova, > 50 000 ans). |
| Dans le texte | |
 |
Figure 2. Modèle d’introgression d’allèles archaïques dans les populations européennes modernes. Des haplotypes néandertaliens sont retrouvés dans les génomes des populations européennes et asiatiques modernes, témoignant d’un flux de gènes qui s’est effectué entre les hommes archaïques et les hommes modernes non-africains lors de la cohabitation de ces populations. Les datations estimées des ancêtres communs les plus récents entre l’homme et le chimpanzé (6-6,5 millions d’années) ainsi que l’homme moderne et les hommes archaïques (550 000-760 000 ans) sont indiquées (Ka : milliers d’années ; Ma : millions d’années). |
| Dans le texte | |
 |
Figure 3. Représentation schématique de la pression de sélection exercée par les pathogènes au cours de l’histoire évolutive humaine. Les pathogènes ont exercé une pression de sélection particulièrement forte tout au long de l’évolution de l’espèce humaine. Jusqu’au début du XXe siècle, l’espérance de vie à la naissance n’excédait ainsi pas 25 ans. L’amélioration des conditions d’hygiène et l’avènement des traitements préventifs et curatifs ont permis une réduction de l’incidence des maladies infectieuses (courbe pointillée bleue) et une augmentation de l’espérance de vie à la naissance (courbe orange). En parallèle, l’incidence des troubles immunologiques tels que les maladies auto-immunes et les allergies a augmenté (courbe violette), suggérant que certaines variations génétiques touchant la réponse immunitaire favorisées au cours de l’évolution de l’homme sont aujourd’hui impliquées dans des défaillances de la régulation de la réponse immune, du fait de la baisse de la charge pathogénique à laquelle sont soumis les organismes. |
| Dans le texte | |
 |
Figure 4. Action de la sélection purificatrice sur les gènes de l’immunité innée chez l’homme. Nous avons établi une liste de 1 553 gènes impliqués dans la réponse immunitaire immédiate chez l’homme que nous avons classés en fonction de leur principale fonction : 274 senseurs (récepteurs et co-récepteurs), 46 adaptateurs, 245 molécules impliquées dans la transduction du signal, 93 facteurs et co-facteurs de transcription, 284 effecteurs, 70 récepteurs secondaires, 310 régulateurs de la réponse immunitaire innée, 68 molécules accessoires et 163 molécules dont la fonction ne permet pas de les classer dans les familles décrites précédemment. Nous avons par la suite estimé l’intensité de l’action de la sélection purificatrice agissant sur l’ensemble des séquences codantes du génome, qu’elles soient impliquées ou non dans l’immunité innée (A) à l’aide de la valeur f [22]. Nous avons constaté que, bien que l’action de la sélection négative sur l’ensemble des séquences codantes du génome est fortement hétérogène, la valeur f moyenne des gènes impliqués dans l’immunité innée est significativement inférieure à celle des autres gènes. Nous avons ensuite comparé les distributions des valeurs f des gènes impliqués dans l’immunité innée en nous basant sur leur principale fonction (B) . Plus la valeur f est faible (proche de 0), plus la séquence codante est contrainte, laissant supposer un rôle fonctionnel particulièrement important. |
| Dans le texte | |
 |
Figure 5. Répartition géographique des haplotypes archaïques retrouvés au locus TLR1-6-10. Sont représentées en orange les fréquences des haplotypes « archaïques » (Néandertaliens et/ou Dénisoviens) et en bleu celles des haplotypes non-archaïques dans les populations du projet 1000 Genomes. YRI : Yoruba d’Ibadan, Nigéria ; LWK : Luhya de Webuye, Kenya ; GWD : Gambiens de la division de West Coast, Gambie ; MSL : Mende, Sierra Leone ; ESN : Esan, Nigéria ; ASW : Afro-Américains du Sud-Ouest des États-Unis ; ACB : Afro-Caribéens de la Barbade ; CEU : résidents de l’Utah d’origines d’Europe du Nord et de l’Ouest ; TSI : Toscans, Italie ; FIN : Finlandais, Finlande ; GBR : Britanniques, Angleterre et Écosse ; IBS : Ibériques, Espagne ; CHB : Chinois Han de Pékin, Chine ; JPT : Japonais de Tokyo, Japon ; CHS : Chinois Han du Sud, Chine ; CDX : Chinois Dai de Xishuangbanna, Chine ; KHV : Kinh de Hô-Chi-Minh-Ville, Vietnam ; GIH : Indiens Gujarati de Houston, États-Unis ; PJL : Pendjabis de Lahore, Pakistan ; BEB : Bengalis, Bengladesh ; STU : Tamil du Sri Lanka, Royaume-Uni ; ITU : Indiens Télugus, Royaume-Uni ; MXL : résidents de Los Angeles d’origines Mexicaines, États-Unis ; PUR : Portoricains, Porto Rico ; CLM : Colombiens de Medellin, Colombie ; PEL : Péruviens de Lima, Pérou. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




