Figure 3.
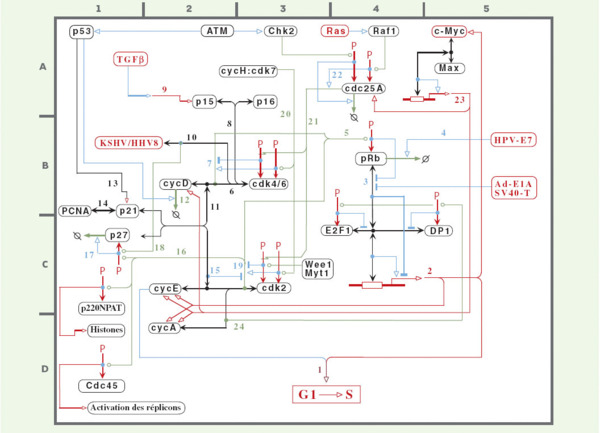
Interactions moléculaires de la transition G1→S et du contrôle du facteur de transcription E2F. Les symboles sont définis par Kohn [6]. (1) [coordonnées D3] La transition G1→S implique les gènes contrôlés par E2F et la cycline E. (2) [C5] Les gènes dépendants de E2F codent pour des protéines régulatrices telles que la cycline E et la protéine Myc, et des gènes dont les produits sont des enzymes impliquées dans la réplication (non incluses sur le schéma). Ces gènes sont activés par les complexes E2F1:DP1 (et d’autres hétérodimères E2F) et négativement réglés lorsque E2F est lié à pRb. Cette dernière possède donc un rôle majeur. (3) [B4] La liaison de pRb à l’hétérodimère d’E2F est inhibée lorsque pRb est phosphorylée. pRb est également inhibée par les protéines virales E1A et l’antigène grand T (SV40-T). (4) [B5] La dégradation de pRb est stimulée par la protéine E7 du virus du papillome. (5) [B4] pRb est phosphorylée par les complexes cycline D:Cdk4/6 et cycline E:Cdk2. Ces deux complexes kinases sont nécessaires pour phosphoryler pRb complètement et bloquer l’interaction entre pRb et E2F. (6)[B.2] Cdk4 et Cdk6 sont actives quand elles sont couplées à la cycline D. (7)[B2] Cdk4 et Cdk6 peuvent être phosphorylées à des sites qui stimulent ou bloquent leur activité. (8)[B2] Les CKI p15 ou p16 peuvent se fixer à Cdk4 ou Cdk6 et bloquer leur fixation de la cycline D. Cela a pour effet d’inactiver les kinases. (9)[A2] Le TGFβ active la transcription de p15 par la voie des Smad (non montré). (10)[B2] La protéine Cdk6 peut être inactivée par déplacement de la cycline D par la protéine virale KSHV/HHV8 du virus du sarcome de Kaposi. (11)[B2] Les hétérodimères cycline D:Cdk4/6 peuvent se lier à p21cip1 ou p27kip1, ce qui a pour effet de les stabiliser. (12)[B2] La cycline D a une demi-vie courte, d’environ 30 minutes. Elle est dégradée dans le protéasome après ubiquitinylation. (13)[B.2] La protéine p53 active p21cip1. (14)[B2] p21cip1 se fixe à PCNA, un co-facteur de l’ADN polymérase. (15)[C.2] p21cip1 ou p27kip1 se lient aux complexes cycline E:Cdk2 et bloquent leur activité kinase (par opposition aux complexes cycline D:Cdk4/6 (voir [11] ci-dessus). (16)[C2] p27kip1 est phosphorylée par la cycline E:Cdk2. p27kip1 est donc à la fois un substrat et un inhibiteur des complexes kinases cycline E. (17)[C1] p27kip1 est dégradée après phosphorylation. (18)[C2] La liaison de la protéine virale KSHV/HHV8 stimule la dégradation de p27kip1. (19)[C2-3] Les complexes cycline E:Cdk2 (et cycline D:Cdk4/6) sont phosphorylés au niveau de sites d’activation et d’inhibition. (20)[A3] Cdk2, Cdk4 et Cdk6 sont phosphorylées et activées par la cycline H:Cdk7 (encore connue sous le nom de CAK), qui est un composant du facteur de transcription TFIIH. (21)[B3] Cdk2, Cdk4 et Cdk6 sont activées par déphosphorylation par la phosphatase Cdc25A. (22)[A4] Cdc25A est activée par phosphorylation par Raf1 dans la voie de signalisation Ras. (23)[A5] La transcription du gène de Cdc25A est activée par les complexes c-Myc:Max. (24)[D2-3] Le complexe cycline A:Cdk2 phosphoryle E2F1 et DP1, ce qui produit la dissociation du complexe E2F1:DP1, avec pour effet d’arrêter l’activité de E2F en fin de phase S. Les conventions utilisées pour le choix des couleurs sont les suivantes; en rouge, modifications covalentes et voies de transcription; en vert, modifications enzymatiques; en bleu: effets stimulants ou inhibiteurs; en noir, liaisons intermoléculaires; lettres rouges: inputs et outputs.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




