| Numéro |
Med Sci (Paris)
Volume 41, Numéro 6-7, Juin-Juillet 2025
|
|
|---|---|---|
| Page(s) | 543 - 546 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2025090 | |
| Publié en ligne | 7 juillet 2025 | |
Culture en trois dimensions de cellules épithéliales polarisées sur billes pour l’imagerie subcellulaire de l’infection par les virus influenza
3D culture of polarised epithelial cells on beads for subcellular imaging of influenza virus infection
1
Institut Curie, PSL Research University, Sorbonne Université, CNRS UMR144, Paris, France
2
Institut Pasteur, Université Paris Cité, CNRS UMR3569, Biologie des ARN et virus Influenza, Paris, France
Les virus influenza ou virus de la grippe, responsables d’épidémies saisonnières, représentent la cause principale d’infections respiratoires sévères, et sont à l’origine d’environ 500 000 décès annuels. Les virus influenza de type A, du fait de l’existence d’un réservoir animal, sont régulièrement à l’origine de l’émergence de nouveaux virus zoonotiques ou pandémiques, avec des conséquences potentiellement dévastatrices [1]. L’expansion, depuis 2021, de virus aviaires hautement pathogènes de sous-type H5N1 (clade 2.3.4.4b), aujourd’hui présents dans la plupart des régions du monde et capables d’infecter un large spectre de mammifères, est particulièrement inquiétante [2]. Le développement de nouvelles stratégies thérapeutiques est donc plus que jamais nécessaire. Il requiert une compréhension approfondie des mécanismes moléculaires de la réplication virale, notamment les mécanismes d’exploitation des voies de transport intracellulaire pour assurer l’adressage des composants viraux vers les sites d’assemblage de nouveaux virus.
Pour des raisons historiques et pratiques, la plupart des études utilisent des lignées de cellules immortalisées non polarisées cultivées en deux dimensions. Bien que ces modèles cellulaires simples aient permis des avancées substantielles, ils ont le défaut de ne pas reproduire l’organisation subcellulaire des cellules cibles naturelles des virus influenza : les cellules de l’épithélium respiratoire. En effet, dans les lignées cellulaires non polarisées, le centrosome, centre organisateur principal des microtubules, est localisé dans la région périnucléaire, où il assure la nucléation des microtubules qui croissent en direction de la membrane plasmique : les extrémités négatives des microtubules sont donc situées au centre de la cellule, tandis que les extrémités positives en croissance sont périphériques. Au contraire, dans les cellules épithéliales polarisées, le centrosome se trouve au pôle apical et assure la croissance des microtubules vers l’intérieur de la cellule [3]. Du fait de cette organisation inversée, le transport de molécules vers la périphérie cellulaire est contrôlé par des moteurs moléculaires distincts : une kinésine dans le cas des cellules non polarisées [4], et la dynéine dans les cellules épithéliales polarisées [5] (Figure 1A). Pour étudier le transport intracellulaire des composants viraux, il est donc plus pertinent d’utiliser des cellules polarisées. Des cultures de cellules épithéliales humaines primaires différenciées, d’origine nasale ou bronchique, sont commercialisées et de plus en plus utilisées, et permettent par exemple de tester l’activité antivirale de molécules [6]. En revanche, ces cultures se prêtent mal à l’imagerie à l’échelle subcellulaire, notamment du fait de la présence d’une membrane de polyester rigide sur laquelle les cellules adhèrent à l’interface air-liquide, et qui réfracte la lumière et rend difficile la préparation de coupes de tissus. Pour contourner ces difficultés techniques et mettre en place un système maniable et simple d’utilisation, nous avons cultivé un sousclone de cellules épithéliales de côlon humain capables de se polariser et de se différencier rapidement [7], les cellules Caco-2/TC7, sur des billes de dextran de 175 µm de diamètre recouvertes de gélatine porcine (billes Cytodex 3), de manière à obtenir une monocouche de cellules polarisées [8, 9] (Figure 1B).
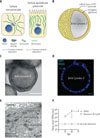 |
Figure 1 Polarisation des cellules Caco-2/TC7 cultivées sur billes de dextran Cytodex 3. A. Représentation schématique de l’organisation du cytosquelette de microtubules dans des cellules non polarisées et dans des cellules épithéliales polarisées. B. Représentation schématique de cellules épithéliales cultivées en trois dimensions (3D) sur billes Cytodex 3. C. Aspect columnaire des cellules Caco-2/TC7 après deux semaines de culture sur billes Cytodex 3. Barre d’échelle = 100 µm. D. Image, par microscopie confocale, de cellules Caco-2/TC7 polarisées sur Cytodex 3. Un anticorps dirigé contre PALS1 marque la membrane cytoplasmique apicale (en vert). L’agent intercalant DAPI marque l’ADN (en bleu). Barre d’échelle = 25 µm. E. Image de microscopie électronique à transmission d’une coupe ultrafine de cellules Caco-2/TC7 polarisées sur Cytodex 3. Différents compartiments cellulaires sont indiqués par des flèches noires : villosités, jonction intercellulaire serrée, appareil de Golgi, réticulum endoplasmique, mitochondrie. Barre d’échelle = 1 µm F. Titrage de la quantité de particules virales infectieuses présentes dans le milieu de culture à 24, 48 et 72 heures post-infection (hpi) par la souche A/WSN/33 (avec une multiplicité d’infection = 0,01 PFU/cellule), en présence ou en absence de l’inhibiteur de la protéase virale Baloxavir (0,1 µM). La ligne en trait pointillé correspond à la limite de détection : 250 PFU/mL. |
Un système maniable, rapide et peu coûteux pour la production d’un épithélium polarisé infectable par le virus influenza
Après quatre jours de culture des cellules Caco-2/TC7 sur billes Cytodex 3, la quasi-totalité des billes sont recouvertes d’une monocouche de cellules. Au bout de deux semaines, les cellules acquièrent une morphologie columnaire évocatrice d’un état épithélial polarisé (Figure 1C). Pour nous assurer du caractère polarisé des cellules, nous avons dans un premier temps procédé à un immuno-marquage indirect fluorescent de la protéine PALS1 (polarity protein associated with LIN seven-1), un marqueur spécifique de la membrane apicale des cellules épithéliales. Le signal obtenu indique la mise en place d’une membrane apicale à la périphérie des cellules Caco-2/TC7 cultivées sur Cytodex [9] (Figure 1D). Nous avons ensuite observé les cellules en microscopie électronique à transmission afin d’accéder à leur ultrastructure. Ainsi, nous avons constaté des caractéristiques spécifiques des cellules épithéliales polarisées : la présence de jonctions serrées, la localisation basale de l’appareil de Golgi, et la présence de microvillosités caractéristiques du pôle apical des cellules épithéliales digestives [9] (Figure 1E).
Pour vérifier que les cellules Caco-2/TC7 polarisées sur Cytodex sont permissives à l’infection par les virus influenza, nous avons réalisé des expériences d’infection avec une souche de laboratoire de soustype H1N1 (A/WSN/33), « à faible multiplicité d’infection » (0,01 PFU1/cellule), puis mesuré la quantité de particules infectieuses relarguées par les cellules dans le milieu extracellulaire toutes les 24 heures pendant trois jours. Nous avons constaté une concentration croissante de particules virales infectieuses dans le milieu de culture, atteignant jusqu’à 106 PFU/mL dès 48 heures après le début de l’infection. En revanche, en présence de la molécule antivirale Baloxavir, la concentration de virus infectieux est restée sous le seuil de détection pour la méthode utilisée [9] (Figure 1F). Ce résultat a permis de montrer que des particules infectieuses néoformées étaient bien produites à partir de ce système de culture de cellules épithéliales polarisées, qui peut donc être utilisé pour étudier les différentes phases du cycle réplicatif viral.
Différences et similitudes des altérations viro-induites des endomembranes selon l’état de polarisation des cellules infectées
Nous avons ensuite souhaité étudier, dans le système de cellules Caco-2/TC7 polarisées sur Cytodex, la capacité du virus à altérer certaines structures membranaires, comme cela a été décrit auparavant dans des modèles cellulaires en deux dimensions. Nous nous sommes d’abord intéressés à l’interaction des segments d’ARN génomiques viraux, associés à de multiples copies de la nucléoprotéine virale NP, avec la petite GTPase cellulaire RAB11 [10]. Des cellules Caco-2/TC7 cultivées sur billes ont été infectées, « à forte multiplicité d’infection » (10 PFU/cellule), pendant 8 heures, puis fixées, et enfin marquées par un mélange d’anticorps dirigés contre la protéine virale NP et la protéine RAB11 (Figure 2A). Les échantillons ont ensuite été analysés par microscopie optique confocale avec un objectif à grossissement 93X doté d’une grande distance de travail, permettant l’observation à l’échelle subcellulaire. Dans le cytoplasme des cellules infectées, nous avons constaté la colocalisation de structures ponctiformes marquées par l’anticorps anti-NP et correspondant aux génomes viraux, avec la GTPase RAB11 (Figure 2A, 2B), comme précédemment dans les cellules non polarisées. Nous avons également constaté l’accumulation de génomes viraux, mais non de RAB11, à la membrane plasmique, où a lieu l’assemblage et le bourgeonnement de nouvelles particules virales [9] (Figure 2A, 2B). La quantité de cellules dans lesquelles les génomes viraux ont atteint la périphérie de la cellule à 8 heures post-infection est plus grande dans les cellules polarisées que dans les cellules Caco-2/TC7 cultivées traditionnellement en deux dimensions (Figure 2C), ce qui suggère que le mode de transport intracellulaire est plus rapide et plus efficace dans les cellules épithéliales polarisées.
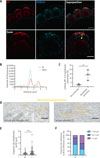 |
Figure 2 Imagerie des cellules Caco-2/TC7 cultivées sur billes Cytodex 3 et infectées avec le virus influenza A/WSN/33. A. Des cellules Caco-2/TC7 cultivées sur billes Cytodex 3 ont été infectées par la souche A/WSN/33 du virus influenza (avec une multiplicité d’infection = 10 PFU/cellule). Au bout de 8 heures, elles ont été fixées, puis immuno-marquées avec des anticorps dirigés contre la nucléoprotéine NP (en rouge) et contre RAB11 (en cyan), avant d’être visualisées par microscopie confocale avec un objectif 93x dont la distance de travail était de 300 μm. Les images obtenues ont ensuite été déconvoluées à l’aide du logiciel de traitement d’images Huygens. Barre d’échelle = 10 μm, et 5 μm pour les images agrandies (zoom). L’intensité de fluorescence a été mesurée le long de la ligne indiquée en jaune, depuis la sphère jusqu’à l’extérieur de la cellule, et le résultat de cette mesure est représenté en B. C. Pourcentage des cellules non polarisées et polarisées présentant une accumulation de la nucléoprotéine NP à la membrane plasmique.** indique une différence statistiquement significative (p < 0,01). D. Images représentatives, en microscopie électronique à transmission, d’une coupe ultrafine de cellules Caco-2/TC7 polarisées sur billes Cytodex 3, puis infectées pendant 8 heures par la souche A/WSN/33 (avec une multiplicité d’infection = 10 PFU/cellule), ou non infectées. Barre d’échelle = 0,5 μm. Certaines structures membranaires du réticulum endoplasmique (RE) qui ont été mesurées (voir en E) sont surlignées en jaune à titre d’exemple. E. Quantification de la longueur des structures membranaires du RE dans des cellules non-infectées (NI) ou infectées (I). *** indique une différence statistiquement significative (p < 0,001). F. Classification des structures membranaires du RE mesurées en (E) selon leur ordre de longueur, dans les cellules polarisées non infectées (NI) ou infectées (I) par le virus. |
Dans un second temps, nous avons cherché à déterminer si, comme constaté précédemment dans des cellules non polarisées, l’infection virale induisait un remaniement des membranes du réticulum endoplasmique [11]. Des cellules Caco-2/TC7 polarisées sur Cytodex ont été infectées, fixées 8 heures après, et analysées par microscopie électronique à transmission. Nous avons constaté un remaniement beaucoup plus marqué dans les cellules polarisées, avec notamment la présence de longues structures membranaires issues du réticulum endoplasmique, localisées à la périphérie cellulaire et organisées parallèlement à la membrane plasmique (Figure 2D). Cette différence phénotypique entre cellules polarisées et non polarisées a été quantifiée en mesurant la longueur des membranes du réticulum endoplasmique (Figure 2E) et en les classant selon leur taille [9] (Figure 2F).
Perspectives
En résumé, nous avons mis au point un système maniable, rapide et peu coûteux permettant l’établissement d’un épithélium polarisé susceptible à l’infection par le virus influenza A/WSN/33. En appliquant à ce système diverses méthodes de microscopie optique et électronique, nous avons montré que certaines altérations cellulaires consécutives à l’infection grippale préalablement identifiées dans des modèles cellulaires traditionnels, à savoir le détournement de la petite GTPase RAB11 et la perturbation de l’organisation des membranes du réticulum endoplasmique, existent également dans les cellules épithéliales polarisées. Singulièrement, dans ces cellules qui en principe reproduisent plus fidèlement l’organisation intracellulaire des cellules cibles naturelles du virus, le remodelage des membranes du réticulum endoplasmique paraît exacerbé, et l’adressage des génomes viraux à la membrane plasmique est plus efficace. Des mécanismes moléculaires de transport particuliers semblent donc être mis en jeu. D’autres travaux de recherche devraient permettre de les caractériser, et d’évaluer si l’utilisation du système de cellules Caco-2/TC7 polarisées sur billes Cytodex peut être étendue à d’autres virus respiratoires, tels que le virus respiratoire syncytial.
Remerciements
Nous remercions Christophe Chehade (UMR144, Institut Curie, Paris, France) pour son aide à la représentation schématique des cellules polarisées sur billes Cytodex 3.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Krammer F, Smith GJD, Fouchier RAM, et al. Influenza. Nat Rev Dis Primers 2018 ; 4 : 3. [CrossRef] [PubMed] [Google Scholar]
- Webby RJ, Uyeki TM. An update on highly pathogenic avian Influenza A (H5N1) virus, clade 2.3.4.4b. J Infect Dis 2024 ; 230 : 533–42. [CrossRef] [PubMed] [Google Scholar]
- Fourriere L, Jimenez AJ, Perez F, et al. The role of microtubules in secretory protein transport. J Cell Sci 2020 ; 133 : jcs237016. [CrossRef] [PubMed] [Google Scholar]
- Grigoriev I, Splinter D, Keijzer N, et al. Rab6 regulates transport and targeting of exocytotic carriers. Dev Cell 2007 ; 13 : 305–14. [CrossRef] [Google Scholar]
- Brault J, Bardin S, Lampic M, et al. RAB6 and dynein drive post-Golgi apical transport to prevent neuronal progenitor delamination. EMBO Reports 2022 ; 23 : e54605. [CrossRef] [Google Scholar]
- Pizzorno A, Padey B, Dulière V, et al. Interactions between severe acute respiratory syndrome coronavirus 2 replication and major respiratory viruses in human nasal epithelium. J Infect Dis 2022 ; 226 : 2095–104. [CrossRef] [PubMed] [Google Scholar]
- Postal BG, Aguanno D, Thenet S, et al. Rapid evaluation of intestinal paracellular permeability using the human enterocytic-like Caco-2/TC7 cell line. In : Turksen K, editor. Permeability Barrier. Methods in molecular biology. New York, NY : Springer US, 2021 : 13–26. [CrossRef] [PubMed] [Google Scholar]
- Jakob PH, Kehrer J, Flood P, et al. A 3-D cell culture system to study epithelia functions using microcarriers. Cytotechnology 2016 ; 68 : 1813–25. [CrossRef] [Google Scholar]
- Brault J-B, Thouvenot C, Cannata Serio M, et al. A polarized cell system amenable to subcellular resolution imaging of influenza virus infection. PLoS One 2024 ; 19 : e0292977. [CrossRef] [PubMed] [Google Scholar]
- Amorim MJ. A comprehensive review on the interaction between the host GTPase Rab11 and Influenza A virus. Front Cell Dev Biol 2019 ; 6 : 176. [CrossRef] [PubMed] [Google Scholar]
- De Castro Martin IF, Fournier G, Sachse M, et al. Influenza virus genome reaches the plasma membrane via a modified endoplasmic reticulum and Rab11-dependent vesicles. Nat Commun 2017 ; 8 : 1396. [CrossRef] [PubMed] [Google Scholar]
© 2025 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1 Polarisation des cellules Caco-2/TC7 cultivées sur billes de dextran Cytodex 3. A. Représentation schématique de l’organisation du cytosquelette de microtubules dans des cellules non polarisées et dans des cellules épithéliales polarisées. B. Représentation schématique de cellules épithéliales cultivées en trois dimensions (3D) sur billes Cytodex 3. C. Aspect columnaire des cellules Caco-2/TC7 après deux semaines de culture sur billes Cytodex 3. Barre d’échelle = 100 µm. D. Image, par microscopie confocale, de cellules Caco-2/TC7 polarisées sur Cytodex 3. Un anticorps dirigé contre PALS1 marque la membrane cytoplasmique apicale (en vert). L’agent intercalant DAPI marque l’ADN (en bleu). Barre d’échelle = 25 µm. E. Image de microscopie électronique à transmission d’une coupe ultrafine de cellules Caco-2/TC7 polarisées sur Cytodex 3. Différents compartiments cellulaires sont indiqués par des flèches noires : villosités, jonction intercellulaire serrée, appareil de Golgi, réticulum endoplasmique, mitochondrie. Barre d’échelle = 1 µm F. Titrage de la quantité de particules virales infectieuses présentes dans le milieu de culture à 24, 48 et 72 heures post-infection (hpi) par la souche A/WSN/33 (avec une multiplicité d’infection = 0,01 PFU/cellule), en présence ou en absence de l’inhibiteur de la protéase virale Baloxavir (0,1 µM). La ligne en trait pointillé correspond à la limite de détection : 250 PFU/mL. |
| Dans le texte | |
 |
Figure 2 Imagerie des cellules Caco-2/TC7 cultivées sur billes Cytodex 3 et infectées avec le virus influenza A/WSN/33. A. Des cellules Caco-2/TC7 cultivées sur billes Cytodex 3 ont été infectées par la souche A/WSN/33 du virus influenza (avec une multiplicité d’infection = 10 PFU/cellule). Au bout de 8 heures, elles ont été fixées, puis immuno-marquées avec des anticorps dirigés contre la nucléoprotéine NP (en rouge) et contre RAB11 (en cyan), avant d’être visualisées par microscopie confocale avec un objectif 93x dont la distance de travail était de 300 μm. Les images obtenues ont ensuite été déconvoluées à l’aide du logiciel de traitement d’images Huygens. Barre d’échelle = 10 μm, et 5 μm pour les images agrandies (zoom). L’intensité de fluorescence a été mesurée le long de la ligne indiquée en jaune, depuis la sphère jusqu’à l’extérieur de la cellule, et le résultat de cette mesure est représenté en B. C. Pourcentage des cellules non polarisées et polarisées présentant une accumulation de la nucléoprotéine NP à la membrane plasmique.** indique une différence statistiquement significative (p < 0,01). D. Images représentatives, en microscopie électronique à transmission, d’une coupe ultrafine de cellules Caco-2/TC7 polarisées sur billes Cytodex 3, puis infectées pendant 8 heures par la souche A/WSN/33 (avec une multiplicité d’infection = 10 PFU/cellule), ou non infectées. Barre d’échelle = 0,5 μm. Certaines structures membranaires du réticulum endoplasmique (RE) qui ont été mesurées (voir en E) sont surlignées en jaune à titre d’exemple. E. Quantification de la longueur des structures membranaires du RE dans des cellules non-infectées (NI) ou infectées (I). *** indique une différence statistiquement significative (p < 0,001). F. Classification des structures membranaires du RE mesurées en (E) selon leur ordre de longueur, dans les cellules polarisées non infectées (NI) ou infectées (I) par le virus. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




