| Numéro |
Med Sci (Paris)
Volume 41, Numéro 6-7, Juin-Juillet 2025
|
|
|---|---|---|
| Page(s) | 540 - 542 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2025091 | |
| Publié en ligne | 7 juillet 2025 | |
Édition génique par transfert de complexes ribonucléoprotéiques de CRISPR/Cas9 dans la rétine
Gene editing by direct delivery of Cas9 protein in the retina
Sorbonne Université, Inserm, CNRS, UMRS968, Institut de la vision, Paris, France
Les dystrophies rétiniennes héréditaires représentent une cause majeure de cécité. Elles sont souvent dues à la mutation d’un gène affectant les cellules photoréceptrices. Les stratégies de thérapie génique actuelles sont fondées sur une supplémentation génique, consistant à introduire une version fonctionnelle du gène défectueux via un vecteur viral. Le succès de Luxturna, première thérapie génique approuvée pour traiter une maladie rétinienne héréditaire, a montré le potentiel de ces stratégies de supplémentation génique. Cependant, elles peuvent difficilement être utilisées lorsque le gène thérapeutique est trop grand pour rentrer dans un vecteur viral adéquat. En outre, elles sont adaptées pour traiter les maladies récessives, qui sont dues à une « perte de fonction », mais elles sont insuffisantes dans le cas des maladies dominantes pour lesquelles le gène altéré produit une protéine toxique. Une désactivation ou modification du gène muté est alors nécessaire pour prévenir la dégénérescence rétinienne. Depuis son développement et sa consécration par le prix Nobel de chimie en 2020, la méthode CRISPR (clustered regularly interspaced short palindromic repeat)/Cas9 s’est imposée comme un outil d’édition génomique polyvalent, facile d’utilisation et très efficace, capable d’introduire des modifications précises dans l’ADN. L’outil CRISPR/Cas9 est composé d’une protéine enzymatique Cas9 qui, grâce à un petit ARN guide (ARNg), va couper la molécule d’ADN à un endroit précis. Puis, des mécanismes endogènes vont permettre de réparer la cassure de l’ADN, ce qui peut entraîner de petits défauts de la séquence nucléotidique appelés indels (insertions ou délétions). Dérivée de la méthode CRISPR/Cas9, « l’édition de base » (en anglais, base editing) permet de corriger spécifiquement un nucléotide sans cassure de l’ADN : les « éditeurs de base » ont donc la capacité de directement corriger la mutation responsable de la maladie. Toutefois, l’utilisation thérapeutique de la méthode CRISPR/Cas9 ou de méthodes qui en sont dérivées [1] (→) soulève des défis considérables, notamment en termes de génotoxicité. En effet, des mésappariements de l’ARNg à une région de l’ADN non ciblée peuvent entraîner des modifications indésirables de l’ADN, telles que des indels ou des réarrangements chromosomiques [2, 3]. Du fait de l’origine bactérienne de CRISPR/Cas9, son utilisation comporte également un risque d’induire une réponse immunitaire. Il est donc primordial de choisir un vecteur, ou véhicule, qui permettra de transporter correctement ces outils vers les cellules cibles, afin de limiter les risques thérapeutiques, tout en maintenant une forte efficacité d’édition de l’ADN.
(→) Voir m/s n° 10, 2024, page 748
Le vecteur le plus utilisé pour le transfert de matériel génétique dans la rétine est le virus adéno-associé (AAV), qui permet une expression prolongée du matériel génétique transporté dans les cellules cibles. Bien que souhaitable pour une supplémentation génique, cette expression prolongée devient problématique avec CRISPR/Cas9, en raison du risque accru de modifications « hors cible » de l’ADN, de réarrangements génomiques, et d’insertion du génome de l’AAV dans celui de la cellule hôte [4]. Un transfert transitoire permettrait de limiter la durée d’activité de l’outil, et donc de limiter les effets génotoxiques indésirables. Le transfert de CRISPR/Cas9 sous forme d’ARN messager (ARNm) n’a pour l’instant montré qu’une efficacité très limitée dans l’épithélium pigmentaire rétinien, tissu de soutien des cellules photoréceptrices, et s’est avéré inefficace pour cibler ces cellules [5, 6]. Une alternative prometteuse réside dans le transfert transitoire de complexes ribonucléoprotéiques (RNP), composés de la protéine Cas9 et d’un ARNg déjà fonctionnels, et qui sont dégradés dans les cellules après quelques jours [7]. Cependant, cette technique n’avait encore jamais été utilisée dans les cellules photoréceptrices de la rétine.
Nous avons montré que ces complexes RNP de CRISPR/Cas9 ou d’éditeur de base, injectés directement dans l’espace sous-rétinien chez la souris, permettent une édition efficace et ciblée du génome des cellules photoréceptrices de la rétine (Figure 1A) [8]. Les taux d’édition lorsqu’on cible un gène fortement exprimé dans ces cellules, Sag (S-antigen / visual arrestin), atteignent 10 % d’allèles édités, des performances comparables à celles des vecteurs viraux. De plus, nous avons analysé les principaux effets « hors-cible » de ce transfert de RNP en comparaison avec le transfert par AAV. Alors que la cible indésirable considérée comme la plus à risque in silico présentait un faible taux d’édition génique avec CRISPR/Cas9 AAV, aucun effet hors cible n’a au contraire été détecté avec les RNP.
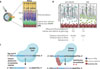 |
Figure 1 Transfert dans la rétine de complexes ribonucléoprotéiques contenant CRISPR/Cas9 ou un éditeur de base. A. Schéma de l’injection sous-rétinienne des complexes ribonucléoprotéiques (RNP) et des vecteurs AAV. Les différentes couches de la rétine, incluant les cellules photoréceptrices (photorécepteurs) et l’épithélium pigmentaire rétinien (EPR), sont représentées. OS (outer segment) : segment externe des cellules photoréceptrices ; ONL (outer nuclear layer) : couche constituée par les noyaux des cellules photoréceptrices. B. Comparaison entre les vecteurs AAV et les complexes RNP. Les AAV (représentés par les petits hexagones gris) ont une diffusion étendue et permettent une présence prolongée de l’outil d’édition, mais avec, de ce fait, un risque accru de modifications « hors cible » de l’ADN. Les RNP (représentés en vert), ont une diffusion plus limitée, et offrent un profil de sécurité amélioré grâce à une activité intracellulaire transitoire (quelques jours) de l’outil d’édition. C. Mécanismes d’action de CRISPR/Cas9 et des éditeurs de bases (ABE) sur le gène Sag. L’outil CRISPR/Cas9 induit des cassures double-brin de l’ADN, qui sont réparées par NHEJ (non-homologous end joining), entraînant des insertions/délétions qui inactivent le gène. |
Nous avons également montré que des barrières physiques, telles que la membrane limitante externe1 et la densité des segments externes des cellules photoréceptrices, semblent freiner la diffusion des RNP de CRISPR/Cas9 dans les noyaux de ces cellules. En effet, les RNP semblent bloqués au niveau des segments externes de ces cellules, qui sont de longs prolongements constitués par un empilement de disques de phospholipides essentiel au processus de phototransduction impliqué dans la vision (Figure 1B).
Pour surmonter cet obstacle au transfert des RNP dans le noyau des cellules photoréceptrices, l’utilisation de vecteurs non viraux a été testée, incluant celle de vecteurs lipidiques comme la Lipofectamine 2000 ou d’un peptide pénétrant (SP10). Aucun de ces vecteurs n’a montré d’amélioration significative de l’efficacité d’édition par rapport aux RNP sans vecteur. De plus, les vecteurs lipidiques ont entraîné des signes de toxicité, incluant une dégénérescence localisée des cellules photoréceptrices. Ces résultats soulignent l’importance de développer des vecteurs non viraux adaptés, capables de surmonter les barrières physiques de la rétine tout en minimisant la toxicité et la dose injectée.
Nous avons testé l’efficacité de différents ARNg pour plusieurs gènes cibles, dont Sag et Vegfa (Vacular endothelial growth factor A). Les résultats indiquent que l’efficacité d’édition dépend du niveau d’expression génique dans le tissu ciblé. Par exemple, un ARNg ciblant Sag, qui est exprimé dans les cellules photoréceptrices, a permis d’obtenir un taux d’édition de ce gène jusqu’à 25 % dans ces cellules, mais de seulement 2,5 % dans celles de l’épithélium pigmentaire. Inversement, le taux d’édition avec un ARNg ciblant Vegfa, qui est exprimé dans les cellules de l’épithélium pigmentaire mais pas dans les cellules photoréceptrices, était plus élevé dans les premières que dans les secondes. Ce constat suggère que le niveau d’expression des gènes dans les types cellulaires cibles doit être pris en compte lors de la conception des ARNg. Toutefois, la séquence nucléotidique de l’ARNg semble être le facteur principal de l’efficacité. En effet, un seul des trois ARNg ciblant le gène Sag que nous avons testés s’est avéré efficace, ce qui n’était pas prévisible d’après les prédictions in silico et les résultats in vitro sur des lignées cellulaires humaines. Le transfert d’éditeurs de bases a également été évalué pour leurs capacités à induire des substitutions nucléotidiques précises sans introduire de cassure double brin, réduisant ainsi le risque de mutations « hors cible » ou de réarrangements génomiques (Figure 1C) [3]. Un taux de substitution ciblée atteignant 10,7 % a été observé pour le gène Sag, sans substitution à côté de la cible, ni effet hors cible. Ces résultats confirment que le transfert transitoire de RNP est applicable à plusieurs outils d’édition génique, y compris les plus récents.
En conclusion, le transfert direct de complexes RNP de CRISPR/Cas9 ou d’éditeurs de bases constitue une avancée majeure pour la thérapie génique des dystrophies rétiniennes héréditaires. Cette méthode permet un transfert transitoire, d’une durée de quelques jours, des outils d’édition génique, ce qui réduit le risque de modifications « hors cible » de l’ADN associé à une présence prolongée de ces outils, tout en maintenant une efficacité d’édition comparable à celle des vecteurs AAV. Cependant, des défis subsistent, notamment en termes de diffusion des complexes RNP dans les cellules photoréceptrices et de la nécessité de minimiser les doses pour limiter les effets toxiques. Ces obstacles pourraient être surmontés par des innovations dans la conception des vecteurs non viraux. À plus long terme, l’utilisation des complexes RNP pour transférer les différents outils d’édition génique aux cellules cibles pourrait permettre d’élargir le spectre des maladies traitables par thérapie génique.
Liens d’intérêt
Le Dr Dalkara est co-inventeur du brevet numéro 9193956 (« Adeno-associated virus virions with variant capsid and methods of use thereof »), avec des redevances versées à Adverum Biotechnologies et sur les demandes de brevets en attente pour des méthodes non invasives visant les photorecepteurs à cône (EP17306429.6 et EP17306430.4) sous licence à Gamut Tx, devenu maintenant SparingVision. Le Dr Deniz Dalkara a également des intérêts financiers personnels dans Tenpoint Tx. et SparingVision, en dehors de la portée du travail soumis.
Références
- Bouchard C, Godbout K et Tremblay J P. La correction de mutations pathogènes par Prime editing. Med Sci (Paris) 2024 ; 40 : 748–56. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Botto C, Rucli M, Tekinsoy MD, et al. Early and late stage gene therapy interventions for inherited retinal degenerations. Prog Retin Eye Res 2021 ; 100975. [PubMed] [Google Scholar]
- Pulman J, Sahel J-A, Dalkara D. New editing tools for gene therapy in inherited retinal dystrophies. CRISPR J 2022 ; 5 : 377–88. [CrossRef] [Google Scholar]
- Hanlon KS, Kleinstiver BP, Garcia SP, et al. High levels of AAV vector integration into CRISPR-induced DNA breaks. Nat Commun 2019 ; 10 : 4439. [PubMed] [Google Scholar]
- Herrera-Barrera M, Ryals RC, Gautam M, et al. Peptide-guided lipid nanoparticles deliver mRNA to the neural retina of rodents and nonhuman primates. Sci Adv 2023 ; 9 : eadd4623. [CrossRef] [PubMed] [Google Scholar]
- Gautam M, Jozic A, Su GL-N, et al. Lipid nanoparticles with PEG-variant surface modifications mediate genome editing in the mouse retina. Nat Commun 2023 ; 14 : 6468. [CrossRef] [PubMed] [Google Scholar]
- Kim K, Park SW, Kim JH, et al. Genome surgery using Cas9 ribonucleoproteins for the treatment of age-related macular degeneration. Genome Res 2017 ; 27 : 419–26. [CrossRef] [PubMed] [Google Scholar]
- Pulman J, Botto C, Malki H, et al. Direct delivery of Cas9 or base editor protein and guide RNA complex enables genome editing in the retina. Mol Ther Nucleic Acids 2024 ; 102349. [CrossRef] [PubMed] [Google Scholar]
© 2025 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1 Transfert dans la rétine de complexes ribonucléoprotéiques contenant CRISPR/Cas9 ou un éditeur de base. A. Schéma de l’injection sous-rétinienne des complexes ribonucléoprotéiques (RNP) et des vecteurs AAV. Les différentes couches de la rétine, incluant les cellules photoréceptrices (photorécepteurs) et l’épithélium pigmentaire rétinien (EPR), sont représentées. OS (outer segment) : segment externe des cellules photoréceptrices ; ONL (outer nuclear layer) : couche constituée par les noyaux des cellules photoréceptrices. B. Comparaison entre les vecteurs AAV et les complexes RNP. Les AAV (représentés par les petits hexagones gris) ont une diffusion étendue et permettent une présence prolongée de l’outil d’édition, mais avec, de ce fait, un risque accru de modifications « hors cible » de l’ADN. Les RNP (représentés en vert), ont une diffusion plus limitée, et offrent un profil de sécurité amélioré grâce à une activité intracellulaire transitoire (quelques jours) de l’outil d’édition. C. Mécanismes d’action de CRISPR/Cas9 et des éditeurs de bases (ABE) sur le gène Sag. L’outil CRISPR/Cas9 induit des cassures double-brin de l’ADN, qui sont réparées par NHEJ (non-homologous end joining), entraînant des insertions/délétions qui inactivent le gène. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




