| Issue |
Med Sci (Paris)
Volume 40, Number 8-9, Août-Septembre 2024
Les microbes, l’Anthropocène et nous
|
|
|---|---|---|
| Page(s) | 654 - 660 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2024101 | |
| Published online | 20 September 2024 | |
Le microbiome humain : 340 ans d’histoire, 140 ans d’interrogations, d’innovations technologiques et émergence de la « médecine microbienne »
The human microbiome: 340 years of history, 140 years of interrogations, technological innovations and emergence of “microbial medicine”
1
Université Paris-Saclay, INRAE, MetaGenoPolis, AgroParisTech, MICALIS, Jouy-en-Josas, France
2
Institut Pasteur, Paris
3
Collège de France, Paris
*
philippe.sansonetti@pasteur.fr
On sait depuis 340ans que le corps humain héberge des microbes, alors appelés « animalcules ». Depuis plus d’un siècle, après la démonstration de la responsabilité de certains de ces microbes dans les maladies, on s’interroge sur le rôle de ceux – largement majoritaires – qui colonisent les surfaces cutanées et muqueuses humaines, particulièrement le riche écosystème microbien de l’intestin, le microbiote intestinal. De l’invention de la vie sans germe (axénie), qui a permis de valider expérimentalement la symbiose entre êtres humains et microbes, fruit d’une longue coévolution, à la mise au point des méthodes de culture anaérobies, puis à l’invention du diagnostic moléculaire, du séquençage profond ouvrant les approches métagénomiques et omiques en général, une formidable course s’est déroulée entre innovations technologiques et avancées conceptuelles. Cette course, au-delà de la description exhaustive du microbiote dans sa diversité intraet interhumaine, des fonctions symbiotiques essentielles du microbiome, a ouvert la voie d’un nouveau domaine de la médecine : la médecine microbienne.
Abstract
For 350 years, we have known that the human body hosts microbes, then called “animalcules”. For over a century, following the demonstration of the role of some of these microbes in diseases, questions have arisen about the role of the largely predominant ones colonizing human skin and mucous surfaces, particularly the rich microbial ecosystem of the intestine, the gut microbiota. From the invention of germ-free life – axenism – which experimentally validated the human-microbe symbiosis, resulting from a long coevolution, to the development of anaerobic culture methods, then to the invention of molecular diagnosis, deep sequencing opening up metagenomic and omics approaches in general, a remarkable race has taken place between technological innovations and conceptual advances. This race, beyond the exhaustive description of the microbiota in its intra- and inter-human diversity, and the essential symbiotic functions of the microbiome, has paved the way for a new field of medicine: microbial medicine.
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Vignette (© Philippe Sansonetti).
Dans le grand récit écologique actuel, associé au constat des effets désastreux des changements climatiques, la perte de biodiversité apparaît comme un thème majeur, mais porte essentiellement sur le monde macroscopique. Le monde des microbes, qui représente 95 % de la biodiversité du vivant, est régulièrement occulté. Il semble pourtant qu’au temps de l’anthropocène – terme encore discuté par les géologues [35] (→) – , la diversité microbienne, en particulier bactérienne, tende à se réduire avec des conséquences encore difficiles à évaluer. Qu’en est-il du microbiome humain ? C’est chez l’homme que cette évolution est la plus patente et que les conséquences sur la santé de la « paucibiose »1 et des « dysbioses »2 qu’elle entraîne sont le mieux étudiées [1].
(→) Voir Les mots de la science, page 671 de ce numéro
Une brève histoire de « l’invention » du microbiome
C’est vers la fin du XVIIe siècle qu’en même temps que les microbes fut découvert le microbiote. On sous-estime volontiers les technologies en regard des intuitions fulgurantes comme moteurs de la découverte, mais qui aurait pu imaginer l’existence d’un monde microscopique sans microscope ? Dès 1683, à Delft, Antonie van Leeuwenhoek, grâce au premier microscope à lumière directe (Figure 1), découvre, sur des échantillons de salive et de tartre dentaire, des êtres inconnus qu’il appelle « animalcules » (Figure 2). Deux siècles plus tard, la communauté scientifique s’empare des animalcules, les baptise « microbes », les cultive, et réalise l’étendue de leurs activités. Les travaux de Louis Pasteur et de son école sur la fermentation, débouchent alors sur la théorie infectieuse des maladies. Les maladies de cette époque étaient en effet essentiellement infectieuses et en l’espace de quatre décennies, de 1870 à 1910, la plupart des microbes pathogènes responsables des grandes endémies furent découverts, particulièrement les pathogènes bactériens et parasitaires, comme le paludisme. Robert Koch et ses disciples s’interrogèrent alors sur l’établissement des liens de causalité entre microbe et maladie, proposant, en 1884, les « postulats de Koch »3. Mais on s’interroge simultanément sur la présence et le rôle, chez l’homme et chez l’animal, d’une diversité de microbes à l’évidence non pathogènes. Dès 1885, Theodor Escherich, pédiatre et microbiologiste allemand, s’étonnait de « l’apparition au hasard de bactéries au sein de selles et d’un tube digestif sains, une situation semblant créée par un millier de coïncidences ». Il exprimait sa conviction que « la connaissance précise de ces conditions permettrait non seulement d’en comprendre le rôle physiologique, mais aussi la pathologie et le traitement des infections intestinales » [2, 3]. En 1907, Elie Metchnikoff, dans son troisième ouvrage La prolongation de la vie, proposait la théorie du « pourrissement intérieur » selon laquelle les bactéries productrices de toxines de la flore intestinale concouraient au processus de vieillissement. Ayant observé que la population bulgare, qui consommait une grande quantité de produits à base de lait fermenté, avait le taux le plus élevé de centenaires, il proposa que les lactobacilles, dont Lactobacillus bulgaricus, pouvaient inverser le processus, et inventa le concept de « probiotique ». Dès 1909, Arthur I Kendall, aux États-Unis, identifiant dans les urines certains métabolites caractéristiques de la combinaison d’un régime alimentaire et de certaines espèces bactériennes, proposait de rechercher les bactéries générant des métabolites bioactifs particuliers. Ainsi naissait, il y a un siècle, la « métabolomique » et le concept de « postbiotiques » (Kendall AI. Some observations on the study of the intestinal bacteria. J Biol Chem 1909 ; 6 : 499-507. Reproduit dans [4]).
 |
Figure 1. Premier microscope à éclairage direct utilisé par Antonie van Leeuwenhoek à Delft à la fin du XVIIe siècle. |
 |
Figure 2. Première observation d’« animalcules » par Antonie van Leeuwenhoek au sein d’une suspension aqueuse d’un échantillon de tartre dentaire humain. Croquis illustrant ses rapports à la Royal Society de Londres (Collected Letters, vol. IV, Pl. X ; Gemeentelijke Archiefdiens). On y reconnaît facilement la forme des grandes familles bactériennes (cocci, bacilles, spirilles, etc.). À noter des trajectoires curvilignes pointillées, essentielles car illustrant le fait que ces objets étaient animés, donc vivants, d’où le nom d’animalcules, peutêtre plus évocateur que celui de « microbes » qu’on leur donna deux siècles plus tard… |
Axénie : du concept à la pratique
En 1885, Louis Pasteur et Émile Duclaux avaient posé la question, pour eux essentielle, du rôle de la flore commensale humaine. Le premier s’interrogeait sur « […] l’intérêt qu’il y aurait à nourrir un jeune animal, dès sa naissance, avec […] des produits alimentaires qu’on priverait artificiellement et complétement des microbes communs ». Pasteur concluait : « Sans vouloir rien affirmer, je ne cache pas que j’entreprendrais cette étude, si j’en avais le temps, avec la pensée préconçue que la vie, dans ces conditions, deviendrait impossible »4. Envisageant la possibilité technique de l’axénie (l’élevage d’animaux sans germes), Pasteur envisageait donc que le monde microscopique était indispensable à la vie macroscopique. Douze ans plus tard, George Nuttal et Hans Thier-Felder, à l’Université de Göttingen, mirent au point un modèle d’isolateur extraordinaire de complexité et de sophistication pour l’époque (Figure 3), et démontrèrent que l’absence de flore commensale n’affectait ni la digestion, ni le développement, ni la survie de cobayes axéniques [5–7]. Dans les décennies qui suivirent, la « gnotoxénie » (vie en environnement microbien contrôlé) se développa, en particulier à l’université Notre-Dame (Indiana, États-Unis). James Reyniers, Philippe Trexler et Bob Ervin développèrent, dans les années 1940, une gamme d’enceintes axéniques adaptées à différentes espèces animales, dont les enceintes d’aujourd’hui sont héritières (Figure 4). Cette « vie sans microbes » fascina les esprits.
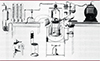 |
Figure 3. Première enceinte anaérobie conçue par Nuttal et Thier-Felder à Göttingen en 1897. Un bijou de Deutsche Technologie. |
 |
Figure 4. Système d’enceinte pour l’élevage de souris axéniques utilisé aujourd’hui. |
Ce succès technologique ouvrit alors des opportunités pour la médecine de pointe requérant des conditions de contrôle strict de l’environnement microbien de certains patients afin de prévenir des infections gravissimes du fait de fonctions immunitaires déficientes [8]. Cependant, le suivi prolongé d’animaux axéniques montra qu’ils étaient hypotrophiques car malnutris. Ils étaient en effet incapables d’hydrolyser les polyosides alimentaires, dont la fermentation bactérienne est une source calorique et nutritionnelle considérable, sous forme d’acides gras à chaînes courtes, comme le butyrate. Ils étaient aussi sévèrement carencés en certaines vitamines (A, B12, K). Dans les élevages axéniques, il fallait donc compenser ces manques [9] (→).
(→) Voir la Synthèse de A. El Kaoutari et al., m/s n° 3, mars 2014, page 259
La gnotobiologie, l’étude des organismes exempts de germes, a permis de déchiffrer la logique des « successions écologiques », la construction d’un microbiote mature grâce à des interactions multiples entre micro-organismes. Dans les années 1960-1970, Pierre Raibaud et Robert Ducluzeau, à l’INRA de Jouy en Josas, lancèrent les modèles murins gnotoxéniques5 pour étudier les étapes de la maturation du microbiote intestinal [10].
La comparaison de données obtenues avec des modèles axéniques et avec des modèles conventionnels (pourvus d’un microbiote) s’est avérée cruciale pour identifier les grandes fonctions du microbiote. Ces comparaisons ont révélé et précisé son implication dans des processus physiologiques fondamentaux, tels que la digestion des fibres alimentaires et la production de métabolites, et aussi la fonction d’exclusion compétitive de microorganismes intrus, éventuellement pathogènes, aussi qualifiée d’effet de barrière, et bien d’autres fonctions encore, toutes liées aux interactions fonctionnelles entre le microbiote et son hôte.
La gnotobiologie a connu un essor spectaculaire avec les travaux de l’équipe de Jeff Gordon (université de Washington, St Louis, États-Unis), à partir des années 1990. Ils ont en effet mis en évidence l’importance des premiers stades de colonisation par le microbiote intestinal dans la programmation à long terme de la santé métabolique et immunitaire, ouvrant de nouvelles voies pour la compréhension des maladies liées aux altérations du microbiote. Ils ont montré, par exemple, le rôle majeur de la flore intestinale dans l’angiogenèse [11]. Ce groupe et d’autres ont également montré le rôle fondamental du microbiote intestinal dans la maturation du système immunitaire muqueux et systémique [12]. Rapidement fut aussi révélé le rôle de la flore intestinale dans la maturation post-natale du système nerveux central, d’où la proposition d’un nouveau concept, l’« axe intestin-cerveau » [13].
Vers un catalogue de l’écosystème microbien intestinal
À cette flore symbiotique, il fallait donner un nom et une composition. Pour la flore intestinale, on s’aperçut très tôt qu’elle était dominée par des espèces anaérobies ne prospérant qu’en l’absence d’oxygène et avec un potentiel redox très négatif. Theodor Rosebury, à l’université de Washington (St Louis, États-Unis), s’inscrit à un double titre dans cette histoire. Du début des années 1940 à 1962, ce spécialiste de microbiologie orale s’attela à la culture et à l’identification des principales espèces de la flore intestinale humaine [14]. Dans son ouvrage au titre évocateur, Life on man, il qualifia la flore microbienne humaine de « microbial biota », terme qui, par fusion, donna « microbiote » (→).
(→) Voir le numéro thématique « Le microbiote : cet inconnu qui réside en nous », m/s n° 11, volume 32, novembre 2016
« Microbiome », souvent utilisé actuellement, recouvre le microbiote en tant que collection d’espèces et les fonctions biologiques qu’elles offrent dans la symbiose établie avec l’hôte. Rosebury insistait sur la dominance des anaérobies stricts, tels Bacteroides, et des anaérobies métaboliques, comme Lactobacillus, mais son catalogue resta limité, de nombreuses espèces étant impossible à cultiver. Robert E Hungate, à l’université d’Austin au Texas, puis à Stanford en Californie, opéra un bond technologique avec le « Hungate roll tube », qui permit d’isoler et de cultiver les microorganismes responsables de la lyse du cellulose dans le rumen, et d’inaugurer la culture des archées productrices de méthane [15]. Le rumen, avec sa diversité microbienne, a inspiré des études pionnières sur les interactions entre bactéries anaérobies, archées méthanogènes et animaux hôtes, qui ont fondé les sciences microbiomiques, dès le début des années 1970.
« Great plate count anomaly » et naissance de la microbiologie moléculaire
La confrontation entre microscopie et culture anaérobie mit en évidence le « great plate count anomaly »6, à savoir qu’on ne peut cultiver au laboratoire qu’une partie des microorganismes impliqués dans des écosystèmes complexes : longtemps à peine quelques pourcents pour le sol et 20 à 30 % pour l’intestin humain. Même si l’amélioration des techniques de culture anaérobie augmenta significativement ces performances, elles laissaient un déficit d’identification préoccupant.
Dans les années 1970, Carl Woese (université de l’Illinois, Urbana-Champaign, États-Unis) proposa une identification phylogénétique bactérienne fondée sur les ARN des ribosomes (ARNr) présents dans tout le spectre vivant. Une petite sous-unité, l’ARNr 16S, dont la séquence est hautement conservée chez les bactéries, mais présentant des variations suffisantes détectables par hybridation croisée (sémantides), permet de distinguer différents genres, voire différentes espèces. Carl Woese put ainsi reconstruire les parentés phylogénétiques, confirmant l’indépendance du groupe des archées, et permettant l’identification des bactéries endosymbiotiques7, strictement incultivables du fait de la perte de larges portions de leurs génomes au cours de leur co-évolution intracellulaire avec des organismes eucaryotes. Dès lors, une nouvelle classification reposa sur la séquence de l’ARNr 16S des bactéries. Cette classification phylogénétique révéla ainsi des branches de l’arbre du vivant, dont certaines étaient encore inaccessibles par la culture [16]. L’écologie moléculaire, discipline nouvelle utilisant directement les ADN ou ARN bactériens, permit d’apprécier la diversité et la complexité des communautés microbiennes. Dès le milieu des années 1980, David A Stahl (université de Washington, Seattle, États-Unis) exploita l’ARNr 16S pour étudier les communautés microbiennes du rumen [17, 18], complétant ainsi les travaux de Hungate [15].
Vers une connaissance exhaustive de la composition du microbiote intestinal humain : de la métataxonomie 16S à la métagénomique du « second génome humain »
Il s’est agi, jusqu’à présent, d’analyses microbiologiques moléculaires appliquées à des écosystèmes microbiens environnementaux et des microbiotes animaux. L’application de la méthode métataxonomique 16S à l’ADN extrait de selles humaines permit de compléter la connaissance du microbiote intestinal humain, dont Joël Doré et David Relman en révélèrent la diversité [19]. Les méthodes se simplifièrent avec les progrès du séquençage permettant, après amplification par PCR (polymerase chain reaction), puis clonage de séquences variables, suivi de séquençage, de mettre en évidence, de manière exhaustive, la diversité microbienne d’un contenu intestinal humain. Joël Doré en décrivit les particularités dans la maladie de Crohn, une maladie inflammatoire chronique de l’intestin, en particulier l’absence de Faecalibacterium prauznizii, une espèce constante et dominante au sein du microbiote intestinal humain [20]. David Relman, quant à lui, en étudia les variations en réponse à des antibiotiques [21].
Révolution métagénomique et émergence de la « médecine microbienne »
La métagénomique représente un « saut quantique » dans la connaissance et la compréhension des fonctions symbiotiques du microbiome humain.
Contrairement à la génomique classique, qui se concentre sur l’ADN des organismes individuels, la métagénomique permet d’étudier l’équipement génétique de tous les microorganismes présents dans un écosystème. Le séquençage à haut débit de fragments courts, appliqué à toute l’information génétique accessible, a été une avancée majeure dans l’étude du microbiome humain. Elle a permis de reconstruire le catalogue des gènes et des génomes et a offert une vision plus complète des interactions entre les espèces bactériennes (Figure 5) et des voies métaboliques, des cibles immunitaires, des neurorécepteurs éventuellement impliqués, ainsi que des changements associés à divers états de santé ou de maladie.
 |
Figure 5. Pipeline de l’analyse métagénomique d’un écosystème microbien complexe. |
Deux projets majeurs, MetaHit (Metagenomics of the Human Intestinal Tract) en Europe, et le Human Microbiome Project (HMP) aux États-Unis, ont jeté, à partir de 2008, les bases de la métagénomique pour l’étude du microbiome humain.
MetaHit
MetaHit explorait le microbiote intestinal humain à une échelle sans précédent, grâce à des techniques de séquençage de nouvelle génération pour analyser les échantillons provenant de milliers de volontaires, cartographiant ainsi la diversité microbienne dans différentes populations [22] et identifiant des associations avec des maladies, telles que l’obésité, le diabète et les maladies inflammatoires intestinales [23].
Le HMP
Le HMP, des National Institutes of Health (NIH), avait pour objectif de caractériser le microbiome humain dans différentes parties du corps : la peau, la bouche, le tractus gastro-intestinal et le système reproducteur. Le projet a produit une immense quantité de données servant de référence pour le microbiome humain [24, 25].
Ces études métagénomiques ont ainsi révélé des différences marquées dans la composition du microbiote entre individus, mais aussi des similitudes fondamentales au niveau des fonctions métaboliques essentielles. L’inattendu a été la description de quelques grandes organisations écologiques des microbiotes humains, comme les entérotypes8 [26], dont les limites réelles restent à préciser, et l’observation d’une hétérogénéité dans la richesse en gènes des microbiotes avec, parfois même, une paucibiose [27], que l’on relie aujourd’hui à un risque accru de maladies chroniques. Ces travaux ont inauguré les recherches sur les mécanismes participant aux variations du microbiome, et sur la façon dont ces variations peuvent influencer les maladies humaines et la métagénomique est devenue incontournable pour l’étude du microbiome humain. Grâce aux techniques de séquençage de nouvelle génération, on obtient des analyses plus rapides, plus précises et… à moindre coût.
Les approches multi-omiques, combinant génomique, transcriptomique, protéomique et métabolomique, offrent aujourd’hui une vue d’ensemble plus complète du microbiome et de ses potentiels fonctionnels. L’étude des inter-microbiomes, c’est-à-dire des interactions entre les microbiomes peuplant différentes parties du corps humain, permet d’étudier leur influence sur la santé et la maladie.
Les applications médicales de la métagénomique vont du diagnostic précoce de certaines maladies, à la prédiction des réponses à certains traitements, fondé sur des signatures microbiennes, jusqu’à des ouvertures en prévention et en thérapeutique par restauration de l’intégrité du microbiote et de l’harmonie de la symbiose [28].
Cette « médecine microbienne » pourrait transformer la compréhension et le traitement d’une large gamme de maladies, de l’obésité aux troubles gastro-intestinaux, en passant par les maladies autoimmunes, neurodégénératives et les troubles neuropsychiatriques. Méfions-nous cependant d’un enthousiasme naïf. Des études en population se multiplient à travers le monde, visant aujourd’hui des centaines de milliers d’individus, comme le « microbiote français » (French Gut)9 et le « Million Microbiomes from Humans Project »10 à l’échelle internationale. La globalisation des données est indispensable et doit porter sur diverses régions et populations de la planète, afin d’apprécier la richesse et de la diversité du microbiote humain. Ces programmes s’inscriront dans le cadre « Une Seule Santé » (One Health), appelant un suivi similaire des autres écosystèmes animaux et végétaux [29]. Il est temps que la biodiversité microbienne s’inscrive dans la pensée écologique.
Des modéles informatiques peuvent aider la compréhension des interactions complexes au sein du microbiome. Le concept de « guildes microbiennes » désigne des réseaux d’interactions entre différentes espèces, qui conditionnent la robustesse de l’écosystème [31]. À un niveau supérieur, la modélisation est applicable à la relation de symbiose établie entre microbiome et hôte. Des boucles de rétroaction relient notamment les altérations du microbiote : la perméabilité intestinale, le statut immuno-inflammatoire et le stress oxydant. Cela a conduit à montrer la discontinuité entre les états de santé et de maladie, et le potentiel basculement brutal dans un contexte pathologique durable entretenu par un cercle vicieux [32–34]. Une symbiose altérée pourrait caractériser un grand nombre de maladies chroniques dont l’incidence ne cesse d’augmenter depuis presque trois générations11. Selon l’Organisation mondiale de la santé (OMS), on parle de maladies épidémiques non-transmissibles post-modernes. La symbiose semble aussi pouvoir moduler les réponses aux thérapies nutritionnelles ou médicamenteuses, notamment l’immunité. Sous réserve de l’établissement du lien de causalité entre dysbiose et maladie, objet du prochain article de cette série thématique de médecine/sciences, la prise en compte de l’importance de la qualité de l’interface entre l’être humain et son microbiome pourrait amener – dans certaines maladies – à réviser significativement diagnostic, prévention, voire traitement. Les outils de cette « médecine microbienne » en devenir sont prêts à être transférés au laboratoire de biologie clinique, compte tenu de la « démocratisation » rapide de l’accès aux outils de séquençage de nouvelle génération et de la bioinformatique.
Le statut microbien de l’humain nous inscrit dans un nouvel univers, celui de l’holobionte12 qui intègre la structure complexe de l’humain-microbien qui conditionnerait jusqu’à l’évolution de l’espèce.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Les auteurs tiennent à remercier très chaleureusement Anna Salvetti pour ses encouragements, ses suggestions et son soutien à toutes les étapes de l’écriture de cet article.
Le micro-organisme doit être présent en abondance dans tous les organismes souffrant de la maladie, mais absent des organismes sains.
Le micro-organisme doit pouvoir être isolé et croître en milieu de culture pur (ne contenant que ce seul microbe).
Le micro-organisme cultivé doit déclencher la même maladie chez un animal de laboratoire sensible.
Le micro-organisme doit être à nouveau isolé du nouvel organisme hôte rendu malade puis identifié comme étant identique à l’agent infectieux original.
Références
- Dieterich W, Schink M, Zopf Y. Microbiota in the Gastrointestinal Tract. Med Sci (Basel) 2018 ; 6. [PubMed] [Google Scholar]
- Escherich T. Die Darmbakterien des Neugeborenen und Sauglings. Fortschr Med 1885 ; 3 : 515–22. [Google Scholar]
- Escherich T. Die Darmbakterien des Neugeborenen und Sauglings. Fortschr Med 1885 ; 3 : 547–57. [Google Scholar]
- Aziz RK. A hundred-year-old insight into the gut microbiome! Gut Pathog 2009 ; 1h 21. [CrossRef] [PubMed] [Google Scholar]
- Nuttal GHF, Thierfelder H. Thierisches Leben ohne Bakterien im Vergaundskanal. Hoppe-Seyl Z Physiol Chem 1897 ; 21 : 109–21. [Google Scholar]
- Nuttal GHF, Thierfelder H. Thierisches Leben ohne Bakterien im Vergaundskanal. . Hoppe-Seyl Z Physiol Chem 1897 ; 22 : 62–73. [CrossRef] [Google Scholar]
- Nuttal GHF, Thierfelder H. Thierisches Leben ohne Bakterien im Vergaundskanal. Hoppe-Seyl Z Physiol Chem 1897 ; 23 : 213-34, 1895-6, 6–7. [Google Scholar]
- Reyniers JA, Trexler PC, et al. A complete life-cycle in the germ-free bantam chicken. Nature 1948 ; 162 : 67. [PubMed] [Google Scholar]
- El Kaoutari A, Armougom F, Raoult D, Henrissat B. Le microbiote intestinal et la digestion des polysaccharides. Med Sci (Paris) 2014 ; 30 : 259–65. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Raibaud P, Ducluzeau R. [Etude de la colonisation bacterienne du tractus gastro-intestinal a l’aide de modeles experimentaux]. Rev Sci Tech 1989 ; 8 : 361–73. [CrossRef] [PubMed] [Google Scholar]
- Stappenbeck TS, Hooper LV, Gordon JI. Developmental regulation of intestinal angiogenesis by indigenous microbes via Paneth cells. Proc Natl Acad Sci U S A 2002 ; 99 : 15451–5. [CrossRef] [PubMed] [Google Scholar]
- Chung H, Pamp SJ, Hill JA, et al. Gut immune maturation depends on colonization with a host-specific microbiota. Cell 2012 ; 149 : 1578–93. [CrossRef] [PubMed] [Google Scholar]
- Diaz Heijtz R, Wang S, Anuar F, et al. Normal gut microbiota modulates brain development and behavior. Proc Natl Acad Sci U S A 2011 ; 108 : 3047–52. [CrossRef] [PubMed] [Google Scholar]
- Rosebury T. Microorganisms indigenous to man. New York : McGraw-Hill, 1962. [Google Scholar]
- Hungate RE. Symposium: selected topics in microbial ecology. I. Microbial ecology of the rumen. Bacteriol Rev 1960 ; 24 : 353–64. [CrossRef] [PubMed] [Google Scholar]
- Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. Proc Natl Acad Sci U S A 1977 ; 74 : 5088–90. [CrossRef] [PubMed] [Google Scholar]
- Stahl DA, Flesher B, Mansfield HR, Montgomery L. Use of phylogenetically based hybridization probes for studies of ruminal microbial ecology. Appl Environ Microbiol 1988 ; 54 : 1079–84. [CrossRef] [PubMed] [Google Scholar]
- Amann RI, Binder BJ, Olson RJ, et al. Combination of 16S rRNA-targeted oligonucleotide probes with flow cytometry for analyzing mixed microbial populations. Appl Environ Microbiol 1990 ; 56 : 1919–25. [CrossRef] [PubMed] [Google Scholar]
- Suau A, Bonnet R, Sutren M, et al. Direct analysis of genes encoding 16S rRNA from complex communities reveals many novel molecular species within the human gut. Appl Environ Microbiol 1999 ; 65 : 4799–807. [CrossRef] [PubMed] [Google Scholar]
- Manichanh C, Rigottier-Gois L, Bonnaud E, et al. Reduced diversity of faecal microbiota in Crohn’s disease revealed by a metagenomic approach. Gut 2006 ; 55 : 205–11. [CrossRef] [PubMed] [Google Scholar]
- Dethlefsen L, Huse S, Sogin ML, Relman DA. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing. PLoS Biol 2008 ; 6 : e280. [CrossRef] [PubMed] [Google Scholar]
- Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature 2010 ; 464 : 59–65. [CrossRef] [PubMed] [Google Scholar]
- Le Chatelier E, Nielsen T, Qin J, et al. Richness of human gut microbiome correlates with metabolic markers. Nature 2013 ; 500 : 541–6. [CrossRef] [PubMed] [Google Scholar]
- Grice EA, Segre JA. The human microbiome: our second genome. Annu Rev Genomics Hum Genet 2012 ; 13 : 151–70. [CrossRef] [PubMed] [Google Scholar]
- Human Microbiome Project C. Structure, function and diversity of the healthy human microbiome. Nature 2012 ; 486 : 207–14. [CrossRef] [PubMed] [Google Scholar]
- Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human gut microbiome. Nature 2011 ; 473 : 174–80. [CrossRef] [PubMed] [Google Scholar]
- Cotillard A, Kennedy SP, Kong LC, et al. Dietary intervention impact on gut microbial gene richness. Nature 2013 ; 500 : 585–8. [CrossRef] [PubMed] [Google Scholar]
- Dore J, Multon MC, Behier JM, participants of Giens Xxxii RTN. The human gut microbiome as source of innovation for health: Which physiological and therapeutic outcomes could we expect? Therapie 2017 ; 72 : 21–38. [CrossRef] [PubMed] [Google Scholar]
- Banerjee S, van der Heijden MGA. Soil microbiomes and one health. Nat Rev Microbiol 2023 ; 21 h 6–20. [CrossRef] [PubMed] [Google Scholar]
- Anand S, Hallsworth JE, Timmis J, et al. Weaponising microbes for peace. Microb Biotechnol 2023 ; 16 : 1091–111. [CrossRef] [PubMed] [Google Scholar]
- Wu G, Zhao N, Zhang C, et al. Guild-based analysis for understanding gut microbiome in human health and diseases. Genome Med 2021 ; 13 h 22. [CrossRef] [PubMed] [Google Scholar]
- van de Guchte M, Blottiere HM, Dore J. Humans as holobionts: implications for prevention and therapy. Microbiome 2018 ; 6 : 81. [CrossRef] [PubMed] [Google Scholar]
- Van de Guchte M, Burz SD, Cadiou J, et al. Alternative stable states in the intestinal ecosystem: proof of concept in a rat model and a perspective of therapeutic implications. Microbiome 2020 ; 8 : 153. [CrossRef] [PubMed] [Google Scholar]
- van de Guchte M, Mondot S, Dore J. Dynamic Properties of the Intestinal Ecosystem Call for Combination Therapies, Targeting Inflammation and Microbiota, in Ulcerative Colitis. Gastroenterology 2021 ; 161 : 1969-81 e12. [CrossRef] [PubMed] [Google Scholar]
- Gilgenkrantz H. Anthropocène. Med Sci (Paris) 2024 ; 40 : ???. [Google Scholar]
Liste des figures
 |
Figure 1. Premier microscope à éclairage direct utilisé par Antonie van Leeuwenhoek à Delft à la fin du XVIIe siècle. |
| Dans le texte | |
 |
Figure 2. Première observation d’« animalcules » par Antonie van Leeuwenhoek au sein d’une suspension aqueuse d’un échantillon de tartre dentaire humain. Croquis illustrant ses rapports à la Royal Society de Londres (Collected Letters, vol. IV, Pl. X ; Gemeentelijke Archiefdiens). On y reconnaît facilement la forme des grandes familles bactériennes (cocci, bacilles, spirilles, etc.). À noter des trajectoires curvilignes pointillées, essentielles car illustrant le fait que ces objets étaient animés, donc vivants, d’où le nom d’animalcules, peutêtre plus évocateur que celui de « microbes » qu’on leur donna deux siècles plus tard… |
| Dans le texte | |
 |
Figure 3. Première enceinte anaérobie conçue par Nuttal et Thier-Felder à Göttingen en 1897. Un bijou de Deutsche Technologie. |
| Dans le texte | |
 |
Figure 4. Système d’enceinte pour l’élevage de souris axéniques utilisé aujourd’hui. |
| Dans le texte | |
 |
Figure 5. Pipeline de l’analyse métagénomique d’un écosystème microbien complexe. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




