| Issue |
Med Sci (Paris)
Volume 40, Number 3, Mars 2024
|
|
|---|---|---|
| Page(s) | 251 - 257 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2024012 | |
| Published online | 22 March 2024 | |
Un mécanisme temporel pour la génération de la diversité neuronale
A temporal mechanism for the generation of neuronal diversity
Université Paris Cité, CNRS UMR7592, Institut Jacques Monod, Paris, France
L’un des plus grands défis des neurosciences est de comprendre comment une structure complexe, telle que le cerveau, se construit. L’encodage spatial et temporel des progéniteurs neuronaux permet la génération de l’essentiel de la diversité neuronale. Cette revue se concentre sur l’expression séquentielle de facteurs de transcription temporels, qui modifie la capacité des cellules souches à générer différents types de neurones et qui est conservée chez plusieurs espèces animales. Des publications récentes ont permis, en particulier, une compréhension fine de ce processus au cours du développement du système visuel de la drosophile, en éclairant la manière dont il contribue à la spécification de diverses identités neuronales. Le système visuel des insectes constitue un modèle unique pour étudier l’évolution des mécanismes neurodéveloppementaux qui génèrent la diversité neuronale.
Abstract
One of the greatest challenges in neuroscience is to understand how a complex structure, such as the brain, is built. Spatial and temporal patternings of neuronal progenitors are responsible for the generation of most of the neuronal diversity observed in the brain. This review focuses on the temporal patterning of neuronal progenitors, i.e. the sequential expression of transcription factors that changes the capacity of stem cells to generate different neuronal types, and which is conserved in animals. Recent papers have offered a near complete understanding of the mechanism of temporal patterning in the developing visual system of Drosophila, and of how this contributes to the specification of diverse neuronal identities, which are then maintained by the sustained expression of downstream transcription factors. The insect visual system provides a unique model to study the evolution of neuronal cell types, as well as the evolution of neurodevelopmental mechanisms that generate them.
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Vignette (© Nikolaos Konstantinides).
Chez les animaux, le système nerveux offre une complexité inégalée dans les autres organes du corps. Le cerveau humain, qui est une des structures neuronales animales les plus complexes, se compose d’environ cent milliards de neurones qui forment des milliers de milliards de synapses [1]. Cette structure complexe doit se développer dans chaque embryon pour générer des individus fonctionnels. Ainsi, des erreurs commises au cours de ce processus vont entraîner des troubles neurodéveloppementaux délétères. Comprendre le développement du cerveau humain est donc une condition sine qua non pour prévenir ou traiter ces troubles. Néanmoins, sa complexité et la difficulté d’y accéder ne permettent pas l’étude directe des processus neurodéveloppementaux. Malgré des efforts considérables, il reste difficile d’appréhender l’ampleur de la diversité existante et, plus important encore, la manière dont cette diversité est générée ou dont les circuits fonctionnels sont organisés. Il est donc nécessaire d’établir des modèles d’étude alternatifs.
L’étude de structures neuronales à la fois complexes et génétiquement accessibles, telles que le cerveau de la mouche, du poisson-zèbre ou de la souris, a permis des avancées considérables. Le cerveau de ces animaux, bien que beaucoup plus simple que celui de l’homme, est constitué de centaines de milliers, voire de millions de neurones qui forment des circuits complexes. Cela est à l’origine d’un large éventail de comportements qui permettent aux animaux de survivre dans leur environnement. Il est important de noter que les mécanismes régulant la génération de la diversité neuronale et la formation des circuits neuronaux semblent être conservés entre les invertébrés et les vertébrés, ce qui rend l’utilisation de modèles invertébrés d’autant plus pertinente [2, 3].
Les mécanismes à l’origine de la diversité neuronale
Quatre mécanismes sont à l’origine de la diversité cellulaire dans les différents systèmes neuronaux des animaux :
L’encodage spatial (spatial patterning) différencie les cellules en division provenant de différents domaines progéniteurs. Un exemple d’encodage spatial chez les vertébrés est celui des progéniteurs neuronaux situés le long de l’axe dorso-ventral du tube neural au cours du développement. Des gradients opposés de Wnt et de BMP (bone morphogenetic protein) provenant de la plaque du toit du tube neural, d’une part, et de Sonic Hedgehog (Shh) provenant de la plaque du plancher, d’autre part, conduisent ainsi à générer des domaines de progéniteurs spatialement distincts qui donneront naissance à des types neuronaux très différents [4] (Figure 1A).
 |
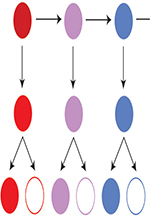
Figure 1. Encodage spatial et temporel dans le système visuel en développement de la drosophile. A. Encodage spatial dans la moelle épinière des vertébrés (à gauche). Chaque domaine spatial (DS) est défini par l’expression de gènes distincts le long de l’axe dorso-ventral, grâce à des gradients de concentration opposés de Shh et de Wnt/BMP. Chez la drosophile (à droite), la compartimentation en 8 domaines spatiaux du neuroépithélium de l’OPC ajoute un niveau de complexité et de complétude à la différenciation neuronale. B.Encodage temporel dans les neuroblastes de l’OPC chez la drosophile. À chaque fenêtre d’expression temporelle, le neuroblaste se divise asymétriquement pour se renouveler et donner naissance à une cellule mère ganglionnaire (CMG). Chaque fenêtre d’expression temporelle conduit à la génération d’un type de cellule neuronale spécifique. C. Destin binaire Notch-dépendant des neurones. Les CMG se divisent de manière asymétrique pour générer deux neurones, un NotchOFF et un NotchON. Les cellules barrées indiquent la mort cellulaire. FT : facteur de transcription. |
L’encodage temporel (temporal patterning) des progéniteurs neuronaux permet de générer une descendance dont le destin varie en fonction de leur âge [5]. Ce changement de destin est généralement déterminé par l’expression temporelle d’une série de facteurs de transcription, et fera l’objet de cette revue (Figure 1B).
Les progéniteurs intermédiaires multipotents peuvent générer différentes descendances neuronales grâce à une décision binaire du destin cellulaire exercée par la voie Notch. Cette différence de destin est décidée par la présence ou l’absence d’activité Notch (NotchON/NotchOFF) [6] (Figure 1C).
Enfin, la mort cellulaire programmée, modulée au cours du développement, élimine les neurones post-mitotiques au cours de leur différenciation pour sculpter finement des réseaux neuronaux fonctionnels [7].
L’encodage temporel chez les insectes et les vertébrés
Nous nous concentrerons ici sur l’encodage temporel des progéniteurs neuronaux, décrit pour la première fois dans les neuroblastes embryonnaires de la corde nerveuse ventrale (VNC pour ventral nerve cord) de la drosophile [8, 9]. Les neuroblastes sont les cellules souches neuronales du système nerveux de la drosophile, et constituent un modèle d’étude classique qui a permis des avancées majeures dans notre compréhension des mécanismes de la diversité neuronale. Les neuroblastes se divisent de manière asymétrique1, ce qui permet à chacun d’entre eux de générer plusieurs neurones et cellules gliales. Au fur et à mesure que les neuroblastes vieillissent, ils expriment séquentiellement une série de facteurs de transcription temporels (FTt) : Hunchback (Hb) → Krüppel → Pdm → Castor → Grainy head. Cela permet de générer des progéniteurs neuronaux spécifiques de chacune des différentes fenêtres temporelles. Il a depuis été montré que dans d’autres tissus, les neuroblastes expriment également des séries de FTt, dont l’identité varie d’une structure à l’autre [10].
Chez les vertébrés, les facteurs temporels intrinsèques identifiés lors de la neurogenèse sont souvent homologues à ceux trouvés chez la drosophile. Ikaros, qui est l’orthologue du FTt précoce Hb dans la VNC de la mouche, est également exprimé précocement dans les progéniteurs corticaux et rétiniens de la souris, et il spécifie le destin des neurones précoces dans les deux tissus [11, 12]. Pou2f2 et Casz1, qui sont respectivement les orthologues, chez les vertébrés, des FTt plus tardifs chez la mouche, Pdm et Castor, sont exprimés dans les progéniteurs rétiniens plus âgés, spécifiant le destin des neurones tardifs [13–15]. Les séries temporelles du tube neural et du cerveau des vertébrés sont constituées de FTt très différents [16]. Il est intéressant de noter que cette série de facteurs de transcription est exprimée spatialement dans l’embryon de drosophile au stade blastoderme [17], ce qui a des implications sur l’évolution de l’encodage spatio-temporel [18]. Les FTt permettent ainsi de générer une diversité neuronale à la fois chez les insectes et chez les vertébrés.
Le système dans lequel l’encodage temporel a été décrit de la manière la plus détaillée est la partie visuelle du cerveau larvaire de la drosophile [19, 20] (Figure 2). La grande majorité des neurones du système visuel adulte se développe au cours du troisième stade larvaire, à partir d’une structure neuroépithéliale appelée centre externe de prolifération (OPC pour outer proliferation center). À la fin du deuxième stade larvaire, les cellules du bord médian de l’OPC se transforment en neuroblastes au cours d’une vague pro-neurale qui se déplace. Ces neuroblastes se divisent de manière asymétrique afin de s’auto-renouveler d’une part, et de générer un précurseur intermédiaire (appelé cellule mère ganglionnaire ou CMG) d’autre part. La CMG se divise une seule fois pour générer deux cellules filles (neurones et/ou cellules gliales), qui diffèrent par la présence ou l’absence de l’activité Notch [21] (Figure 2).
 |
Figure 2. Modèle schématique de la neurogenèse dans la médulla. Les cellules du neuroépithélium (NE, carrés gris) se convertissent progressivement en neuroblastes (NB, grands cercles). Ces derniers se divisent de manière asymétrique pour générer des cellules mères ganglionnaires (CMG), qui se divisent en neurones. Au fur et à mesure que les NB de la médulla vieillissent, ils expriment séquentiellement une série de facteurs de transcription conduisant à la genèse d’une colonne de neurones d’identités différentes (petits cercles). Cette différenciation se produit médialement vers le côté latéral du lobe optique. Les neurones NotchON sont représentés par des cercles pleins, tandis que les neurones NotchOFF sont représentés par des cercles vides. Les flèches en pointillés représentent le sens de la vague neuronale. hth : homothorax ; ey : eyeless ; slp : sloppy-paired ; D : Dichaete ; tll : tailless. |
En vieillissant, les neuroblastes expriment une séquence de facteurs de transcription temporels donnant lieu à la production de types neuronaux distincts. Initialement, une série de cinq facteurs de transcription temporels avait été identifiée [19] : Homothorax (Hth), Eyeless (Ey), Sloppy-paired (Slp), Dichaete (D) et Tailless (Tll) (Figure 1B), qui sont exprimés successivement dans les neuroblastes vieillissants, permettant la génération de divers types de neurones. Vingt heures après la formation de la pupe2, 40 000 neurones et plus de 100 types cellulaires auront été générés, dont la plupart deviendront des neurones du cortex médullaire. Cependant, certains facteurs de transcription manquaient à cette série temporelle pour expliquer qu’une telle diversité neuronale soit générée.
Le séquençage de l’ARNm de cellule unique et l’inférence de trajectoire pour identifier les FTt
Alors que la plupart des FTt ont été identifiés à l’aide de l’immunohistochimie, les progrès de la technique de séquençage des ARNm de cellule unique ont offert de nouvelles possibilités [22] pour l’identification de nouveaux FTt. Deux études publiées récemment [20, 23] ont ainsi utilisé cette technique de séquençage de l’ARNm de cellule unique suivi d’une inférence de trajectoire3 pour identifier l’intégralité des FTt dans les neuroblastes de l’OPC. En raison de la progression de la vague pro-neurale dans l’OPC, le cerveau de la larve de troisième stade tardif contient simultanément des neuroblastes d’âges différents. En utilisant le séquençage de l’ARNm de cellule unique à un temps donné, les auteurs ont ainsi pu identifier le transcriptome des neuroblastes à différents stades de leur développement. Des outils d’inférence de trajectoire sont ensuite capables de détecter les modifications mineures dans le transcriptome qui se produisent au cours de la différenciation des neuroblastes afin d’ordonner ces cellules en fonction de leur âge. Ces outils ont permis d’identifier tous les gènes (et en particulier ceux codant les facteurs de transcription) exprimés dans les neuroblastes au cours du développement du cerveau larvaire. Ces résultats fournissent donc un répertoire de gènes potentiellement impliqués dans la capacité des neuroblastes à générer différents types de neurones. Enfin, les FTt identifiés in silico ont été validés in vivo à l’aide d’outils génétiques disponibles chez la drosophile, de l’immunohistochimie et de l’hybridation in situ.
Notons que le séquençage de l’ARNm de cellule unique a été utilisé avec succès dans d’autres systèmes neuronaux, tels que la moelle épinière en développement de la souris [24].
Comment se déroule la série temporelle ?
L’expression précise et successive des facteurs de transcription temporels est cruciale pour la genèse de l’ensemble neuronal approprié. Comment cette progression est-elle assurée ? Dans le lobe optique en développement de la drosophile, la plupart des facteurs de transcription sont non seulement exprimés de manière temporelle, mais ils participent également à un réseau complexe de régulation génique qui permet à la série temporelle de progresser (Figure 3A). Dans un premier temps, il avait été montré que chaque FTt active le suivant et inhibe le précédent [19]. Cependant, des expériences plus récentes indiquent que le réseau de régulation des gènes est en fait plus complexe [20, 23]. Tous les FTt ne fonctionnent pas de la même manière lors de la progression de la cascade temporelle. Certains, tels que Eyeless et Dichaete, sont régulés par deux FTt différents (respectivement Opa/Erm et Slp/Scro). Opa régule la temporalité de l’expression de Ey plutôt que son expression. Le fait que l’intégralité de la série FTt soit achevée en environ 20 heures suggère que les FTt se lient directement aux régions des enhancers des FTt qu’ils régulent. La nature complexe du réseau de régulation génique de la série temporelle est probablement un produit du processus évolutif qui a conduit à ce dernier (Figure 3A).
 |
Figure 3. Interactions génétiques et série temporelle finale lors du développement du lobe optique de la larve de drosophile. A. Interactions génétiques entre les FTt précédemment identifiés et les nouveaux FTt identifiés dans les neuroblastes de la médulla. B. Schéma représentant les séries temporelles mises à jour dans les neuroblastes du centre externe de prolifération (OPC), les cellules mères ganglionnaires (CMG) et le destin Notch des neurones, ainsi que le nom des neurones générés par chaque fenêtre temporelle (identité du neurone). hth : homothorax ; opa : odd paired ; erm : earmuff ; ey : eyeless ; hbn : homeobrain ; scro : scarecrow ; B-H1 : BarH1 ; slp : sloppy-paired ; D : Dichaete ; tll : tailless. |
Ce réseau forme une horloge purement transcriptionnelle qui permet à la série de progresser et de s’achever au cours de la vie d’un neuroblaste. On ne sait cependant toujours pas comment cette horloge transcriptionnelle est synchronisée avec le cycle cellulaire des neuroblastes. De plus, si cette horloge peut être opérationnelle dans des cellules souches neuronales à courte durée de vie et à division rapide, il est difficile d’imaginer comment elle pourrait fonctionner dans des cellules souches qui se divisent une fois par jour (comme la glie radiale de la souris) ou même une fois par semaine (glie radiale de l’homme). Néanmoins, un mode de régulation similaire a été identifié dans les cellules progénitrices rétiniennes de souris, où Ikzf1 établit la fenêtre temporelle précoce avant de réguler l’expression de Pou2f1, qui à son tour active Pou2f2 et empêche l’expression du facteur temporel moyen/tardif Casz1 [14].
Comment la série temporelle contribue-t-elle à la genèse de différents types neuronaux ?
Chez la drosophile, les FTt que nous avons mentionnés définissent au moins onze fenêtres temporelles d’expression différentes, qui génèrent des neurones (et des cellules gliales) différents, en raison de la superposition de leur expression. Au fur et à mesure qu’ils sont générés, les neurones nouvellement nés déplacent les neurones générés précédemment en les éloignant du neuroblaste parent (c’est-à-dire de la surface du cerveau). Ce processus crée une disposition en colonnes des corps cellulaires dans le cortex de la médulla représentant ainsi leur ordre de naissance (Figure 2). Les neurones nés dans chaque fenêtre temporelle expriment des effecteurs en aval des FTt (par exemple Bsh, Runt [Run] et Vvl) appelés « gènes concentriques » en raison de leur patron d’expression [25]. Sur la base de l’expression de ces gènes dans les différents types de neurones, l’ordre de naissance de tous les neurones de la médulla a pu être établi (Figure 3B). Cette analyse a aussi montré que les neurones NotchON sont toujours cholinergiques, sauf dans la fenêtre temporelle impliquant D/Bar-H1, où ils sont glutamatergiques ou dopaminergiques. Les neurones NotchOFF sont, quant à eux, GABAergiques lorsqu’ils sont générés au cours des fenêtres temporelles précoces, et glutamatergiques lorsqu’ils sont générés au cours des fenêtres temporelles plus tardives. Une exception notable est la fenêtre temporelle D/Bar-H1, où l’inverse se produit, les neurones NotchON étant alors glutamatergiques ou dopaminergiques et les neurones NotchOFF cholinergiques [20].
De nombreux gènes effecteurs en aval des FTt restent exprimés pendant toute la durée de vie des neurones, définissant leur identité [26], ce qui fournit un bon modèle pour étudier la manière dont des destins neuronaux spécifiques sont déclenchés et maintenus dans les neurones post-mitotiques. Le concept de détermination de l’identité neuronale par un groupe limité de facteurs de transcription maintenus au cours du temps a été décrit à la fois chez Caenorhabditis elegans [27, 28] et chez la drosophile [29]. Le modèle d’expression de ces facteurs de transcription concentriques dans les neurones rappelle la localisation des différents neurones dans les différentes couches du cortex des vertébrés, où leur ordre de naissance est également représenté de l’intérieur vers l’extérieur [2]. Il est donc probable que la compréhension des mécanismes conduisant à la détermination de l’identité neuronale chez les invertébrés contribuera à comprendre comment est générée la diversité neuronale dans le cerveau des mammifères.
Un modèle uniforme pour générer de la diversité
Comme nous l’avons déjà mentionné, l’encodage spatio-temporel, associé à une décision binaire de l’état de Notch (NotchON et NotchOFF) sur le devenir des cellules est le principal facteur de l’identité neuronale. En effet, dans le système visuel de la drosophile, l’intégration des onze fenêtres temporelles distinctes (dont dix génèrent des neurones) avec huit domaines spatiaux et la décision binaire de Notch peut expliquer la genèse d’environ 160 types de cellules (10 × 8 × 2), ce qui est proche de la diversité totale des types de neurones de la médulla de la drosophile.
Une question qui reste en suspens est de savoir comment les informations spatio-temporelles sont intégrées par les progéniteurs neuronaux. Différents modèles peuvent être envisagés :
La coopération directe : les facteurs spatio-temporels pourraient coopérer directement en formant un complexe et en régulant les gènes en aval, y compris les régulateurs post-mitotiques.
La mémoire épigénétique : dans les cas où les facteurs spatiaux et temporels ne sont pas exprimés dans les mêmes cellules (par exemple, dans le système visuel de la drosophile, où les facteurs spatiaux sont exprimés dans l’épithélium tandis que les facteurs temporels sont exprimés dans les neuroblastes), les facteurs spatiaux pourraient modifier le paysage chromatinien et définir un sous-ensemble de cibles potentielles, qui varie selon le domaine où est produit le type neuronal, et qui pourra être activé par les facteurs temporels qui sont exprimés ultérieurement.
Le relais régulateur : enfin, les facteurs spatiaux qui sont exprimés plus tôt pourraient activer un facteur en aval qui serait exprimé de manière persistante et qui coopérerait directement avec les facteurs temporels pour activer un ensemble spécifique de régulateurs post-mitotiques [30].
La compréhension de la manière dont les facteurs spatio-temporels interagissent afin d’encoder les identités neuronales maintenues par la présence de facteurs de transcription exprimés continuellement en aval, permettra de comprendre la manière dont un système neuronal complexe se construit.
Évolution de la diversité neuronale
Malgré les progrès considérables réalisés dans notre compréhension du développement des structures neuronales complexes, nous avons très peu de connaissances sur l’évolution de ces structures. Cette lacune pourrait être comblée par l’utilisation des techniques de séquençage de l’ARNm de cellules uniques qui permettent aujourd’hui non seulement d’identifier et de comparer la composition des types cellulaires de n’importe quel tissu neuronal chez n’importe quel animal auquel nous avons accès au niveau transcriptomique, mais aussi d’étudier les mécanismes neurodéveloppementaux à l’origine de cette diversité [31].
Le système visuel des insectes représente un modèle idéal pour de telles études [32]. La nature répétitive des neuropiles4 du lobe optique, qui se traduit par des types cellulaires comprenant un grand nombre de cellules, facilite leur regroupement et leur annotation par séquençage sur cellules uniques. La publication récente d’un atlas cellulaire détaillé et annoté dans les lobes optiques de la drosophile adulte, ainsi qu’au cours de son développement, fournit une base parfaite pour les comparaisons et les transferts d’annotation à venir entre différentes espèces [26, 33]. La détermination de l’origine développementale (spatiale et temporelle) de tous les types de cellules (comme décrit précédemment) facilitera également l’identification des types cellulaires homologues, car on s’attend à ce que les types de cellules homologues proviennent du même lignage chez différents animaux. Bien qu’il s’agisse d’une structure relativement simple (comparée au cerveau central), le lobe optique de la drosophile est suffisamment complexe, composé d’environ 200 types de neurones, qui peuvent être comparés à ceux d’autres insectes et révéler ainsi des histoires évolutives différentes.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Nous remercions Marianne Malartre et Félix Simon pour leurs commentaires sur le manuscrit. Nos travaux sur le développement et l’évolution de la diversité neuronale et de l’encodage temporel sont soutenus par le European Research Council (ERC) dans le cadre du programme de recherche et d’innovation Horizon 2020 de l’Union européenne (convention de subvention n° 949 500).
Références
- Azevedo FAC, Carvalho LRB, Grinberg LT, et al. Equal numbers of neuronal and nonneuronal cells make the human brain an isometrically scaled-up primate brain. J Comp Neurol 2009 ; 513 : 532–541. [CrossRef] [PubMed] [Google Scholar]
- Holguera I, Desplan C. Neuronal specification in space and time. Science 2018 ; 362 : 176–180. [CrossRef] [PubMed] [Google Scholar]
- Sanes JR, Zipursky SL. Design Principles of Insect and Vertebrate Visual Systems. Neuron 2010 ; 66 : 15–36. [CrossRef] [PubMed] [Google Scholar]
- Wilson L, Maden M. The mechanisms of dorsoventral patterning in the vertebrate neural tube. Dev Biol 2005 ; 282 : 1–13. [CrossRef] [PubMed] [Google Scholar]
- Rossi AM, Fernandes VM, Desplan C. Timing temporal transitions during brain development. Curr Opin Neurobiol 2017 ; 42 : 84–92. [CrossRef] [PubMed] [Google Scholar]
- Bertet C, Li X, Erclik T, et al. Temporal patterning of neuroblasts controls Notch-mediated cell survival through regulation of Hid or Reaper. Cell 2014 ; 158(1): 173–186. [Google Scholar]
- Pinto-Teixeira F, Konstantinides N, Desplan C. Programmed cell death acts at different stages of Drosophila neurodevelopment to shape the central nervous system. FEBS Lett 2016 ; 590(2): 435–453. [Google Scholar]
- Brody T, Odenwald WF. Programmed transformations in neuroblast gene expression during Drosophila CNS lineage development. Dev Biol 2000 ; 226 : 34–44. [CrossRef] [PubMed] [Google Scholar]
- Isshiki T, Pearson B, Holbrook S, Doe CQ. Drosophila neuroblasts sequentially express transcription factors which specify the temporal identity of their neuronal progeny. Cell 2001 ; 106 : 511–521. [CrossRef] [PubMed] [Google Scholar]
- Mira H, Morante J. Neurogenesis From Embryo to Adult - Lessons From Flies and Mice. Front Cell Dev Biol 2020; 8. [PubMed] [Google Scholar]
- Alsiö JM, Tarchini B, Cayouette M, et al. Ikaros promotes early-born neuronal fates in the cerebral cortex. Proc Natl Acad Sci USA 2013 ; 110 : E716–E725. [Google Scholar]
- Elliott J, Jolicoeur C, Ramamurthy V, Cayouette M. Ikaros confers early temporal competence to mouse retinal progenitor cells. Neuron 2008 ; 60 : 26–39. [CrossRef] [PubMed] [Google Scholar]
- Mattar P, Ericson J, Blackshaw S, Cayouette M. A conserved regulatory logic controls temporal identity in mouse neural progenitors. Neuron 2015 ; 85 : 497–504. [CrossRef] [PubMed] [Google Scholar]
- Javed A, Mattar P, Lu S, et al. Pou2f1 and Pou2f2 cooperate to control the timing of cone photoreceptor production in the developing mouse retina. Development 2020; 147. [Google Scholar]
- Konstantinides N, Rossi AM, Desplan C. Common temporal identity factors regulate neuronal diversity in fly ventral nerve cord and mouse retina. Neuron 2015 ; 85 : 447–449. [CrossRef] [PubMed] [Google Scholar]
- Sagner A, Zhang I, Watson T, et al. A shared transcriptional code orchestrates temporal patterning of the central nervous system. PLoS Biol 2021; 19. [Google Scholar]
- Isshiki T, Pearson B, Holbrook S, Doe CQ. Drosophila neuroblasts sequentially express transcription factors which specify the temporal identity of their neuronal progeny. Cell 2001 ; 106 : 511–521. [CrossRef] [PubMed] [Google Scholar]
- Filippopoulou K, Konstantinides N. Evolution of patterning. FEBS J 2023. doi : 10.1111/febs.16995. [PubMed] [Google Scholar]
- Li X, Erclik T, Bertet C, et al. Temporal patterning of Drosophila medulla neuroblasts controls neural fates. Nature 2013 ; 498 : 456–462. [CrossRef] [PubMed] [Google Scholar]
- Konstantinides N, Holguera I, Rossi AM, et al. A complete temporal transcription factor series in the fly visual system. Nature 2022; 604 : 316–22. [CrossRef] [PubMed] [Google Scholar]
- Nériec N, Desplan C, Neriec N, Desplan C. From the Eye to the Brain : Development of the Drosophila Visual System. Curr Top Dev Biol 2016 ; 116 : 247–271. [CrossRef] [PubMed] [Google Scholar]
- Simon F, Konstantinides N. Single-cell transcriptomics in the Drosophila visual system : Advances and perspectives on cell identity regulation, connectivity, and neuronal diversity evolution. Dev Biol 2021; 479 : 107–22. [CrossRef] [PubMed] [Google Scholar]
- Zhu H, Zhao SD, Ray A, et al. A comprehensive temporal patterning gene network in Drosophila medulla neuroblasts revealed by single-cell RNA sequencing. Nat Commun 2022; 13. [PubMed] [Google Scholar]
- Sagner A, Zhang I, Watson T, et al. A shared transcriptional code orchestrates temporal patterning of the central nervous system. PLoS Biol 2021; 19. [Google Scholar]
- Hasegawa E, Kitada Y, Kaido M, et al. Concentric zones, cell migration and neuronal circuits in the Drosophila visual center. Development 2011 ; 138 : 983–993. [CrossRef] [PubMed] [Google Scholar]
- Özel MN, Simon F, Jafari S, et al. Neuronal diversity and convergence in a visual system developmental atlas. Nature 2021; 589 : 88–95. [CrossRef] [PubMed] [Google Scholar]
- Hobert O.. Regulatory logic of neuronal diversity : terminal selector genes and selector motifs. Proc Natl Acad Sci USA 2008 ; 105(20): 067–071. [Google Scholar]
- Hobert O.. Regulation of terminal differentiation programs in the nervous system. Annu Rev Cell Dev Biol 2011 ; 27 : 681–696. [CrossRef] [PubMed] [Google Scholar]
- Özel MN, Gibbs CS, Holguera I, et al. Coordinated control of neuronal differentiation and wiring by sustained transcription factors. Science 2022; 378. [Google Scholar]
- Chen YC, Konstantinides N. Integration of Spatial and Temporal Patterning in the Invertebrate and Vertebrate Nervous System. Front Neurosci 2022; 16. [Google Scholar]
- Konstantinides N, Degabriel S, Desplan C. Neuro-evo-devo in the single cell sequencing era. Curr Opin Syst Biol 2018 ; 11 : 32–40. [CrossRef] [PubMed] [Google Scholar]
- Perry M, Konstantinides N, Pinto-Teixeira F, Desplan C. Generation and Evolution of Neural Cell Types and Circuits : Insights from the Drosophila Visual System. Annu Rev Genet 2017 ; 51 : 501–527. [CrossRef] [PubMed] [Google Scholar]
- Kurmangaliyev YZ, Yoo J, Valdes-Aleman J, et al. Transcriptional Programs of Circuit Assembly in the Drosophila Visual System. Neuron 2020; 108 : 1045–57. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Encodage spatial et temporel dans le système visuel en développement de la drosophile. A. Encodage spatial dans la moelle épinière des vertébrés (à gauche). Chaque domaine spatial (DS) est défini par l’expression de gènes distincts le long de l’axe dorso-ventral, grâce à des gradients de concentration opposés de Shh et de Wnt/BMP. Chez la drosophile (à droite), la compartimentation en 8 domaines spatiaux du neuroépithélium de l’OPC ajoute un niveau de complexité et de complétude à la différenciation neuronale. B.Encodage temporel dans les neuroblastes de l’OPC chez la drosophile. À chaque fenêtre d’expression temporelle, le neuroblaste se divise asymétriquement pour se renouveler et donner naissance à une cellule mère ganglionnaire (CMG). Chaque fenêtre d’expression temporelle conduit à la génération d’un type de cellule neuronale spécifique. C. Destin binaire Notch-dépendant des neurones. Les CMG se divisent de manière asymétrique pour générer deux neurones, un NotchOFF et un NotchON. Les cellules barrées indiquent la mort cellulaire. FT : facteur de transcription. |
| Dans le texte | |
 |
Figure 2. Modèle schématique de la neurogenèse dans la médulla. Les cellules du neuroépithélium (NE, carrés gris) se convertissent progressivement en neuroblastes (NB, grands cercles). Ces derniers se divisent de manière asymétrique pour générer des cellules mères ganglionnaires (CMG), qui se divisent en neurones. Au fur et à mesure que les NB de la médulla vieillissent, ils expriment séquentiellement une série de facteurs de transcription conduisant à la genèse d’une colonne de neurones d’identités différentes (petits cercles). Cette différenciation se produit médialement vers le côté latéral du lobe optique. Les neurones NotchON sont représentés par des cercles pleins, tandis que les neurones NotchOFF sont représentés par des cercles vides. Les flèches en pointillés représentent le sens de la vague neuronale. hth : homothorax ; ey : eyeless ; slp : sloppy-paired ; D : Dichaete ; tll : tailless. |
| Dans le texte | |
 |
Figure 3. Interactions génétiques et série temporelle finale lors du développement du lobe optique de la larve de drosophile. A. Interactions génétiques entre les FTt précédemment identifiés et les nouveaux FTt identifiés dans les neuroblastes de la médulla. B. Schéma représentant les séries temporelles mises à jour dans les neuroblastes du centre externe de prolifération (OPC), les cellules mères ganglionnaires (CMG) et le destin Notch des neurones, ainsi que le nom des neurones générés par chaque fenêtre temporelle (identité du neurone). hth : homothorax ; opa : odd paired ; erm : earmuff ; ey : eyeless ; hbn : homeobrain ; scro : scarecrow ; B-H1 : BarH1 ; slp : sloppy-paired ; D : Dichaete ; tll : tailless. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




