| Issue |
Med Sci (Paris)
Volume 40, Number 2, Février 2024
|
|
|---|---|---|
| Page(s) | 127 - 129 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2023204 | |
| Published online | 27 February 2024 | |
La ribonucléase PA-X du virus de la grippe A différencie les ARN viraux et cellulaires grâce à sa préférence de clivage
The influenza A virus ribonuclease PA-X can differentiate between cellular and viral RNAs through its cut site preference
1
Université de Strasbourg, Architecture et réactivité de l’ARN, Institut de biologie moléculaire et cellulaire du CNRS, Strasbourg, France
2
Institute for molecular virology and department of medical microbiology and immunology, Université de Wisconsin-Madison, Madison, États-Unis
* l.gaucherand@ibmc-cnrs.unistra.fr
** marta.gaglia@wisc.edu
Les cellules humaines possèdent différents moyens de combattre les infections virales. La première étape commune de défense antivirale est la détection de la présence du virus dans les cellules en tant que corps étranger, ou « non-soi ». Le concept de reconnaissance du non-soi occupe une place centrale en immunologie. Par exemple, certaines protéines peuvent détecter la présence d’ARN modifié ou d’ADN dans le cytoplasme, et ainsi activer la réponse immunitaire. À l’opposé, on peut se demander si les virus sont eux-aussi capables de distinguer le soi du non-soi, et de cibler uniquement des macromolécules cellulaires lors de l’infection. Les résultats de nos travaux de recherche sur le virus de la grippe A semblent confirmer cette hypothèse. Nous avons en effet montré que ce virus a la capacité de différencier l’ARN viral de l’ARN cellulaire grâce à sa protéine PA-X [1].
Le virus de la grippe A est un virus pathogène pour l’homme, mais aussi pour les mammifères et les oiseaux. Le nombre de décès provoqués par une infection grippale est estimé entre 290 000 et 645 000 par an dans le monde [2]. Bien que plusieurs vaccins existent, leur efficacité varie en fonction des années et de l’évolution du virus saisonnier. De plus, la réponse immunitaire à ces vaccins ne permet pas une protection de longue durée. Quelques médicaments antiviraux existent également, mais certaines souches résistantes à ces molécules ont déjà été identifiées. Au risque saisonnier d’infection par le virus de la grippe s’ajoute le risque constant de pandémie. Il y a donc un réel intérêt à développer de nouveaux outils pour la prévention et le traitement de cette infection. Le développement de solutions innovantes passe par l’étude du virus lui-même.
Le virus de la grippe A est un virus à ARN simple brin de polarité négative, composé de huit segments d’ARN. Pour se répliquer, le virus doit détourner la machinerie cellulaire afin de synthétiser les ARN viraux et fabriquer les protéines virales. Ce détournement passe par le blocage de l’expression des gènes cellulaires par le virus, un phénomène nommé « host shutoff », qui permet de rediriger les ressources de la cellule vers la fabrication de nouvelles particules virales, et empêche la cellule de se défendre contre l’infection. Pour le virus de la grippe A, le host shutoff est principalement réalisé grâce à la protéine virale PA-X. Cette endoribonucléase dégrade les ARN cellulaires, ce qui supprime leur activité ou empêche leur traduction en protéine, libérant ainsi les ribosomes pour la traduction des ARN viraux [3]. Puisque PA-X ne dégrade pas l’ARN viral [4], il importe de comprendre comment elle distingue les ARN cellulaires des ARN viraux. Nous avions précédemment montré que PA-X cible préférentiellement les ARN épissés aux dépens des ARN qui ne contiennent pas d’introns [5]. Comme la majorité des segments d’ARN du virus de la grippe A ne sont pas épissés, ce mécanisme pourrait expliquer en partie comment les ARN viraux échappent à l’action de PA-X. Cependant, le virus de la grippe possède deux ARN épissés. Il existe donc un autre mécanisme permettant de faire la distinction entre les ARN cellulaires et les ARN viraux, mais lequel ?
Pour tenter de répondre à cette question, nous avons développé une méthode d’identification des ARN préférentiellement coupés par PA-X (Figure 1A). Nous avons adapté la technique de 5’RACE-PCR (rapid amplification of cDNA ends by polymerase chain reaction) au séquençage nucléotidique à haut débit afin de marquer, puis séquencer tous les ARN coupés après une infection par le virus de la grippe A de génotype « sauvage », ou par un virus mutant incapable de produire PA-X. Nous avons ensuite optimisé le programme PyDegradome [6] pour identifier les fragments d’ARN plus nombreux en présence de PA-X. Cette méthode a permis d’identifier les principaux sites de clivage de l’ARN cellulaire par PA-X, et plusieurs de ces sites ont ensuite été validés par la technique 5’RACE-PCR classique. De plus, en introduisant la séquence nucléotidique des sites validés dans un ARN rapporteur, nous avons constaté l’apparition d’un nouveau clivage de l’ARN par PA-X à cet emplacement, ce qui indique que cette séquence est suffisante pour provoquer le clivage (Figure 1B). Nous avons également confirmé que la même séquence est aussi clivée par les ribonucléases PA-X de plusieurs souches différentes du virus de la grippe A représentatives de virus actuellement en circulation, ce qui atteste de son importance du point de vue de l’évolution. Nous avons donc analysé la séquence nucléotidique des principaux sites de clivage identifiés, et découvert que plus de la moitié contiennent le motif GCUG au site du clivage. En introduisant des mutations dans l’ARN rapporteur, nous avons montré que ce motif est nécessaire, mais pas suffisant pour un clivage efficace de l’ARN par PA-X (Figure 1B). Les prédictions de structure secondaire de l’ARN autour de chaque site de clivage avec le programme LinearFold [7] ont montré que les séquences préférentiellement coupées par PA-X forment le plus souvent une structure d’ARN en épingle à cheveux, le site de clivage GCUG étant situé dans la boucle simple brin de l’épingle. En introduisant de nouvelles mutations dans l’ARN rapporteur pour déstabiliser cette structure, nous avons confirmé son importance pour un clivage efficace de l’ARN par PA-X (Figure 1B).
 |
Figure 1. A. Stratégie expérimentale pour identifier, dans le transcriptome cellulaire, les sites de clivage de l’ARN par l’endoribonucléase virale PA-X. Nous avons infecté une lignée cellulaire de cellules épithéliales de poumon humain avec le virus de la grippe A de génotype « sauvage » (wild-type, WT) ou avec un virus muté pour ne plus exprimer la protéine PA-X (∆X), puis purifié l’ARN de chaque échantillon. Suivant le principe de la technique de 5’RACE-PCR, nous avons marqué le site de clivage en liant un adaptateur à l’extrémité 5’ de chaque fragment d’ARN clivé, transformé ces fragments d’ARN en ADNc par transcription inverse (reverse transcription, RT), puis ajouté les adaptateurs de séquençage aux deux extrémités de ces ADNc par PCR. Après l’étape de séquençage nucléotidique à haut débit, nous avons identifié les sites clivés enrichis en présence de PA-X (virus WT versus virus ∆X) grâce au programme PyDegradome. Pour valider les sites clivés identifiés, nous avons suivi le même protocole expérimental, mais en effectuant cette fois deux PCR emboîtées avec des amorces spécifiques du gène étudié. Nous avons ensuite visualisé les fragments amplifiés sur un gel d’agarose, et confirmé leur identité par purification sur gel et séquençage nucléotidique par la méthode de Sanger. B. Différentes versions de l’ARN rapporteur utilisées pour les expériences de clivage de l’ARN par PA-X. L’ajout de la séquence de sites de clivages identifiés fait apparaître un nouveau site de clivage du rapporteur à cet endroit. Ce site n’est plus clivé si le motif GCUG de sa séquence nucléotidique est remplacé par UAGC. De même, il n’est pas clivé si seul le motif GCUG est ajouté et pas le reste de la séquence du site de clivage, ou si cette séquence est mutée pour ne plus former une structure en épingle à cheveux. C. Différentes modifications expérimentales apportées au virus de la grippe A : ajout de la séquence d’un site de clivage validé en présence ou en absence de PA-X, et ajout de cette séquence en remplaçant le motif GCUG central en UAGC en présence ou en absence de PA-X. L’ajout de ces séquences dans le génome du virus n’est toléré qu’en l’absence de PA-X, ou si le motif GCUG central est remplacé par UAGC. Sinon, elles sont rapidement éliminées par mutation. |
PA-X clive donc principalement les ARN dont la séquence nucléotidique possède le motif GCUG lorsque celui-ci est présent dans la boucle d’une structure ARN en épingle à cheveux. Comment ce clivage préférentiel aide-t-il à différencier les ARN cellulaires des ARN viraux ? Il se trouve que le motif GCUG est un des tétramères les plus fréquents dans les séquences nucléotidiques des ARN cellulaires humains, alors qu’il est bien moins fréquent dans les ARN du virus de la grippe A. Ce motif est aussi moins fréquent dans les séquences des ARN viraux de plusieurs souches de virus de grippes aviaires et porcines que dans celles des ARN de leurs hôtes respectifs. De plus, les quelques motifs GCUG présents dans les séquences des ARN viraux sont plus souvent situés dans des structures double brin plutôt qu’en épingle à cheveux. La préférence de clivage de l’ARN par PA-X lui permettrait donc de dégrader prioritairement les ARN cellulaires plutôt que les ARN viraux. Afin de tester cette hypothèse, nous avons introduit une séquence préférentiellement clivée par PA-X dans l’un des segments épissés de l’ARN du virus de la grippe. Après quelques cycles d’infection seulement, le virus mute naturellement pour supprimer la séquence introduite artificiellement. En revanche, cette séquence n’est pas supprimée lorsque le virus utilisé est un mutant incapable d’exprimer PA-X, ou si la séquence introduite possède une mutation du motif GCUG qui empêche son clivage par PA-X (Figure 1C). Ce résultat suggère que posséder une séquence préférentiellement clivée par PA-X est désavantageux pour le virus, et explique que la présence de telles séquences dans les ARN cellulaires permette à PA-X de cibler davantage ces ARN (Figure 2).
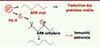 |
Figure 2. Mécanisme du ciblage des ARN cellulaires par PA-X. Les résultats expérimentaux indiquent que PA-X coupe préférentiellement l’ARN à la séquence nucléotidique GCUG lorsque celle-ci est présente dans la boucle simple brin d’une structure en épingle à cheveux. Ce motif étant plus abondant dans les ARN cellulaires que dans les ARN viraux, ce mode de dégradation des ARN par PA-X permet au virus de cibler préférentiellement les ARN cellulaires. Ce mécanisme limite donc la réponse antivirale, tout en « libérant » les ribosomes de la cellule hôte pour la synthèse des protéines virales. |
Ces résultats viennent étayer le concept de distinction entre le soi et le non-soi par les virus. D’autres virus ont d’ailleurs développé des stratégies différentes pour éviter que leurs propres ARN soient clivés par leurs ribonucléases. Par exemple, la protéine nsp1 des coronavirus MERS (middle east respiratory syndrome), SARS (severe acute respiratory syndrome) et SARS-CoV-2 reconnaît un motif situé au début de la séquence de l’ARN viral, qui empêche son clivage [8]. Au contraire, le virus de l’herpès associé au sarcome de Kaposi utilise sa ribonucléase SOX pour dégrader l’ARN viral des transcrits précoces, en plus des ARN cellulaires, et faciliter ainsi l’expression des gènes viraux tardifs [9]. En utilisant une technique similaire, nous avions précédemment découvert que la ribonucléase SOX reconnaît également un motif spécifique dans la séquence nucléotidique des ARN qu’elle clive [6] : de même que pour PA-X, le motif reconnu par SOX est assez simple, et présent dans de nombreux ARN. Il est vraisemblable que d’autres virus parviennent aussi à différencier le soi du non-soi en utilisant d’autres stratégies, qu’il reste à découvrir.
Liens d’intérêt
Les auteures déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Gaucherand L, Iyer A, Gilabert I, et al. Cut site preference allows influenza A virus PA-X to discriminate between host and viral mRNAs. Nat Microbiol 2023; 8 : 1 304–17. [Google Scholar]
- Iuliano AD, Roguski KM, Chang HH, et al. Estimates of global seasonal influenza-associated respiratory mortality : a modelling study. Lancet 2018 ; 391 : 1 285–300. [Google Scholar]
- Jagger BW, Wise HM, Kash JC, et al. An overlapping protein-coding region in influenza A virus segment 3 modulates the host response. Science 2012 ; 337 : 199–204. [CrossRef] [PubMed] [Google Scholar]
- Khapperskyy DA, Schmaling S, Larkins-Ford J, et al. Selective degradation of host RNA polymerase II transcripts by influenza A virus PA-X host shutoff protein. PLoS Pathog 2016 ; 12 : e1005427. [CrossRef] [PubMed] [Google Scholar]
- Gaucherand L, Porter BK, Levene RE, et al. The influenza A virus endoribonuclease PA-X usurps host mRNA processing machinery to limit host gene expression. Cell Rep 2019 ; 27 : 776–92 e7. [CrossRef] [PubMed] [Google Scholar]
- Gaglia MM, Rycroft CH, Glaunsinger BA. Transcriptome-wide cleavage site mapping on cellular mRNAs reveals features underlying sequence-specific cleavage by the viral ribonuclease SOX. PLoS Pathog 2015 ; 11 : e1005305. [CrossRef] [PubMed] [Google Scholar]
- Huang L, Zhang H, Denget D, et al. LinearFold : linear-time approximate RNA folding by 5’-to-3’dynamic programming and beam search. Bioinformatics 2019 ; 35 : i295–i304. [CrossRef] [PubMed] [Google Scholar]
- Yuan S, Balaji S, Lomakin IB, et al. Coronavirus Nsp1 : Immune response suppression and protein expression inhibition. Front Microbiol 2021; 12 : 752 214. [Google Scholar]
- Abernathy E, Clyde K, Yeasmin R, et al. Gammaherpesviral gene expression and virion composition are broadly controlled by accelerated mRNA degradation. PLoS Pathog 2014 ; 10 : e1003882. [CrossRef] [PubMed] [Google Scholar]
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. A. Stratégie expérimentale pour identifier, dans le transcriptome cellulaire, les sites de clivage de l’ARN par l’endoribonucléase virale PA-X. Nous avons infecté une lignée cellulaire de cellules épithéliales de poumon humain avec le virus de la grippe A de génotype « sauvage » (wild-type, WT) ou avec un virus muté pour ne plus exprimer la protéine PA-X (∆X), puis purifié l’ARN de chaque échantillon. Suivant le principe de la technique de 5’RACE-PCR, nous avons marqué le site de clivage en liant un adaptateur à l’extrémité 5’ de chaque fragment d’ARN clivé, transformé ces fragments d’ARN en ADNc par transcription inverse (reverse transcription, RT), puis ajouté les adaptateurs de séquençage aux deux extrémités de ces ADNc par PCR. Après l’étape de séquençage nucléotidique à haut débit, nous avons identifié les sites clivés enrichis en présence de PA-X (virus WT versus virus ∆X) grâce au programme PyDegradome. Pour valider les sites clivés identifiés, nous avons suivi le même protocole expérimental, mais en effectuant cette fois deux PCR emboîtées avec des amorces spécifiques du gène étudié. Nous avons ensuite visualisé les fragments amplifiés sur un gel d’agarose, et confirmé leur identité par purification sur gel et séquençage nucléotidique par la méthode de Sanger. B. Différentes versions de l’ARN rapporteur utilisées pour les expériences de clivage de l’ARN par PA-X. L’ajout de la séquence de sites de clivages identifiés fait apparaître un nouveau site de clivage du rapporteur à cet endroit. Ce site n’est plus clivé si le motif GCUG de sa séquence nucléotidique est remplacé par UAGC. De même, il n’est pas clivé si seul le motif GCUG est ajouté et pas le reste de la séquence du site de clivage, ou si cette séquence est mutée pour ne plus former une structure en épingle à cheveux. C. Différentes modifications expérimentales apportées au virus de la grippe A : ajout de la séquence d’un site de clivage validé en présence ou en absence de PA-X, et ajout de cette séquence en remplaçant le motif GCUG central en UAGC en présence ou en absence de PA-X. L’ajout de ces séquences dans le génome du virus n’est toléré qu’en l’absence de PA-X, ou si le motif GCUG central est remplacé par UAGC. Sinon, elles sont rapidement éliminées par mutation. |
| Dans le texte | |
 |
Figure 2. Mécanisme du ciblage des ARN cellulaires par PA-X. Les résultats expérimentaux indiquent que PA-X coupe préférentiellement l’ARN à la séquence nucléotidique GCUG lorsque celle-ci est présente dans la boucle simple brin d’une structure en épingle à cheveux. Ce motif étant plus abondant dans les ARN cellulaires que dans les ARN viraux, ce mode de dégradation des ARN par PA-X permet au virus de cibler préférentiellement les ARN cellulaires. Ce mécanisme limite donc la réponse antivirale, tout en « libérant » les ribosomes de la cellule hôte pour la synthèse des protéines virales. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




