| Issue |
Med Sci (Paris)
Volume 32, Number 2, Février 2016
|
|
|---|---|---|
| Page(s) | 192 - 197 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20163202013 | |
| Published online | 02 March 2016 | |
Spécification de l’épiblaste et de l’endoderme primitif lors du développement embryonnaire préimplantatoire chez la souris
Une combinaison entre biologie et modélisation mathématique
Epiblast and primitive endoderm cell specification during mouse preimplantation development: a combination between biology and mathematical modeling
1
GReD, Inserm U1103, CNRS UMR6293, Clermont Université, 28, place Henri Dunant, BP38, 63001 Clermont-Ferrand Cedex, France
2
Unité de chronobiologie théorique, faculté des Sciences, université libre de Bruxelles (ULB), campus Plaine, CP 231, B-1050 Bruxelles, Belgique
*
sylvain.bessonnard@pasteur.fr
Lors de son implantation dans l’utérus de la mère chez les mammifères, l’embryon est composé de trois tissus morphologiquement distincts : l’épiblaste (Epi), le trophectoderme (TE) et l’endoderme primitif (EPr). L’Epi et l’EPr se forment à partir de la même population cellulaire homogène appelée la masse cellulaire interne (MCI). Le but de nos études, décrites dans cette revue, est de comprendre les interactions moléculaires nécessaires à la mise en place de ces deux lignages. Pour cela, nous avons combiné une approche biologique et une modélisation mathématique. Nous avons ainsi montré le rôle central du groupe de régulation génique NANOG, FGF4, GATA6, FGFR2 pour la spécification de l’Epi et de l’EPr. Il s’agit d’un projet de recherche fondamental, nécessaire pour la compréhension des mécanismes de spécification au sein de l’embryon précoce. Ces recherches peuvent avoir des retombées importantes dans de nombreux autres sujets de recherche, tels que l’amélioration des protocoles de fécondation in vitro et d’assistance médicale à la procréation, mais aussi pour des applications liées à l’utilisation de cellules souches pour la médecine régénératrice.
Abstract
Upon its implantation in the uterus of the mother in mammals, the embryo is composed by three morphologically distinct tissues: the Epiblast (Epi), the Trophectoderm (TE) and the Primitive Endoderm (PrE). Both Epi and PrE are formed from the same cell homogeneous population called the Inner Cell Mass (ICM). Based on our studies, we discuss in this review what molecular interactions are necessary for the specification of these two lineages. For this, we have combined a biological approach with mathematical modeling. We have shown the central role of the gene regulation group composed by NANOG, FGF4, GATA6 and FGFR2 for Epi/PrE cell specification.
© 2016 médecine/sciences – Inserm
Le développement embryonnaire préimplantatoire comprend toutes les étapes développementales depuis la fécondation jusqu’à l’implantation dans l’utérus de la mère, qui a lieu 4,5 jours après la fécondation (E4,5) chez la souris (Figure 1). À ce stade, l’embryon est composé de trois lignages cellulaires morphologiquement distincts : l’épiblaste (Epi), le trophectoderme (TE) et l’endoderme primitif (EPr). L’Epi est le tissu embryonnaire à partir duquel seront produits tous les tissus et organes présents chez le nouveau-né. Le TE et l’EPr sont deux tissus extra-embryonnaires indispensables pour le développement. Le TE est nécessaire pour l’implantation de l’embryon au sein de l’utérus et pour la mise en place de la partie embryonnaire du placenta. L’EPr est requis pour la formation du sac vitellin. Les deux tissus jouent aussi un rôle majeur dans l’établissement de l’axe antéropostérieur au sein de l’embryon [1] (→).
(→) Voir la Synthèse de C. Chazaud, m/s n° 12, décembre 2008, page 1043
 |
Figure 1. Représentation schématique du développement préimplantatoire chez la souris. MCI : masse cellulaire interne. |
À E2,5 (2,5 jours après la fécondation), l’embryon est composé de 8 cellules totipotentes qui, lors de la compaction, amplifient leurs contacts intercellulaires, assemblent des jonctions d’adhérence et se polarisent, formant le premier pseudo-épithélium dans l’embryon. Les cellules vont alors effectuer successivement deux vagues de division, faisant passer leur nombre de 8 à 32 cellules. On distingue deux types de division : elles sont dites asymétriques quand elles donnent naissance à une cellule polarisée externe et une cellule non polarisée interne, et elles sont dites symétriques quand elles induisent la formation de deux cellules polarisées externes identiques (Figure 1). Les cellules externes vont s’engager vers le devenir TE tandis que les cellules internes restent pluripotentes et forment la masse cellulaire interne (MCI) [1, 2]. Une cavité se forme et repousse les cellules de la MCI à un pôle de l’embryon, qui prend le nom de blastocyste à partir de E3,25. Au même moment, les cellules de la MCI débutent leur spécification en Epi et en EPr [1] (Figure 1).
Au sein de la MCI, les facteurs de transcription NANOG et GATA6 (GATA binding protein 6), le ligand FGF4 (fibroblast growth factor 4) ainsi que son récepteur FGFR2 (fibroblast growth factor receptor 2) jouent un rôle central dans cette spécification. Ils se retrouvent co-exprimés dans toutes les cellules à E2,5 puis se restreignent aux cellules de la MCI. À partir de E3,25, une expression exclusive et complémentaire se met en place dans quelques cellules, puis se généralise à la majorité de la MCI à E3,75 selon une organisation dite en « poivre et sel », qualifiant une distribution aléatoire des cellules de chaque type [3, 4] (Figure 1). NANOG et FGF4 sont alors co-exprimés dans les cellules Epi, tandis que GATA6 et FGFR2 le sont dans les cellules EPr. Ce mécanisme se fait de manière asynchrone, à savoir que ce phénomène ne se déclenche pas dans toutes les cellules de la MCI en même temps. Cette expression exclusive des marqueurs selon qu’une cellule est de type Epi ou EPr définit la spécification cellulaire, c’est-à-dire l’engagement vers un destin cellulaire donné. En revanche, leur spécification n’implique pas de facto leur détermination, c'est-à-dire leur incapacité à changer d’identité. En effet, une plasticité dans leur devenir cellulaire est possible jusqu’à un stade développemental avancé (E4,0-E4,25). Elle peut être contrôlée par l’activation ou l’inhibition artificielles de la signalisation dépendant des récepteurs à activité tyrosine kinase (RTK), modulée dans l’embryon par la voie de signalisation FGF4-FGFR2-MEK (MAPK/ERK kinase)/ERK (extracellular signal-regulated kinases) (par la suite, cette voie de signalisation sera définie par le terme « signalisation FGF/RTK »). Pour cela, les embryons sont cultivés en présence soit de FGF4 exogène pour activer la signalisation, soit d’inhibiteurs spécifiques des récepteurs FGFR et de la voie MEK-ERK pour l’inhiber [5, 6].
Des études se sont intéressées aux rôles de NANOG et de GATA6 dans la différenciation de la MCI. Tout d’abord, l’invalidation génique de Nanog dans les cellules souches embryonnaires (ES) a montré que ce facteur de transcription est nécessaire pour la dérivation des lignées1, mais ne l’est pas pour leur maintien en culture, une fois les cellules produites, suggérant que Nanog n’est requis que pour l’initiation de l’état Epi [7]. L’analyse d’embryons mutants Nanog-/- à E4,5 montre, en outre, que les cellules de la MCI expriment le marqueur de pluripotence OCT4 (octamer-binding protein 4), mais n’expriment pas les marqueurs EPr GATA4 et 6 [8]. L’invalidation génique de Gata6 entraîne, quant à elle, une absence d’épithélium d’EPr à E4,5 indiquant que Gata6 est requis pour sa mise en place [9]. Néanmoins, ces analyses, pour être plus précises, nécessitaient une étude des phénotypes aux stades plus précoces, avant 4,5 jours après la fécondation. Elles ont été réalisées récemment, notamment par notre équipe. Elles nous ont permis de découvrir des rôles nouveaux pour NANOG, GATA6 et pour la signalisation FGF/RTK. Une approche mathématique nous a également apporté des éléments de réponse à deux questions importantes : les interactions entre NANOG, FGF4, GATA6 et FGFR2 sont-elles suffisantes pour expliquer la mise en place du patron en « poivre et sel » au sein de la MCI ? Et, quelle est la dynamique temporelle de ces marqueurs au sein de chaque cellule de la MCI ?
Des mutants pour comprendre la spécification de l’Epi et de l’EPr
L’analyse d’embryons de souris Nanog-/- montre que, au stade E3,75, toutes les cellules de la masse cellulaire interne (MCI) expriment le marqueur endodermique (EPr) GATA6. Nanog est donc requis pour la spécification des cellules en Epi ; en son absence, toutes les cellules s’engagent vers le devenir EPr (Figure 2). Cependant, bien que toutes les cellules soient engagées, elles n’expriment pas les marqueurs plus tardifs de l’EPr, tels que SOX17 (SRY [sex determining region Y]-box 17), GATA4 ou PDFGRα (platelet-derived growth factor receptor, alpha). Nanog est donc nécessaire pour la différenciation de l’EPr [8, 10]. Cela s’explique par le fait qu’en absence de Nanog, les cellules de la MCI n’expriment pas FGF4 [10, 11]. De fait, la culture des embryons mutants en présence de FGF4 exogène permet d’induire l’expression des marqueurs tardifs de l’EPr, ce qui montre que FGF4 est suffisante pour induire la différenciation des cellules en EPr. Les mutants Nanog-/- ont été cultivés en présence d’inhibiteurs spécifiques de la signalisation FGF/RTK afin de comprendre la façon dont l’expression de GATA6 est contrôlée. Alors que la culture des embryons sauvages en présence des inhibiteurs de signalisation permet d’engager toutes les cellules de la MCI vers le devenir Epi, en activant NANOG et en inhibant GATA6 jusqu’au stade E4,0-E4,25 dans les embryons mutants, l’expression de GATA6 est insensible aux inhibiteurs dès le stade E3,25 [10].
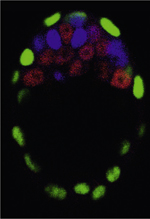
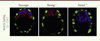 |
Figure 2. Détection par immunofluorescence de NANOG, GATA6 et CDX2 (caudal type homeobox 2) au stade E3,75 dans les embryons sauvages, Nanog-/- et Gata6-/-. Dans les embryons Nanog-/- ou GATA6-/- (GATA binding protein 6), l’expression en « poivre et sel » disparaît, faisant place à des MCI composées entièrement d’EPr (exprimant GATA6) ou d’Epi (exprimant NANOG). CDX2 est utilisé pour repérer le tropherdoderme. Epi : épiblaste ; EPr : endoderme primitif ; MCI : masse cellulaire interne. |
L’analyse des embryons de souris Gata6-/- à E3,75 montre que toutes les cellules de la MCI expriment NANOG (ce qui indique qu’elles adoptent le devenir Epi) démontrant que Gata6 est requis pour que les cellules de la MCI adoptent un destin EPr (Figure 2) [12]. Pour déterminer si, comme dans les cas des embryons Nanog-/-, FGF4 est suffisante pour l’expression des marqueurs tardifs de l’EPr (SOX17 et GATA4), les mutants Gata6-/- ont été cultivés en présence de FGF4 ectopique. Dans le cas de ces embryons mutants, aucune cellule de la MCI n’exprime SOX17 ou GATA4. Gata6 est donc nécessaire à la maturation des cellules d’EPr et, en son absence, l’action de FGF4 n’est pas suffisante pour induire l’expression des marqueurs tardifs de ces cellules. Ces expériences ont également mis en exergue l’action de la signalisation FGF/RTK sur l’inhibition de NANOG. Pour rappel, la culture d’embryons sauvages en présence de FGF4 entraîne l’expression des marqueurs EPr et l’inhibition de NANOG dans toutes les cellules de la MCI jusqu’à des stades avancés du développement, autour de E4,0-E4,25 [6]. En revanche, la même expérience sur les embryons Gata6-/- montre que l’expression de NANOG devient insensible à la signalisation RTK dès le stade E3,25 [12, 13]. Cette insensibilité précoce, observée lors de l’analyse des deux mutants, suggère que les expressions de NANOG et GATA6 dépendent de la signalisation FGF/RTK jusqu’au stade E3,25 ; elles deviennent indépendantes par la suite.
Un modèle mathématique pour élucider le mécanisme de spécification
Nous avons compilé les résultats que nous avons obtenus avec ceux de la littérature afin de développer une approche de modélisation mathématique de la spécification. Nous avons obtenu une représentation relativement simple des différentes interactions entre les membres du réseau de régulation génique NANOG/FGF4/GATA6/FGFR2 (Figure 3). Ainsi, pour chaque cellule, quatre variables caractérisent le niveau protéique intracellulaire de NANOG, FGFR2, et GATA6 ainsi que l’activité de la signalisation FGF/RTK, qui dépend directement de la concentration extracellulaire de FGF4. Dans cette première approche, la concentration extracellulaire de FGF4 a été fixée par un paramètre, défini au début de la simulation.
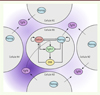 |
Figure 3. Représentation du réseau de régulation génique de la spécification. Les principaux acteurs sont les facteurs de transcription NANOG et GATA6 (GATA binding protein 6), le récepteur FGFR2 (fibroblast growth factor receptor 2), son ligand FGF4 (fibroblast growth factor 4) et la voie de signalisation MEK (MAPK/ERK kinase)/ERK (extracellular signal-regulated kinases). La sécrétion de FGF4 dans le milieu extracellulaire permet la communication intercellulaire. La concentration de FGF4 dépend de sa production par la cellule (N5) et de ses quatre cellules voisines (N1 à N4). L’intensité de la couleur violette reflète la concentration du FGF4 (adapté de Bessonnard et al. [12]). |
Des analyses ont été réalisées tout d’abord sur une cellule isolée afin de déterminer le nombre et la nature des états stables du système. Un état stationnaire stable est un état, indépendant de l’état initial, que les cellules tendent à atteindre au cours du temps. Dans les conditions que nous avons utilisées (Figure 4), les résultats suggèrent que le système présente une tri-stabilité. En effet, pour une certaine gamme de concentrations en FGF4, représentée par la zone jaune de la Figure 4A , la cellule peut atteindre trois états stables distincts qui diffèrent par leurs concentrations de NANOG et de GATA6 et le niveau d’activité FGF/RTK. Il s’agit d’un état dit « MCI » (en gris dans la Figure 4A ), dans lequel GATA6 et NANOG sont co-exprimés à des niveaux faibles ; un état « Epi » (en bleu dans la Figure 4A ) dans lequel seul NANOG est exprimé ; et un stade « EPr » (en rouge dans la Figure 4A ) dans lequel seul GATA6 est exprimé. Dans nos conditions, la seule modification de la concentration extracellulaire de FGF4 est suffisante pour contrôler l’état stable dans lequel la cellule va s’engager : une faible concentration en FGF4 oriente les cellules vers l’état « Epi » alors qu’une forte concentration les engage vers le stade « EPr ».
 |
Figure 4. Résultats de la modélisation. Le panneau A décrit le comportement du réseau de régulation génétique associé à une cellule isolée, tandis que les panneaux B et C illustrent le comportement d’une population de 25 cellules. A. Les courbes de GATA6 en fonction de la concentration en FGF4 montrent les états stables possibles de la cellule. Selon les concentrations faibles ou élevées de FGF4, seul l’état Epi (concentration faible) ou l’état EPr (concentration élevée) est possible, comme illustré sur les panneaux a et c. Pour des concentrations intermédiaires, la cellule peut évoluer vers l’un des trois états stables en fonction des conditions initiales (panneau b). B. Les trajectoires de 25 cellules dont l’évolution est régie par le même réseau de régulation génétique, mais où la sécrétion de FGF4 par les cellules exprimant NANOG est prise en compte, sont montrées. Une sous-population de cellules évolue vers l’état Epi, une autre vers l’état EPr alors que quelques-unes restent dans l’état MCI. La situation finale est représentée en C où le fond coloré reflète la concentration en FGF4 extracellulaire. Les cellules d’EPr (GATA6 >> NANOG) apparaissent en rouge, celles d’Epi (NANOG >> GATA6) en bleu et celles de la MCI (GATA6 ≈ NANOG) en gris. Epi : épiblaste ; EPr : endoderme primitif ; MCI : masse cellulaire interne. Les concentrations sont exprimées en unité arbitraire. |
Des simulations ont été réalisées avec 25 cellules placées dans une grille 5 × 5. Cette situation correspond au nombre de cellules composant la MCI et nous permet d’étudier la population cellulaire totale (Figure 4B, 4C), ce qui permet de reproduire plus fidèlement la situation qui prévaut in vivo au sein de la MCI, en termes de nombre de cellules et d’impact des interactions entre cellules environnantes. Une cinquième variable représentant la concentration extracellulaire de FGF4 a été ajoutée. Ainsi, la concentration de FGF4 n’est plus un paramètre fixé au début de la simulation comme précédemment, mais elle devient dynamique, fonction de la quantité de FGF4 produite par la cellule elle-même et par les cellules qui l’entourent. Au début de la simulation (Figure 4B), GATA6 et NANOG ne sont pas exprimés. À ce moment, la signalisation FGF/RTK est élevée en raison de la forte concentration de FGF4, en accord avec des mesures réalisées sur cellule unique [4]. Dès le début de la simulation, toutes les cellules commencent à co-exprimer GATA6 et NANOG. Elles tendent ainsi vers l’état stable « MCI ». Dans le même temps, la concentration extracellulaire en FGF4 diminue de manière hétérogène autour des cellules. Cette baisse de concentration s’explique par le fait que NANOG n’est pas assez exprimé par les cellules de la MCI pour permettre son maintien. En conséquence, l’expression de NANOG va augmenter dans certaines cellules. Elles vont changer d’état passant de l’état MCI vers l’état Epi. Ces cellules vont alors produire des quantités importantes de FGF4, provoquant une augmentation locale de sa concentration extracellulaire qui induit l’expression de GATA6 et la spécification en EPr des cellules de la MCI qui sont à proximité.
La spécification se fait donc par l’intermédiaire d’interactions locales, de proche en proche, qui mènent à l’établissement de deux populations de cellules, Epi et EPr, organisées en « poivre et sel ». Pour cela, l’élément fondateur et déclencheur est la spécification de certaines cellules de la MCI en Epi, ce qui a été confirmé in situ [12]. Ce caractère asynchrone, inhérent au modèle, ne nécessite qu’une seule cellule Epi spécifiée pour enclencher le système et obtenir le patron en « poivre et sel ». Pour fonctionner, ce modèle repose sur une hétérogénéité des taux de FGF4 extracellulaire. Cette hypothèse d’une hétérogénéité des taux de FGF4 pour l’organisation en « poivre et sel », a été renforcée par l’analyse des embryons de souris Fgf4-/-. Dans ces mutants, comme pour les embryons de souris Gata6-/-, toutes les cellules de la MCI expriment NANOG. Afin de restaurer l’organisation en « poivre et sel », des cultures en présence de FGF4 exogène ont été réalisées. Une concentration homogène de FGF4 permet de changer l’identité de toutes les cellules de la MCI en EPr, mais elle ne recrée pas le patron en « poivre et sel » [13, 14]. L’hétérogénéité du taux de FGF4 est donc un élément clé pour expliquer la spécification cellulaire en Epi et en EPr au sein de la MCI. En définitive, la modélisation mathématique reproduit les données biologiques observées in vivo. Le réseau de régulation génique NANOG/FGF4/GATA6/FGFR2 est donc suffisant pour expliquer la mise en place de la distribution « poivre et sel ».
La modélisation pour expliquer le comportement des mutants
Le modèle mathématique que nous avons développé a été utilisé pour simuler les interactions moléculaires dans les embryons mutants Gata6-/-. Il montre qu’en l’absence de ce facteur de transcription, toutes les cellules expriment fortement NANOG et atteignent l’état stable Epi plus précocement que ne le font les cellules Epi des embryons sauvages, les rendant insensibles vis-à-vis de la signalisation FGF/RTK. Cette insensibilité n’est pas le fait d’une action différentielle de la voie de signalisation au cours du temps, comme suggéré précédemment. Il s’agit en fait d’une détermination précoce des cellules Epi dans les embryons Gata6-/-. Ces résultats démontrent donc que l’expression de GATA6 retarde la spécification des cellules de l’Epi.
Le modèle a également permis d’expliquer le phénotype des embryons hétérozygotes Gata6+/-. Dans ces embryons, on observe une diminution importante du nombre de cellules de l’EPr à E3,75, sans que le nombre total de cellules de la MCI ne soit perturbé. C’est le choix du destin des cellules MCI, Epi ou EPr, qui est modifié, indiquant une anomalie lors de la spécification chez les embryons Gata6+/-. De plus, l’activation artificielle de la signalisation FGF/RTK chez ces embryons Gata6+/- montre, là-aussi, une insensibilité des cellules Epi dès le stade E3,25, similaire à celle observée dans les embryons mutants Gata6-/-. Or, les cellules hétérozygotes possèdent un allèle fonctionnel du gène Gata6. Grâce à la modélisation, il nous a été possible de simuler l’effet de cette haplo-insuffisance sur la spécification cellulaire. Nous avons mis en évidence qu’une diminution de seulement 15 % de l’activité de GATA6 permettait d’obtenir un biais important de la spécification avec des pourcentages Epi/EPr similaires à ceux observés in vivo. Comme précédemment, la spécification des cellules Epi hétérozygotes est plus précoce, comparée aux cellules Epi sauvages, d’où une insensibilité précoce au FGF4. La diminution relativement faible de l’activité GATA6 dans les embryons Gata6+/- a été vérifiée in vivo par la quantification de GATA6 et des transcrits Fgf4 [12]. Grâce à ces deux approches, nous avons pu démontrer que le contrôle de la balance GATA6/NANOG est primordial pour maîtriser la cinétique des événements lors de la spécification cellulaire de la MCI.
Conclusion : adéquation entre résultats obtenus in vivo et in silico
L’aide apportée par la modélisation mathématique à la compréhension des spécifications cellulaires précoces dans l’embryon est importante à plus d’un égard. Cette modélisation a tout d’abord permis de vérifier que le réseau de régulation génique NANOG/FGF4/GATA6/FGFR2 est non seulement requis, mais surtout suffisant, pour expliquer la mise en place d’une distribution en « poivre et sel » des deux types cellulaires au sein de la MCI. La modélisation se base sur un système déterministe, c’est-à-dire qu’une fois le système enclenché, l’évolution observée sera identique dans toutes les simulations réalisées. Néanmoins, le déclencheur du mécanisme, c’est-à-dire le facteur qui va promouvoir l’expression de NANOG dans une ou quelques cellules à E3,0, reste à découvrir. Nous avons postulé que la baisse hétérogène de la concentration de FGF4 extracellulaire pouvait jouer ce rôle. Cette hypothèse s’est vue renforcée par les résultats obtenus lors de l’analyse des mutants Fgf4 [13, 14]. Néanmoins, tout autre facteur stimulant l’expression de NANOG, y compris la variabilité stochastique des taux cellulaires initiaux (la valeur aléatoire du taux de NANOG dans chaque cellule), pourrait aussi expliquer le déclenchement du mécanisme de spécification de la MCI [15] (→).
(→) Voir la Nouvelle de P. Nghe et al., m/s n° 3, mars 2015, page 233
La combinaison des deux approches – modélisation mathématique et analyse biologique – démontre que la maîtrise temporelle de la plasticité cellulaire de l’Epi s’explique par un contrôle de la balance NANOG/GATA6 à des niveaux très précis. Néanmoins, le modèle actuel n’est pas suffisant pour expliquer le mécanisme de détermination cellulaire. Nous l’avons notamment observé lors de l’étude des embryons Gata6+/-. Les expériences in vivo montrent que les cellules Epi sont déterminées dès le stade E3,25, ce qui est en opposition avec les résultats obtenus avec le modèle mathématique actuel. La détermination cellulaire nécessite donc des facteurs supplémentaires, en aval de la spécification, pour prévenir tout changement d’identité.
En définitive, la coopération privilégiée entre les expériences et la modélisation a permis d’effectuer une avancée significative dans le domaine du développement précoce de la souris. La connaissance biologique du système d’intérêt est primordiale pour l’élaboration d’un modèle mathématique fiable. La modélisation peut, quant à elle, tester des hypothèses de travail plus rapidement que la biologie expérimentale. Ces prédictions théoriques doivent ensuite être validées par des expériences à la paillasse. En définitive, un usage généralisé de la modélisation en biologie serait un atout non négligeable pour accroître à la fois la productivité et la qualité scientifiques.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Merci à Claire Chazaud (GReD, Clermont-Ferrand) pour ses commentaires constructifs sur le manuscrit et à Laurane De Mot pour son rôle clé dans l’élaboration du modèle. G.D. est Maître de recherche au Fonds national de la recherche scientifique (FNRS, Belgique).
Glossaire
Spécification : la cellule acquiert un devenir cellulaire qui reste réversible
Détermination : la cellule spécifiée fixe de manière irréversible son devenir cellulaire
Maturation : la cellule déterminée va exprimer de nouveaux marqueurs spécifiques qui seront nécessaires pour atteindre ultérieurement un état différencié fonctionnel. La maturation n’entraîne pas de modification de la structure cellulaire ni l’apparition de nouvelles fonctions
Différenciation : la cellule acquiert une structure et des propriétés spécifiques. L’acquisition de ces nouvelles caractéristiques lui permettra d’effectuer des fonctions particulières
Références
- Chazaud C. L’embryogenèse précoce des mammifères : premières différenciations cellulaires et cellules souches. Med Sci (Paris) 2008 ; 24 : 1043–1048. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Sasaki H. Position- and polarity-dependent Hippo signaling regulates cell fates in preimplantation mouse embryos. Semin Cell Dev Biol 2015 ; 47–48 : 80–87. [Google Scholar]
- Chazaud C, Yamanaka Y, Pawson T, Rossant J. Early lineage segregation between epiblast and primitive endoderm in mouse blastocysts through the Grb2-MAPK pathway. Dev Cell 2006 ; 10 : 615–624. [CrossRef] [PubMed] [Google Scholar]
- Guo G, Huss M, Tong GQ, et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev Cell 2010 ; 18 : 675–685. [CrossRef] [PubMed] [Google Scholar]
- Nichols J, Silva J, Roode M, Smith A. Suppression of Erk signalling promotes ground state pluripotency in the mouse embryo. Development 2009 ; 136 : 3215–3222. [CrossRef] [PubMed] [Google Scholar]
- Yamanaka Y, Lanner F, Rossant J. FGF signal-dependent segregation of primitive endoderm and epiblast in the mouse blastocyst. Development 2010 ; 137 : 715–724. [CrossRef] [PubMed] [Google Scholar]
- Chambers I, Silav J, Colby D, et al. Nanog safeguards pluripotency and mediates germline development. Nature 2007 ; 450 : 1230–1234. [CrossRef] [PubMed] [Google Scholar]
- J. Silva J, Nichols J, Theunissen TW, et al. Nanog is the gateway to the pluripotent ground state. Cell 2009 ; 138 : 722–737. [CrossRef] [PubMed] [Google Scholar]
- Cai KQ, Capo-Chichi CD, Rula ME, et al. Dynamic GATA6 expression in primitive endoderm formation and maturation in early mouse embryogenesis. Dev Dyn 2008 ; 237 : 2820–2829. [CrossRef] [PubMed] [Google Scholar]
- Frankenberg S, Gerbe F, Bessonnard S, et al. Primitive endoderm differentiates via a three-step mechanism involving Nanog and RTK signaling. Dev Cell 2011 ; 21 : 1005–1013. [CrossRef] [PubMed] [Google Scholar]
- Messerschmidt DM, Kemler R. Nanog is required for primitive endoderm formation through a non-cell autonomous mechanism. Dev Biol 2010 ; 344 : 129–137. [CrossRef] [PubMed] [Google Scholar]
- Bessonnard S, De Mot L, Gonze D, et al. Gata6, Nanog and Erk signaling control cell fate in the inner cell mass through a tristable regulatory network. Development 2014 ; 141 : 3637–3648. [CrossRef] [PubMed] [Google Scholar]
- Kang M, Piliszek A, Artus J, Hadjantonakis AK. FGF4 is required for lineage restriction and salt-and-pepper distribution of primitive endoderm factors but not their initial expression in the mouse. Development 2013 ; 140 : 267–279. [CrossRef] [PubMed] [Google Scholar]
- Krawchuk D, Honma-Yamanaka N, Anani S. Yamanaka Y. FGF4 is a limiting factor controlling the proportions of primitive endoderm and epiblast in the ICM of the mouse blastocyst. Dev Biol 2013 ; 384 : 65–71. [CrossRef] [PubMed] [Google Scholar]
- Nghe P, Boulineau S, Tans SJ. Fluctuations aléatoires dans le métabolisme et la croissance cellulaires. Med Sci (Paris) 2015 ; 31 : 233–235. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Représentation schématique du développement préimplantatoire chez la souris. MCI : masse cellulaire interne. |
| Dans le texte | |
 |
Figure 2. Détection par immunofluorescence de NANOG, GATA6 et CDX2 (caudal type homeobox 2) au stade E3,75 dans les embryons sauvages, Nanog-/- et Gata6-/-. Dans les embryons Nanog-/- ou GATA6-/- (GATA binding protein 6), l’expression en « poivre et sel » disparaît, faisant place à des MCI composées entièrement d’EPr (exprimant GATA6) ou d’Epi (exprimant NANOG). CDX2 est utilisé pour repérer le tropherdoderme. Epi : épiblaste ; EPr : endoderme primitif ; MCI : masse cellulaire interne. |
| Dans le texte | |
 |
Figure 3. Représentation du réseau de régulation génique de la spécification. Les principaux acteurs sont les facteurs de transcription NANOG et GATA6 (GATA binding protein 6), le récepteur FGFR2 (fibroblast growth factor receptor 2), son ligand FGF4 (fibroblast growth factor 4) et la voie de signalisation MEK (MAPK/ERK kinase)/ERK (extracellular signal-regulated kinases). La sécrétion de FGF4 dans le milieu extracellulaire permet la communication intercellulaire. La concentration de FGF4 dépend de sa production par la cellule (N5) et de ses quatre cellules voisines (N1 à N4). L’intensité de la couleur violette reflète la concentration du FGF4 (adapté de Bessonnard et al. [12]). |
| Dans le texte | |
 |
Figure 4. Résultats de la modélisation. Le panneau A décrit le comportement du réseau de régulation génétique associé à une cellule isolée, tandis que les panneaux B et C illustrent le comportement d’une population de 25 cellules. A. Les courbes de GATA6 en fonction de la concentration en FGF4 montrent les états stables possibles de la cellule. Selon les concentrations faibles ou élevées de FGF4, seul l’état Epi (concentration faible) ou l’état EPr (concentration élevée) est possible, comme illustré sur les panneaux a et c. Pour des concentrations intermédiaires, la cellule peut évoluer vers l’un des trois états stables en fonction des conditions initiales (panneau b). B. Les trajectoires de 25 cellules dont l’évolution est régie par le même réseau de régulation génétique, mais où la sécrétion de FGF4 par les cellules exprimant NANOG est prise en compte, sont montrées. Une sous-population de cellules évolue vers l’état Epi, une autre vers l’état EPr alors que quelques-unes restent dans l’état MCI. La situation finale est représentée en C où le fond coloré reflète la concentration en FGF4 extracellulaire. Les cellules d’EPr (GATA6 >> NANOG) apparaissent en rouge, celles d’Epi (NANOG >> GATA6) en bleu et celles de la MCI (GATA6 ≈ NANOG) en gris. Epi : épiblaste ; EPr : endoderme primitif ; MCI : masse cellulaire interne. Les concentrations sont exprimées en unité arbitraire. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




