| Issue |
Med Sci (Paris)
Volume 31, Number 3, Mars 2015
|
|
|---|---|---|
| Page(s) | 282 - 290 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20153103014 | |
| Published online | 08 April 2015 | |
La synthèse des protéines par le ribosome
Un chemin semé d’embûches
Protein synthesis by the ribosome: a pathway full of pitfalls
1
Université de Rennes 1, CNRS UMR 6290 IGDR, équipe traduction et repliement, Campus de Beaulieu, Bâtiment 13, 35042 Rennes, France
2
Institut universitaire de France
*
reynald.gillet@univ-rennes1.fr
La synthèse des protéines, également appelée traduction, est assurée dans chaque cellule par des machines moléculaires très sophistiquées : les ribosomes. Compte tenu de l’immense quantité de données biologiques à traiter, il arrive régulièrement que ces machines se bloquent et mettent en péril la survie de la cellule. Chez les bactéries, le principal processus de sauvetage des ribosomes bloqués est la trans-traduction. Il est assuré par un acide ribonucléique (ARN) hybride, l’ARN transfert-messager (ARNtm), associé à une petite protéine basique, SmpB (small protein B). Plusieurs autres systèmes de contrôle qualité ont récemment été mis en évidence, révélant un réseau de maintien de la survie cellulaire très sophistiqué. Cette machinerie du contrôle qualité de la synthèse protéique est une cible très prometteuse pour le développement de futurs antibiotiques.
Abstract
Protein synthesis is accomplished through a process known as translation and is carried out by the ribosome, a large macromolecular complex found in every living organism. Given the huge amount of biological data that must be deciphered, it is not uncommon for ribosomes to regularly stall during the process of translation. Any disruption of this finely tuned process will jeopardize the viability of the cell. In bacteria, the main quality-control mechanism for rescuing ribosomes that undergo arrest during translation is trans-translation, which is performed by transfer-messenger RNA (tmRNA) in association with small protein B (SmPB). However, other rescue systems have been discovered recently, revealing a far more complicated network of factors dedicated to ribosome rescue. These discoveries make it possible to consider inhibition of these pathways as a very promising target for the discovery of new antibiotics.
© 2015 médecine/sciences – Inserm
La traduction : un équilibre délicat entre vitesse et fidélité
L’ADN est le support de l’information génétique chez tous les êtres vivants. Il est fabriqué à partir d’un alphabet de seulement quatre lettres (A, T, G, C), les nucléotides. L’enchaînement de ces nucléotides forme le génome, dont une partie est traduite sous forme de protéines, nécessaires à la vie. L’information portée par l’ADN est d’abord transcrite en un ARN messager (ARNm), également fabriqué à partir de quatre nucléotides (A, U, G, C). Cette première étape, nommée transcription, permet d’amplifier le message de départ tout en préservant l’ADN de la chimie caustique de la cellule. Les ARNm sont ensuite traduits par triplets de nucléotides (ou codons) en l’un des vingt-deux acides aminés composant les protéines. Cette synthèse protéique, ou traduction, permet le passage d’un alphabet de quatre lettres (les nucléotides qui composent l’ADN et l’ARNm) à un alphabet de vingt-deux lettres (les acides aminés qui composent les protéines). Elle est portée par une machine moléculaire très complexe et dynamique : le ribosome [1]. Les ribosomes sont constitués de deux sous-unités distinctes, elles-mêmes composées d’ARN ribosomiques (ARNr) et de protéines. La petite sous-unité permet le décodage de l’information génétique portée par les ARNm, tandis que la grande sous-unité est le siège de la synthèse des protéines via la catalyse des liaisons entre acides aminés (les liaisons peptidiques). Le ribosome possède trois sites de liaison aux ARN de transfert (ARNt), chargés de lui apporter les acides aminés. Le site A, auquel les ARNt aminoacylés se lient après reconnaissance spécifique des codons, le site P, qui est occupé par un ARNt lié à la chaîne polypeptidique naissante, et le site E, par lequel les ARNt déacylés transitent avant d’être éjectés.
La traduction s’effectue en quatre étapes majeures (Figure 1) : le démarrage (ou initiation), l’élongation, la terminaison et le recyclage (pour une description détaillée du processus voir [2]). Chez les bactéries, le démarrage débute par la fixation de l’ARNm à la petite sous-unité ribosomique 30S, via une séquence riche en purines, dite de Shine-Dalgarno (SD). Cette fixation, assistée par trois facteurs d’initiation (IF1, IF2 et IF3), permet de placer précisément le codon de démarrage (AUG, ou, moins communément, GUG ou UUG) dans le site P du ribosome, afin qu’il soit reconnu par un ARNt porteur de l’acide aminé formyl-méthionine. L’arrimage de la grande sous-unité 50S à ce complexe de démarrage permet de reconstituer le ribosome complet 70S, signant ainsi le début de la phase d’élongation. Dans un processus itératif, les codons de l’ARNm sont ensuite décodés dans le site A de la petite sous-unité. S’ensuit un transfert de la chaîne peptidique de l’ARNt occupant le site P vers l’ARNt aminoacylé du site A, ajoutant ainsi un acide aminé à la protéine en cours de synthèse. Après ce transfert peptidique, l’ARNt déacylé (dans le site P) et l’ARNt porteur du peptide (dans le site A) sont transloqués avec leurs codons correspondants vers les sites E et P, respectivement. Les facteurs d’élongation EF-G et EF-Tu permettent de faciliter le processus. La traduction se termine lorsqu’un codon de terminaison (ou codon stop ; UAA, UAG ou UGA) est rencontré sur l’ARNm. Ce codon est reconnu dans le site A par un facteur de terminaison (RF1 [release factor] ou RF2) qui provoque la libération de la protéine nouvellement synthétisée. Les ribosomes sont recyclés dans une ultime étape par la fixation d’un facteur de recyclage RRF (ribosome recycling factor) (pour une revue complète voir [3]). Ce processus est très dynamique et implique de nombreux partenaires protéiques et ARN se liant et se détachant continuellement du ribosome. Il permet ainsi un équilibre entre rapidité (15 à 20 liaisons peptidiques à la seconde) et fidélité au code génétique (une erreur d’incorporation environ tous les 1 000 à 10 000 acides aminés) [4, 5]. Compte tenu de la taille moyenne des protéines chez E. coli (environ 300 acides aminés), ce faible taux d’erreurs assure à quasiment toutes les protéines produites une bonne activité. Les cellules possèdent un large répertoire de systèmes de contrôle qualité permettant d’affronter les nombreuses situations, notamment de stress, susceptibles de rompre ce délicat équilibre entre vitesse de synthèse et fidélité au code génétique.
 |
Figure 1. Présentation sommaire de la traduction canonique bactérienne. Démarrage : après appariement entre l’ARNm et le 30S via des séquences complémentaires d’ARN (interactions Shine-Dalgarno/anti-Shine-Dalgarno), le codon d’initiation AUG (voire GUG ou UUG) est positionné dans le site P. Un complexe initiateur, constitué de l’ARNt-fMet, d’IF2 et de GTP, s’y fixe, en présence des deux autres facteurs d’initiation, IF1 et IF3. La grande sous-unité ribosomique 50S se lie alors au complexe, provoquant l’hydrolyse du GTP et le départ des facteurs d’initiation. élongation : le facteur d’élongation EF-Tu amène les ARNt aminoacylés (aa-ARNt) au site A du ribosome sous la forme d’un complexe ternaire aa-ARNt-EF-Tu·GTP. S’il y a correspondance entre le codon de l’ARNm et l’anticodon de l’ARNt, EF-Tu hydrolyse le GTP et se dissocie, tandis que l’ARNt pénètre profondément dans le site A (étape d’accommodation). Le peptide naissant est transféré à l’ARNtaa accommodé dans le site A, entraînant l’allongement du peptide d’un acide aminé. Par un mouvement de cliquet spontané (ratchet) entre ses deux sous-unités, le ribosome oscille alors entre deux conformations. Le facteur d’élongation EFG- GTP se lie alors au ribosome, et l’hydrolyse du GTP permet la translocation des ARNt du site A au site P, et du site P au site E. S’ensuit le départ d’EF-G-GDP. Le processus est itératif, ce qui provoque l’allongement séquentiel du peptide naissant, tandis que les ARNt se déplacent ° travers les sites ribosomiques A, P et E. Terminaison : lorsqu’un codon de terminaison (ou codon stop UAA, UAG ou UGA) atteint le site A, un facteur de libération de classe I (RF1 ou RF2) se lie et catalyse la séparation entre la chaîne polypeptidique et l’ARNt en place dans le site P. La protéine néosynthétisée est libérée du ribosome, après prise en charge par des machines de repliement co-traductionnel (MRC). Un complexe binaire composé de RF3, un facteur de libération de classe II, et d’une molécule de GTP, facilite la dissociation de RF1 ou RF2 et de l’ARNt déacylé. Recyclage : le facteur de recyclage ribosomique (RRF) associé à EF-G recycle le ribosome en séparant les deux sous-unités, libérant les ARNt et l’ARNm. Un nouveau cycle de traduction peut alors démarrer. aa-ARNt : ARN de transfert aminoacylé ; ARNm : ARN messager ; fMet-ARNt : ARN de transfert N-formylméthionine ; EF-G : facteur d’élongation G ; GDP : guanosine diphosphate ; GTP : guanosine-5’-triphosphate ; EF-Tu : facteur d’élongation thermosensible ; IF : facteur d’initiation ; MRC : machine de repliement co-traductionnel ; RF : facteur de libération ; RRF : facteur de recyclage ribosomique ; SD : séquence de Shine-Dalgarno ; 30S : petite sous-unité ribosomique ; 50S : grande sous-unité ribosomique. |
Cette revue vise à mieux appréhender la manière dont les bactéries affrontent en permanence le blocage ou le ralentissement des ribosomes sur leur ARNm. Les stratégies de sauvetage sont redoutablement efficaces et font de ces systèmes de contrôle qualité une cible particulièrement attractive pour le développement de futurs antibiotiques.
Quand la traduction se grippe…
Les ribosomes avancent à pleine vitesse sur les ARNm afin de les décoder. Deux situations majeures provoquent cependant un arrêt brutal ou un ralentissement conséquent du processus (Figure 2) :
 |
Figure 2. Illustration des différentes situations conduisant à la production de complexe non-stop en haut et de complexe no-go en bas. Les sous-unités 30S et 50S sont en jaune et bleu clair, respectivement. L’ARNm est en bleu foncé (les codons rares en marron et le codon stop en rouge). Le peptide naissant est en violet. L’ARNt du site P est en vert, du site E en orange, les ARNt déacylés en rouge et les ARNt suppresseurs en rose pâle. Les ions Mg2+ sont symbolisés par des points rouges et les antibiotiques par des triangles. Les endo- et exonucléases sont respectivement représentées par une paire de ciseaux et un symbole pac-man ; le stress oxydant par un éclair jaune. |
(1) Lorsque les ribosomes atteignent la fin de l’ARNm sans avoir rencontré de signal de terminaison ; on parle d’ARNm non-stop. Cette situation est généralement causée par l’altération de l’ARNm qui perd son signal de terminaison. Plusieurs raisons à cela : une mutation, une dégradation partielle de l’ARNm (ribonucléases, stress oxydant, etc.), une erreur de lecture due à un antibiotique, un cadre de lecture aberrant ou, plus rarement, quand un codon stop est traduit à tort. Le ribosome se retrouve alors bloqué à l’extrémité 3’ de l’ARNm, avec un codon imparfait (zéro, un ou deux nucléotides au lieu de trois) dans son site de décodage ( Figure 2 , haut).
(2) Lorsque les ribosomes s’arrêtent ou ralentissent fortement en cours de traduction, avant que le codon stop soit atteint, on parle alors d’ARNm no-go. Cette situation se produit lorsque le ribosome synthétise des protéines à partir de séries de codons rares (définis par une fréquence inférieure à 1 % chez E. coli [6]), lorsque les protéines portent des motifs peptidiques bloquants, en conditions de faible croissance (par exemple en cas de déficit en acides aminés), en présence de fortes concentrations de Mg2+, en présence d’inhibiteurs du ribosome (par exemple des antibiotiques bloquant le transfert peptidique ou la translocation), ou encore lorsque l’ARNm porte une séquence de type Shine-Dalgarno capable de s’apparier à l’ARNr de la petite sous-unité ( Figure 2 , bas).
Les deux situations non-stop et no-go sont fréquentes et conduisent à la formation de complexes non productifs composés de ribosomes bloqués, de protéines incomplètes, d’ARNt et d’ARNm. Ces blocages sont graves pour la survie des bactéries, car ils mobilisent une partie du pool de ribosomes, tout en entraînant la synthèse de protéines incomplètes et potentiellement toxiques si elles venaient à être libérées.
Blocage des ribosomes sur des ARNm non-stop
Parvenus à l’extrémité d’un ARNm sans avoir subi l’étape de terminaison, les ribosomes s’accumulent sur l’ARNm en formant des polysomes et bloquent ainsi la machinerie traductionnelle. Trois systèmes de contrôle qualité principaux permettent de rapidement régler ce problème chez la bactérie : la trans-traduction, ainsi que les deux systèmes alternatifs ArfA (alternative ribosome rescue factor A) et ArfB.
La trans-traduction, un système tout-en-un
Chez les bactéries, le principal mécanisme de contrôle qualité permettant de libérer les ribosomes bloqués est la trans-traduction, portée par un ARN particulier : l’ARN transfert-messager (ARNtm) associé à une petite protéine, SmPB (small protein B). L’ARNtm est une molécule hybride possédant à la fois les propriétés d’un ARN de transfert et d’un ARN messager ( Figure 3 ). Il possède donc deux domaines importants ayant pour nom le TLD (tRNA-like domain) et le MLD (mRNA-like domain). Le domaine TLD se lie à la protéine SmPB afin de maintenir sa conformation tridimensionnelle et mimer un ARNt canonique. SmPB permet la distinction entre les ribosomes bloqués ou non en insérant et structurant sa partie carboxy-terminale en fonction de la présence ou de l’absence d’un ARNm dans le canal de lecture du ribosome [7]. Malgré l’absence d’un codon dans le site A, cette partie TLD-SmPB entre dans le ribosome tel un ARNt canonique. Le domaine MLD, correctement positionné grâce à SmPB [8, 9], est aussitôt traduit. Il permet ainsi de redémarrer le processus traductionnel, tout en codant pour un signal marquant le peptide incomplet. Cette étiquette sera spécifiquement reconnue par des protéases qui dégraderont le peptide. La séquence codante du MLD se termine par un codon stop, de telle sorte que le peptide nouvellement marqué est libéré, et le ribosome recyclé. Ainsi, dans un ballet sophistiqué, ce complexe tout-en-un éjecte l’ARNm défectueux du ribosome pour le conduire à sa dégradation par la ribonucléase R, marque la protéine naissante pour une dégradation rapide par les protéases, et enfin permet le recyclage des ribosomes bloqués ( Figure 4 ) (pour une revue complète voir [10]).
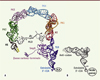 |
Figure 3. Comparaison des structures tridimensionnelles du complexe ARNtm-SmPB et d’un ARNt. A. Complexe ARNtm-SmPB. SmPB est en violet et les domaines de l’ARNtm sont représentés dans différentes couleurs : les deux domaines principaux, le TLD en bleu et le MLD en gris, allant jusqu’au codon stop représenté en jaune. H : hélices ; PK : pseudo-nœuds. B. Un ARNt canonique afin de comparer la similitude structurale entre le complexe ARNtm-SmPB et un ARNt. Les deux extrémités 3’-CCA sont liées à un acide aminé (pour l’ARNtm, c’est toujours une alanine). |
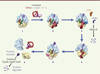 |
Figure 4. Schéma des différentes étapes de la trans-traduction. (1) Ribosome bloqué lors de la traduction d’un ARNm défectueux (bleu) avec un ARNt dans le site E (orange) et un ARNt dans le site P (vert) lié à la protéine naissance (violet). Le site A est vide. (2) Le complexe ARNtm-SmPB-EF-Tu reconnaît le ribosome bloqué. (3) Aidé par EF-G (bleu ciel), le complexe est transloqué dans le ribosome et l’ARNm tronqué est éjecté pour être dégradé par la RNase R. (4) Le peptide incomplet est transféré sur l’ARNtm et la traduction redémarre sur sa partie codante (MLD). (5) Il en résulte l’addition d’une étiquette peptidique à la protéine incomplète. L’ARNtm est translucide car les détails structuraux de cette étape ne sont pas encore connus. (6) Le ribosome bloqué est recyclé grâce à la présence d’un codon de terminaison sur la séquence codante de l’ARNtm, tandis que le peptide étiqueté est dégradé par des protéases. |
Il y a environ 700 molécules d’ARNtm par cellule, ce qui correspond à un rapport de 1 pour 10-20 ribosomes, et la trans-traduction survient au moins une fois toutes les 250 traductions [11, 12]. À l’exception de quelques rares organismes primitifs (certaines algues et diatomées), la trans-traduction est exclusivement retrouvée dans le monde bactérien. Bien que confrontées à la même nécessité de contrôle qualité de la synthèse protéique, les cellules eucaryotes sont dépourvues d’ARNtm et de SmPB. Les systèmes de surveillance mis à leur disposition ont évolué différemment, faisant appel à des mécanismes très distincts [13, 14].
La trans-traduction n’est pas toujours indispensable à la survie cellulaire
Bien que l’ARNtm et SmPB soient toujours présents chez les bactéries, la perturbation du système n’est pas toujours létale. L’explication de ce paradoxe est venue de la récente découverte de deux autres systèmes de secours chez certaines bactéries (y compris Escherichia coli) : ArfA (alternative ribosome rescue factor A) et ArfB, anciennement YhdL et YaeJ, respectivement ( Figure 5 ).
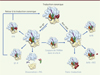 |
Figure 5. Illustration des différents systèmes de sauvetage contre le blocage des ribosomes bactériens. Pendant la traduction, le ribosome peut se gripper de deux façons différentes. On parle de no-go lorsqu’il ralentit ou s’arrête avant que le codon d’arrêt ne soit atteint, ou de non-stop lorsqu’il atteint l’extrémité 3’ d’un ARNm. Les complexes ribosomiques no-go peuvent retourner à la traduction canonique grâce à EF-P, qui aide la lecture des séquences riches en codons proline, ou grâce à EF4, qui remobilise les ribosomes lorsque la concentration intracellulaire en Mg2+ est élevée. Ces mêmes complexes peuvent être recyclés par la PTH qui sépare le peptide naissant lié à l’ARNt lorsque le ribosome est dissocié. Enfin, ces complexes peuvent être convertis en non-stop lorsque l’ARNm est clivé dans le site A. Les complexes non-stop peuvent être sauvés par trans-traduction, par ArfA, en coopération avec RF2, ou par ArfB. Entrées PDB 3HUW pour EF-P ; 3DEG pour EF4 ; 3VJR pour PTH ; 3KIQ pour RelE ; 2WH1 pour RF2 ; 4DH9 pour ArfB ; et 3IYQ pour ARNtm-SmPB. La structure ArfA a été modélisée avec I-TASSER (iterative threading assembly refinement). Son positionnement exact dans le ribosome est encore inconnu. Les éléments des systèmes de sauvetage sont en rouge. Les autres codes couleurs sont les mêmes que ceux employés dans la Figure 4 . |
En effet, ArfA est une petite protéine qui prend en charge le sauvetage des ribosomes bloqués en l’absence de trans-traduction. ArfA coopère avec le facteur de terminaison RF2 pour que ce dernier relâche le peptide incomplet bloqué dans le site P [15]. Le positionnement d’ArfA dans le ribosome en présence de RF2 n’est pas encore connu, mais un scénario est proposé : de façon similaire à SmPB, ArfA pourrait se loger dans le site A vacant du ribosome pendant ou juste avant que RF2 ne déclenche la libération du peptide bloqué. La régulation d’ArfA est particulièrement intéressante puisque son ARNm induit la trans-traduction. Lorsque l’ARNtm et SmPB sont actifs, ils dégradent ArfA en ajoutant le signal de dégradation à la protéine et son ARNm est détruit. En revanche, si la trans-traduction devient inefficace, ArfA n’est plus trans-traduit, et il peut alors prendre le relais [16, 17].
Le second système alternatif, ArfB, est une petite protéine mimant le domaine catalytique de RF2. Contrairement à ArfA, il possède une queue carboxy-terminale qui rentre dans le canal de l’ARNm de la petite sous-unité, permettant ainsi la discrimination entre les ribosomes actifs et bloqués, tout comme le fait SmPB. Il est très certainement moins important que la trans-traduction et ArfA, puisque la double suppression des gènes qui codent pour l’ARNtm et ArfA est mortelle chez E. coli, ce qui n’est pas le cas pour la double mutation ARNtm/ArfB [18, 19].
Pour résumer, la trans-traduction est la première ligne de défense permettant aux bactéries de lutter contre l’accumulation des ribosomes sur des ARNm non-stop. Cependant, chez certaines bactéries, d’autres systèmes de sauvetage moins sophistiqués peuvent partiellement prendre le relais du complexe ARNtm/SmPB quand celui-ci est défaillant ou dépassé [20].
Blocage des ribosomes sur des ARNm no-go
Plusieurs systèmes de contrôle qualité permettent le sauvetage des ribosomes bloqués sur des complexes no-go. En fonction du problème rencontré, la bactérie adapte une réponse spécifique ( Figure 4 ).
Remobilisation ou dissociation des ribosomes bloqués en no-go
Lors d’une trop forte concentration cellulaire en Mg2+, l’étape de translocation dépendante d’EF-G est déficiente, entraînant une accumulation de ribosomes bloqués. Chez E. coli, le facteur d’élongation 4 (EF4, anciennement LepA) est stocké dans les membranes, mais il est libéré dans le cytoplasme en conditions de force ionique élevée. EF4 reconnaît alors les ribosomes bloqués et restaure l’étape de translocation déficiente [21].
Les séquences protéiques riches en prolines aboutissent également à des complexes no-go. En raison de leur nature chimique, les prolines sont en effet de faibles donneurs et accepteurs de peptides. Intervient alors EF-P, un facteur de traduction identifié depuis près de 40 ans, mais dont le rôle biologique exact était jusqu’à récemment inconnu. Il vient d’être démontré qu’il soulage les ribosomes bloqués sur des portions peptidiques riches en prolines en stimulant la formation de liaison peptidique entre deux prolines [22, 23]. Cette situation est fréquente chez E. coli qui possède jusqu’à 270 protéines contenant des successions de plus de trois prolines consécutives. Chez les eucaryotes, la lecture de ces motifs polyprolines est promue par le facteur d’initiation eIF5A (eukaryotic initiation factor 5A), orthologue d’EF-P.
Tout comme EF-P, une nouvelle fonction de la peptidyl-ARNt hydrolase (PTH) a récemment été découverte [24]. La PTH est une estérase qui hydrolyse la liaison ester entre le peptide et l’ARNt. Cette réaction permet de libérer l’ARNt, qui peut être réutilisé pour la synthèse protéique. Au-delà de son rôle traditionnellement attribué, et en collaboration avec plusieurs facteurs de la synthèse protéique (IF1, IF3, EF-G, RF1, RF2, RF3 et RRF), la PTH est capable de libérer un ribosome bloqué avec un peptide-ARNt dans le site P, en hydrolysant la liaison ester entre le peptide naissant et l’ARNt. Toutefois, la coopération entre ces facteurs et PTH dans le cas d’un peptidyl-ARNt bloqué est encore controversée [25].
Lorsque le no-go devient non-stop
Si aucun retour à la traduction canonique ne se produit, les ribosomes bloqués en no-go sont transformés en non-stop, afin d’être secourus par les systèmes vus précédemment. Cette situation est notamment retrouvée lors de la réponse « stringente », par activation de toxines bactériennes [26]. L’exemple qui illustre le mieux ce cas est le système toxine antitoxine RelBE. L’opéron relBE code pour une toxine stable RelE et son antitoxine instable RelB. RelB enveloppe la toxine RelE, bloquant son activité RNase [27]. RelB est instable, en raison de sa dégradation par la protéase Lon, et doit être continuellement synthétisée afin d’empêcher RelE d’agir. En cas de carence en acides aminés, les ribosomes se bloquent par la liaison d’ARNt non chargés dans leur site A, et l’opéron relBE est régulé à la baisse, entraînant une diminution de RelB et une libération de RelE. Une fois disponible, RelE se loge dans le site A et clive spécifiquement les ARNm, convertissant ainsi les complexes no-go en non-stop [28, 29]. Ces derniers peuvent alors être pris en charge par la trans-traduction, ArfA ou ArfB, comme décrit précédemment. À noter que d’autres attaques, que ce soit par d’autres toxines comme MazF ou bien des ribonucléases encore inconnues, peuvent jouer le même rôle et induire le clivage de l’ARNm dans le site A ( Figure 5 ).
Intérêts thérapeutiques
Les systèmes de sauvetage de la traduction forment un réseau de régulation indispensable à la vie des bactéries. De fait, ils représentent des cibles très attractives pour des applications biotechnologiques et médicales. Parmi tous les systèmes décrits ici, la trans-traduction fait l’objet d’une attention particulière et de recherches croissantes. Après le temps de la génétique, de la biochimie et de la structure, qui a permis de comprendre la trans-traduction à l’échelle moléculaire, vient en effet celui des applications. On distinguera l’utilisation de ce système comme cible pour de futurs inhibiteurs de la synthèse protéique bactérienne, pour le développement de vaccins vivants atténués, ou encore comme outils de diagnostic pour l’identification de bactéries.
La trans-traduction, cible pour des antibiotiques inhibiteurs de la synthèse protéique
Les antibiotiques sont des molécules permettant d’attaquer spécifiquement les bactéries, en bloquant leur croissance (antibiotiques bactériostatiques) ou en les détruisant (antibiotiques bactéricides). Ils agissent en inhibant une étape essentielle du développement bactérien : fabrication de leur paroi, de leur ADN, de leur ARN, ou encore de leurs protéines. Malheureusement, l’usage intensif des antibiotiques a rapidement introduit une pression de sélection aboutissant au développement inquiétant de populations de micro-organismes antibiorésistants et à une baisse générale de l’efficacité thérapeutique. L’émergence de ces résistances antimicrobiennes, couplées à la pauvreté actuelle du nombre de nouvelles molécules antibiotiques en cours de développement, font de ce problème l’un des principaux enjeux de santé de notre siècle. C’est pourquoi la découverte de nouvelles cibles constitue un enjeu majeur de santé publique.
Compte tenu de l’absence de trans-traduction chez les eucaryotes [30], le couple ARNtm-SmPB est une cible particulièrement attractive pour de nouveaux antibiotiques à large spectre ou pour augmenter l’activité des inhibiteurs actuels de la synthèse protéique (qui représentent la moitié des antibiotiques utilisés en clinique) [31, 32]. En effet, la trans-traduction est essentielle à la survie de nombreuses bactéries pathogènes (Mycobacterium tuberculosis, Neisseria gonorrhoeae, Helicobacter pylori, ou Shigella flexneri) et elle est requise pour la virulence d’autres espèces (Staphylococcus aureus, Salmonella typhimurium ou Yersinia pestis) [33]. Lorsqu’elle n’est pas létale, la délétion de l’ARNtm ou de SmPB entraîne l’apparition de phénotypes hypersensibles chez de nombreuses espèces bactériennes (par exemple Cyanobacterium Synechocystis sp. strain PCC6803, Escherichia coli, ou Salmonella typhimurium) [34, 35]. Ces mutants ne sont pas viables en présence de faibles doses d’inhibiteurs de la synthèse protéique (chloramphénicol, lincomycine, spiramycine, tylosine, érythromycine, ou spectinomycine). Cela indique que la trans-traduction libère les ribosomes bloqués par ces antibiotiques, et que l’inhibition de l’ARNtm ou de SmPB permettrait d’augmenter fortement l’efficacité de ces antibiotiques couramment employés (effet synergique) [36].
La preuve de concept existe puisqu’il a récemment été démontré que la pyrazinamide, traitement de référence et de première intention contre la tuberculose depuis 60 ans et dont on ne connaissait jusqu’alors pas le mode d’action [37], inhibe la trans-traduction. Cet antibiotique cible uniquement la trans-traduction et non la traduction canonique de M. tuberculosis [38]. Enfin, un criblage réalisé à partir de 663,000 molécules a récemment permis de révéler de nouvelles molécules capables d’inhiber cette voie, ayant un effet bactéricide et à spectre large [39]. Ces travaux valident l’inhibition de cette voie comme une piste majeure de recherche de futurs antibiotiques.
Des mutants ARNtm-SmPB comme vaccins vivants atténués
L’altération de la trans-traduction conduit à une grande variété de phénotypes, selon les espèces bactériennes touchées. Cependant, il est à noter qu’elle peut conduire à l’inhibition de la virulence de plusieurs agents pathogènes, comme Yersinia pestis (l’agent causal de la peste) ou Francisella tularensis (l’agent étiologique de la tularémie). Ainsi, des mutants atténués, dont les gènes codant pour SmPB ou l’ARNtm sont supprimés, induisent une forte réponse immunitaire chez la souris [40]. La mutation spécifique de la voie de trans-traduction est donc particulièrement prometteuse pour le développement de vaccins vivants atténués.
Utilisation des séquences d’ARNtm à des fins diagnostiques
Les séquences d’ARN ribosomiques (notamment l’ARNr 16S, issu de la petite sous-unité) sont couramment utilisées pour l’identification in situ des bactéries. Pourtant, dans certains cas, les comparaisons d’ARNr ne suffisent pas à distinguer des bactéries phylogénétiquement liées. L’ARNtm peut alors représenter une alternative efficace. En effet, les séquences hautement conservées des deux extrémités 5’ et 3’ de l’ARNtm, couplées à sa petite taille, permettent la conception d’amorces universelles qui permettent d’amplifier aisément la partie centrale des gènes codant pour l’ARNtm, dont la divergence est a contrario considérable entre les espèces [41, 42]. La méthode a été employée avec succès pour discriminer de nombreuses espèces bactériennes, que ce soit dans des échantillons biologiques humains ou divers aliments [43].
Conclusion
Les systèmes de sauvetage des ribosomes renferment un côté prodigieux que l’on n’attendait pas. Ils rythment la vie cellulaire tout en étant les mécaniciens de l’indispensable machinerie ribosomique. Alors qu’apparaissent de plus en plus de souches multi-résistantes contre lesquelles nous ne disposons d’aucun traitement efficace, la trans-traduction, universellement conservée dans le monde bactérien, mais étrangement absente des cellules eucaryotes, est une cible très prometteuse pour le développement de futurs antibiotiques. Ce fabuleux mécanisme n’a donc pas fini de nous surprendre ; nul ne doute que des années fastes en termes de développement médical parsèmeront encore la voie de synthèse des protéines par le ribosome, une voie qui n’a pas fini de rencontrer des embûches.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Les auteurs remercient Rennes Métropole, la Ligue contre le cancer, et l’Agence nationale de la recherche (programmes JCJC, Blanc et ASTRID) pour leur soutien financier.
Références
- Romby P, Marzi S, Westhof E. La structure atomique du ribosome en pleine lumière. Med Sci (Paris) 2009 ; 25 : 977–981. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Gillet R, Felden B. Lost in translation : le déblocage des ribosomes bactériens par le mécanisme de trans-traduction. Med Sci (Paris) 2007 ; 23 : 633–639. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Schmeing TM, Ramakrishnan V. What recent ribosome structures have revealed about the mechanism of translation. Nature 2009 ; 461 : 1234–1242. [CrossRef] [PubMed] [Google Scholar]
- Zaher HS, Green R. Quality control by the ribosome following peptide bond formation. Nature 2009 ; 457 : 161–166. [CrossRef] [PubMed] [Google Scholar]
- Wilson DN. Less is more for leaderless mRNA translation. Mol Cell 2009 ; 33 : 141–142. [CrossRef] [PubMed] [Google Scholar]
- Kane JF. Effects of rare codon clusters on high-level expression of heterologous proteins in Escherichia coli. Curr Opin Biotechnol 1995 ; 6 : 494–500. [CrossRef] [PubMed] [Google Scholar]
- Neubauer C, Gillet R, Kelley AC, Ramakrishnan V. Decoding in the absence of a codon by tmRNA and SmpB in the ribosome. Science 2012 ; 335 : 1366–1369. [CrossRef] [PubMed] [Google Scholar]
- Weis F, Bron P, Giudice E, et al. tmRNA-SmpB: a journey to the centre of the bacterial ribosome. EMBO J 2010 ; 29 : 3810–3818. [CrossRef] [PubMed] [Google Scholar]
- Weis F, Bron P, Rolland JP, et al. Accommodation of tmRNA-SmpB into stalled ribosomes: a cryo-EM study. RNA 2010 ; 16 : 299–306. [CrossRef] [PubMed] [Google Scholar]
- Giudice E, Mace K, Gillet R. Trans-translation exposed: understanding the structures and functions of tmRNA-SmpB. Front Microbiol 2014 ; 5 : 113. [CrossRef] [PubMed] [Google Scholar]
- Ito K, Chadani Y, Nakamori K, et al. Nascentome analysis uncovers futile protein synthesis in Escherichia coli. PLoS One 2011 ; 6 : e28413. [CrossRef] [PubMed] [Google Scholar]
- Moore SD, Sauer RT. Ribosome rescue: tmRNA tagging activity and capacity in Escherichia coli. Mol Microbiol 2005 ; 58 : 456–466. [CrossRef] [PubMed] [Google Scholar]
- Graille M, Seraphin B. Surveillance pathways rescuing eukaryotic ribosomes lost in translation. Nat Rev Mol Cell Biol 2012 ; 13 : 727–735. [CrossRef] [PubMed] [Google Scholar]
- Inada T. Quality control systems for aberrant mRNAs induced by aberrant translation elongation and termination. Biochim Biophys Acta 2013 ; 1829 : 634–642. [CrossRef] [PubMed] [Google Scholar]
- Shimizu Y. ArfA recruits RF2 into stalled ribosomes. J Mol Biol 2012 ; 423 : 624–631. [CrossRef] [PubMed] [Google Scholar]
- Schaub RE, Poole SJ, Garza-Sanchez F, et al. Proteobacterial ArfA peptides are synthesized from non-stop messenger RNAs. J Biol Chem 2012 ; 287 : 29765–29775. [CrossRef] [PubMed] [Google Scholar]
- Hayes CS, Keiler KC. Beyond ribosome rescue: tmRNA and co-translational processes. FEBS Lett 2010 ; 584 : 413–419. [CrossRef] [PubMed] [Google Scholar]
- Handa Y, Inaho N, Nameki N. YaeJ is a novel ribosome-associated protein in Escherichia coli that can hydrolyze peptidyl-tRNA on stalled ribosomes. Nucleic Acids Res 2011 ; 39 : 1739–1748. [CrossRef] [PubMed] [Google Scholar]
- Gagnon MG, Seetharaman SV, Bulkley D, Steitz TA. Structural basis for the rescue of stalled ribosomes: structure of YaeJ bound to the ribosome. Science 2012 ; 335 : 1370–1372. [CrossRef] [PubMed] [Google Scholar]
- Keiler KC, Feaga HA. Resolving nonstop translation complexes is a matter of life or death. J Bacteriol 2014 ; 196 : 2123–2130. [CrossRef] [PubMed] [Google Scholar]
- Pech M, Karim Z, Yamamoto H, et al. Elongation factor 4 (EF4/LepA) accelerates protein synthesis at increased Mg2+ concentrations. Proc Natl Acad Sci USA 2011 ; 108 : 3199–3203. [CrossRef] [Google Scholar]
- Doerfel LK, Wohlgemuth I, Kothe C, et al. EF-P is essential for rapid synthesis of proteins containing consecutive proline residues. Science 2013 ; 339 : 85–88. [CrossRef] [PubMed] [Google Scholar]
- Ude S, Lassak J, Starosta AL, et al. Translation elongation factor EF-P alleviates ribosome stalling at polyproline stretches. Science 2013 ; 339 : 82–85. [CrossRef] [PubMed] [Google Scholar]
- Vivanco-Dominguez S, Bueno-Martinez J, Leon-Avila G, et al. Protein synthesis factors (RF1, RF2, RF3, RRF, and tmRNA) and peptidyl-tRNA hydrolase rescue stalled ribosomes at sense codons. J Mol Biol 2012 ; 417 : 425–439. [CrossRef] [PubMed] [Google Scholar]
- Ferbitz L, Maier T, Patzelt H, et al. Trigger factor in complex with the ribosome forms a molecular cradle for nascent proteins. Nature 2004 ; 431 : 590–596. [CrossRef] [PubMed] [Google Scholar]
- Wendrich TM, Blaha G, Wilson DN, et al. Dissection of the mechanism for the stringent factor RelA. Mol Cell 2002 ; 10 : 779–788. [CrossRef] [PubMed] [Google Scholar]
- Gerdes K, Christensen SK, Lobner-Olesen A. Prokaryotic toxin-antitoxin stress response loci. Nat Rev Microbiol 2005 ; 3 : 371–382. [CrossRef] [PubMed] [Google Scholar]
- Christensen SK, Pedersen K, Hansen FG, Gerdes K. Toxin-antitoxin loci as stress-response-elements: ChpAK/MazF and ChpBK cleave translated RNAs and are counteracted by tmRNA. J Mol Biol 2003 ; 332 : 809–819. [CrossRef] [PubMed] [Google Scholar]
- Pedersen K, Zavialov AV, Pavlov MY, et al. The bacterial toxin RelE displays codon-specific cleavage of mRNAs in the ribosomal A site. Cell 2003 ; 112 : 131–140. [CrossRef] [PubMed] [Google Scholar]
- Giudice E, Gillet R. The task force that rescues stalled ribosomes in bacteria. Trends Biochem Sci 2013 ; 38 : 403–411. [CrossRef] [PubMed] [Google Scholar]
- Ramadoss NS, Zhou X, Keiler KC. tmRNA is essential in Shigella flexneri. PLoS One 2013 ; 8 : e57537. [CrossRef] [PubMed] [Google Scholar]
- Huang C, Wolfgang MC, Withey J, et al. Charged tmRNA but not tmRNA-mediated proteolysis is essential for Neisseria gonorrhoeae viability. EMBO J 2000 ; 19 : 1098–1107. [CrossRef] [PubMed] [Google Scholar]
- Keiler KC, Alumasa JN. The potential of trans-translation inhibitors as antibiotics. Future Microbiol 2013 ; 8 : 1235–1237. [CrossRef] [PubMed] [Google Scholar]
- De la Cruz J, Vioque A. Increased sensitivity to protein synthesis inhibitors in cells lacking tmRNA. RNA 2001 ; 7 : 1708–1716. [PubMed] [Google Scholar]
- Vioque A, De la Cruz J. Trans-translation and protein synthesis inhibitors. FEMS Microbiol Lett 2003 ; 218 : 9–14. [CrossRef] [PubMed] [Google Scholar]
- Andini N, Nash KA. Expression of tmRNA in mycobacteria is increased by antimicrobial agents that target the ribosome. FEMS Microbiol Lett 2011 ; 322 : 172–179. [CrossRef] [PubMed] [Google Scholar]
- Cole ST. Microbiology. Pyrazinamide-old TB drug finds new target. Science 2011 ; 333 : 1583–1584. [CrossRef] [PubMed] [Google Scholar]
- Shi W, Zhang X, Jiang X, et al. Pyrazinamide inhibits trans-translation in Mycobacterium tuberculosis. Science 2011 ; 333 : 1630–1632. [CrossRef] [PubMed] [Google Scholar]
- Ramadoss NS, Alumasa JN, Cheng L, et al. Small molecule inhibitors of trans-translation have broad-spectrum antibiotic activity. Proc Natl Acad Sci USA 2013 ; 110 : 10282–10287. [CrossRef] [Google Scholar]
- Okan NA, Mena P, Benach JL, et al. The smpB-ssrA mutant of Yersinia pestis functions as a live attenuated vaccine to protect mice against pulmonary plague infection. Infect Immun 2010 ; 78 : 1284–1293. [CrossRef] [PubMed] [Google Scholar]
- Zhang J, van Aartsen JJ, Jiang X, et al. Expansion of the known Klebsiella pneumoniae species gene pool by characterization of novel alien DNA islands integrated into tmRNA gene sites. J Microbiol Methods 2011 ; 84 : 283–289. [CrossRef] [PubMed] [Google Scholar]
- Wernecke M, Mullen C, Sharma V, et al. Evaluation of a novel real-time PCR test based on the ssrA gene for the identification of group B streptococci in vaginal swabs. BMC Infect Dis 2009 ; 9 : 148. [CrossRef] [PubMed] [Google Scholar]
- O’Grady J, Sedano-Balbas S, Maher M, et al. Rapid real-time PCR detection of Listeria monocytogenes in enriched food samples based on the ssrA gene, a novel diagnostic target. Food Microbiol 2008; 25 : 75–84. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Présentation sommaire de la traduction canonique bactérienne. Démarrage : après appariement entre l’ARNm et le 30S via des séquences complémentaires d’ARN (interactions Shine-Dalgarno/anti-Shine-Dalgarno), le codon d’initiation AUG (voire GUG ou UUG) est positionné dans le site P. Un complexe initiateur, constitué de l’ARNt-fMet, d’IF2 et de GTP, s’y fixe, en présence des deux autres facteurs d’initiation, IF1 et IF3. La grande sous-unité ribosomique 50S se lie alors au complexe, provoquant l’hydrolyse du GTP et le départ des facteurs d’initiation. élongation : le facteur d’élongation EF-Tu amène les ARNt aminoacylés (aa-ARNt) au site A du ribosome sous la forme d’un complexe ternaire aa-ARNt-EF-Tu·GTP. S’il y a correspondance entre le codon de l’ARNm et l’anticodon de l’ARNt, EF-Tu hydrolyse le GTP et se dissocie, tandis que l’ARNt pénètre profondément dans le site A (étape d’accommodation). Le peptide naissant est transféré à l’ARNtaa accommodé dans le site A, entraînant l’allongement du peptide d’un acide aminé. Par un mouvement de cliquet spontané (ratchet) entre ses deux sous-unités, le ribosome oscille alors entre deux conformations. Le facteur d’élongation EFG- GTP se lie alors au ribosome, et l’hydrolyse du GTP permet la translocation des ARNt du site A au site P, et du site P au site E. S’ensuit le départ d’EF-G-GDP. Le processus est itératif, ce qui provoque l’allongement séquentiel du peptide naissant, tandis que les ARNt se déplacent ° travers les sites ribosomiques A, P et E. Terminaison : lorsqu’un codon de terminaison (ou codon stop UAA, UAG ou UGA) atteint le site A, un facteur de libération de classe I (RF1 ou RF2) se lie et catalyse la séparation entre la chaîne polypeptidique et l’ARNt en place dans le site P. La protéine néosynthétisée est libérée du ribosome, après prise en charge par des machines de repliement co-traductionnel (MRC). Un complexe binaire composé de RF3, un facteur de libération de classe II, et d’une molécule de GTP, facilite la dissociation de RF1 ou RF2 et de l’ARNt déacylé. Recyclage : le facteur de recyclage ribosomique (RRF) associé à EF-G recycle le ribosome en séparant les deux sous-unités, libérant les ARNt et l’ARNm. Un nouveau cycle de traduction peut alors démarrer. aa-ARNt : ARN de transfert aminoacylé ; ARNm : ARN messager ; fMet-ARNt : ARN de transfert N-formylméthionine ; EF-G : facteur d’élongation G ; GDP : guanosine diphosphate ; GTP : guanosine-5’-triphosphate ; EF-Tu : facteur d’élongation thermosensible ; IF : facteur d’initiation ; MRC : machine de repliement co-traductionnel ; RF : facteur de libération ; RRF : facteur de recyclage ribosomique ; SD : séquence de Shine-Dalgarno ; 30S : petite sous-unité ribosomique ; 50S : grande sous-unité ribosomique. |
| Dans le texte | |
 |
Figure 2. Illustration des différentes situations conduisant à la production de complexe non-stop en haut et de complexe no-go en bas. Les sous-unités 30S et 50S sont en jaune et bleu clair, respectivement. L’ARNm est en bleu foncé (les codons rares en marron et le codon stop en rouge). Le peptide naissant est en violet. L’ARNt du site P est en vert, du site E en orange, les ARNt déacylés en rouge et les ARNt suppresseurs en rose pâle. Les ions Mg2+ sont symbolisés par des points rouges et les antibiotiques par des triangles. Les endo- et exonucléases sont respectivement représentées par une paire de ciseaux et un symbole pac-man ; le stress oxydant par un éclair jaune. |
| Dans le texte | |
 |
Figure 3. Comparaison des structures tridimensionnelles du complexe ARNtm-SmPB et d’un ARNt. A. Complexe ARNtm-SmPB. SmPB est en violet et les domaines de l’ARNtm sont représentés dans différentes couleurs : les deux domaines principaux, le TLD en bleu et le MLD en gris, allant jusqu’au codon stop représenté en jaune. H : hélices ; PK : pseudo-nœuds. B. Un ARNt canonique afin de comparer la similitude structurale entre le complexe ARNtm-SmPB et un ARNt. Les deux extrémités 3’-CCA sont liées à un acide aminé (pour l’ARNtm, c’est toujours une alanine). |
| Dans le texte | |
 |
Figure 4. Schéma des différentes étapes de la trans-traduction. (1) Ribosome bloqué lors de la traduction d’un ARNm défectueux (bleu) avec un ARNt dans le site E (orange) et un ARNt dans le site P (vert) lié à la protéine naissance (violet). Le site A est vide. (2) Le complexe ARNtm-SmPB-EF-Tu reconnaît le ribosome bloqué. (3) Aidé par EF-G (bleu ciel), le complexe est transloqué dans le ribosome et l’ARNm tronqué est éjecté pour être dégradé par la RNase R. (4) Le peptide incomplet est transféré sur l’ARNtm et la traduction redémarre sur sa partie codante (MLD). (5) Il en résulte l’addition d’une étiquette peptidique à la protéine incomplète. L’ARNtm est translucide car les détails structuraux de cette étape ne sont pas encore connus. (6) Le ribosome bloqué est recyclé grâce à la présence d’un codon de terminaison sur la séquence codante de l’ARNtm, tandis que le peptide étiqueté est dégradé par des protéases. |
| Dans le texte | |
 |
Figure 5. Illustration des différents systèmes de sauvetage contre le blocage des ribosomes bactériens. Pendant la traduction, le ribosome peut se gripper de deux façons différentes. On parle de no-go lorsqu’il ralentit ou s’arrête avant que le codon d’arrêt ne soit atteint, ou de non-stop lorsqu’il atteint l’extrémité 3’ d’un ARNm. Les complexes ribosomiques no-go peuvent retourner à la traduction canonique grâce à EF-P, qui aide la lecture des séquences riches en codons proline, ou grâce à EF4, qui remobilise les ribosomes lorsque la concentration intracellulaire en Mg2+ est élevée. Ces mêmes complexes peuvent être recyclés par la PTH qui sépare le peptide naissant lié à l’ARNt lorsque le ribosome est dissocié. Enfin, ces complexes peuvent être convertis en non-stop lorsque l’ARNm est clivé dans le site A. Les complexes non-stop peuvent être sauvés par trans-traduction, par ArfA, en coopération avec RF2, ou par ArfB. Entrées PDB 3HUW pour EF-P ; 3DEG pour EF4 ; 3VJR pour PTH ; 3KIQ pour RelE ; 2WH1 pour RF2 ; 4DH9 pour ArfB ; et 3IYQ pour ARNtm-SmPB. La structure ArfA a été modélisée avec I-TASSER (iterative threading assembly refinement). Son positionnement exact dans le ribosome est encore inconnu. Les éléments des systèmes de sauvetage sont en rouge. Les autres codes couleurs sont les mêmes que ceux employés dans la Figure 4 . |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




