| Issue |
Med Sci (Paris)
Volume 25, Number 5, Mai 2009
Évaluation des risques et perspectives thérapeutiques en oncologie colorectale
|
|
|---|---|---|
| Page(s) | 524 - 528 | |
| Section | Forum | |
| DOI | https://doi.org/10.1051/medsci/2009255524 | |
| Published online | 15 May 2009 | |
Compétition iGEM 2008
Deux équipes françaises !
iGEM competition 2008, two French teams!
Médecine/Sciences et École Normale Supérieure, Paris, France
Les ingrédients nécessaires et suffisants pour provoquer le foisonnement d’idées ingénieuses dans un domaine interdisciplinaire et révolutionnaire en plein essor, la « biologie synthétique », semblent remarquablement simples à trouver : il suffit de réunir une quinzaine d’étudiants prédoctorants très motivés de formations diverses, d’ajouter une poignée d’encadrants prêts à investir à la fois leurs connaissances et leur sens psychologique, de mettre entre leurs mains une collection de bio-bricks1 et de faire mijoter le tout pendant quatre mois à température d’été, dans un environnement propice doté de préférence de paillasses et d’ordinateurs.
L’imagination au pouvoir
Bienvenue dans le monde des iGEM (international genetically engineered machine competition) qui existe depuis 2003 ; à l’origine, cette compétition consistait simplement à réunir quelques étudiants pour un mois d’activité au Massachusetts Institute of Technology (MIT) dans le but de faire « clignoter » des cellules [1, 2]. Deux ans plus tard, en 2005, 13 équipes s’affrontent au niveau international et en 2008, elles sont 84 équipes réunissant plus de 1 000 participants venus défendre leurs projets en novembre au MIT, projets tous plus ambitieux les uns que les autres. On leur confie la mission suivante : réaliser des systèmes complexes fondés sur la biologie et remplissant des fonctions n’existant pas dans la nature en combinant le contenu d’une boîte à outils de composants dits « biobriques » pouvant s’assembler en machines biologiques selon le modèle des composants standardisés des circuits électroniques. Il suffit de jeter un coup d’œil sur la liste des résumés des projets2 pour être convaincu de l’ingéniosité de ces multiples idées : des circuits génétiques induisant la différenciation de cellules souches en cardiomyocytes pour réparer le tissu cardiaque après un infarctus (Bay Area, Regenerative Sciences Institute), des bactéries capables de dégrader les déchets en plastique et d’autres produisant des polymères pour un « bioplastique » moins polluant (Duke University), des E. coli qui convertissent la cellulose en amidon et bêta-carotène (Edimbourg, Grande-Bretagne), un organisme minimisant son propre génome (Eidgenössische Technische Hochschule, Zurich) et même des E. coli sensibles au toucher (TokyoTech) ou capables de chanter (National Autonomous University of Mexico)…
Évidemment il faut faire une place particulière à l’équipe victorieuse de Slovénie qui a réussi à développer un vaccin contre les cancers gastriques et les ulcères provoqués par Helicobacter pylori en assemblant les « briques immunologiques » nécessaires pour permettre la détection du pathogène par le système immunitaire inné et pour déclencher la synergie avec la formation d’anticorps spécifiques. Hormis l’application médicale pratique parfaitement réalisable, les étudiants ont également pris en compte le facteur sociologique en mettant l’accent sur le fait qu’il s’agit d’une méthode peu coûteuse applicable dans les pays en développement.
L’équipe parisienne
Le soir du 18 décembre se réunit au CRI (Centre de recherches interdisciplinaires) pour la dernière fois l’équipe parisienne des iGEM 2008 autour d’un buffet et d’une coupe de champagne (Figure 1). Mais l’ambiance est loin d’être celle d’un adieu - les échanges de souvenirs vifs se mélangent aux projets pour l’année suivante.
 |
Figure 1. L’équipe parisienne des iGEM 2008. Étudiants : Alexandra Bouziz, Pharmacochimie, Master SPGF, Université Paris 7 Diderot ; Philippe Bouziz, Physico-chimie, M2 Approches Interdisciplinaires du Vivant, Université Paris 5 Descartes ; Guillaume Bouchard, Lycée Jean-Perrin de Lyon ; Fanny Caffin, Biologie moléculaire et cellulaire, Master en cancérologie, Université Paris 11 Orsay ; Benoit D’Hayer, Pharmacie et Génétique, master Européen de Génétique, Université Paris Descartes ; Audrey Desgrange, Biologie, ICES ; Ana Jimenez, Biologie, master Européen de Génétique, Université Paris 7 Diderot ; Cyprien Maisonnier, Biologie, ENS ; Kok-Phen Yan, Biochimie, M2 Biologie Moléculaire et Génétique, Université Paris 6 Pierre et Marie Curie ; Felipe Golib, Mathématiques, Master Approches Interdisciplinaires du Vivant, Université Paris 5 Descartes ; Louis Hedde, Ingénieur, École des Mines ; Yann Le Cunff, Mathématiques appliquées, Supelec ; Hugo Raguet, Ingénieur, Ecole Centrale ; Romain Rousseau, Ingénieur Biotechnologie, ParisTech. Encadrants : Grégory Batt, INRIA Roquencourt ; David Bikard, Institut Pasteur ; Samuel Bottani, Université Paris 7 Denis Diderot ; Ariel Lindner, Université Paris-Descartes, Inserm (coordinateur). Conseillers : Aurélien Rizk, INRIA ; Gilles Vieira ; Genoscope Évry. Aide scientifique : Franck Delaplace, Université d’Évry ; Olivier Michel, Université de Créteil ; Jean Louis Giavitto, Université d’Évry ; Richard Emmanuel Eastes, ENS ; Anne Le Goff, Responsable de la collaboration iGEM/Science Académie. |
Tout avait commencé au sein du club de biologie synthétique et systémique (club SynBioSys) créé par le CRI et hébergé par la faculté de médecine de l’Université Paris Descartes qui avait décidé de lancer une large campagne de publicité pour la compétition iGEM dans les grandes écoles et les universités, mettant également à disposition des étudiants un laboratoire du CRI. Le soutien financier provenait d’un ensemble de mécènes parmi lesquels des fondations, des universités de Paris, des sociétés de recherche et des firmes privées.
L’originalité de l’équipe parisienne est sa diversité car, contrairement à la majorité des autres équipes participantes, ses membres ne sont pas issus d’une seule université mais viennent d’horizons très divers, grandes écoles d’ingénieurs, École normale supérieure et universités parisiennes ; les compétences sont variées : biologistes de diverses spécialités, de la cancérologie à la génétique, informaticiens, mathématiciens, pharmacologues et même un lycéen venu exprès de Lyon après avoir eu vent du projet grâce à la Science Ac (académie). La seule consigne imposée par l’iGEM était que les participants devaient être des prédoctorants, mais aucune autre sélection ne s’est exercée sur les participants, et leur nombre n’était pas limité.
BactériO’Clock : le projet iGEM parisien
Le choix du sujet s’est fait quasiment à l’unanimité après discussion d’un ensemble de propositions faites par les différents membres de l’équipe : il s’inspirait d’une idée développée dans l’ouvrage du chercheur Uri Alon An Introduction to Systems Biology3 qui avait été étudié par le club de biologie synthétique.
La question centrale du projet nommé BacteriO’Clock est celle du contrôle temporel de l’expression génétique. Le but était d’établir un ordre logique d’expression de gènes en s’inspirant de circuits électroniques appelés FIFO (First In First Out) qui reposent sur le principe que l’ordre d’activation de différents processus correspond à leur ordre d’extinction. En termes d’expression génétique, le premier gène activé sera le premier à être éteint.
L’intérêt d’un tel système est de fabriquer un outil pour des chaînes de biosynthèse à l’échelle cellulaire : la coordination des étapes successives garantit l’optimisation de l’usage des ressources qui sont alors produites au bon moment et uniquement pendant la durée nécessaire.
Dans ce contexte, l’équipe s’est inspirée du circuit de contrôle génétique servant à la mise en place du système flagellaire de E. coli, et elle a développé un système constitué de trois gènes qui doivent être contrôlés par deux gènes régulateurs (Figure 2). L’ensemble repose sur l’ajustement précis des « forces » des interactions génétiques qui dépendent des différentes affinités de liaison des produits régulateurs avec leurs séquences cibles, liant ainsi l’allumage et l’extinction successive des gènes à la courbe de la quantité de régulateur présent.
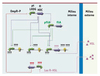 |
Figure 2. Le système de contrôle temporel de l’expression génétique « BacteriO’clock » développé par l’équipe. Le but de la construction est de faire en sorte que les trois gènes contrôlés, représentés par les petites ampoules, s’allument ET s’éteignent successivement dans l’ordre 1, 2, 3 selon d’un principe de FIFO (first in, first out). Pour aboutir à ce fonctionnement séquentiel, les trois gènes sont tout d’abord placés sous le contrôle du gène régulateur A, dont le produit possède une affinité de liaison - et donc une force d’activation décroissante - pour les trois gènes : lorsque l’expression de A est déclenchée, celle du gène 1 est la première à être activée et il suffit d’une faible concentration de produit A en raison de la forte affinité ; le gène 3 s’allume en dernier en présence d’une forte concentration de A. Or, si le système en restait là, lors de l’extinction de A, l’ordre de désactivation se ferait alors dans l’ordre inverse 3, 2, 1. C’est là qu’entre en jeu le gène régulateur B, lui-même activé par A, d’où un certain décalage de l’accumulation du produit B par rapport à celui de A. Les affinités de liaison de B pour les trois gènes contrôlés sont, à l’inverse de A, croissantes dans l’ordre 1, 2, 3. En raison du décalage de la (dés)activation de B par rapport à A, l’influence de B joue surtout au moment de l’extinction du gène A en forçant les gènes à s’éteindre dans l’ordre 1, 2, 3. La désactivation de A est due à un rétrocontrôle négatif par le gène 3. Le concept de ce dernier composant a évolué au cours du projet ; son but est de faire osciller le système. À la place d’une inhibition directe de A par le gène 3 se substitue un mécanisme de quorum sensing : le gène 3 activé produit du HSL qui est sécrété dans le milieu externe et qui est simultanément reconnu à la surface des cellules et internalisé. Lorsqu’il est en quantité suffisante, le HSL endocyté active un gène inhibiteur de A et désactive l’expression de ce dernier jusqu’à ce que la dégradation progressive du HSL permette au système de redémarrer. Outre son rôle dans la boucle de rétrocontrôle, le passage par le milieu extérieur synchronise les oscillations de toute une population bactérienne transformée. |
Afin d’enrichir le système d’un comportement oscillant, un rétrocontrôle négatif via un mécanisme de quorum sensing à base de production de HSL (homosérine lactone) est ajouté.
Au cours des quatre mois de travail, l’équipe a non seulement réussi à fabriquer la quasi-totalité des plasmides requis et à tester une partie du circuit sur des bactéries, mais elle a aussi fourni un travail considérable sous la forme de nombreuses modélisations mathématiques et informatiques. Celles-ci servent à la fois à l’estimation de paramètres inconnus à partir de données expérimentales et à la prédiction du comportement du système en action. En fait, les premiers modèles ne vérifiaient pas le comportement oscillant anticipé en raison d’un phénomène d’amortissement, ce qui demandait un ajustement des paramètres, voire une modification du circuit - démarche qui aurait exigé des semaines de travail de paillasse, et n’a pas pu être réalisé à temps.
L’équipe strasbourgeoise
Quelle meilleure preuve d’internationalité que la composition de l’équipe strasbourgeoise ? Les sept étudiants (Figure 3) étaient en majorité suisses et allemands en dernière année de maîtrise de l’École supérieure de biotechnologie de Strasbourg (ESBS) ; l’ESBS est un partenariat entre les universités de Fribourg et de Karlsruhe en Allemagne, l’université de Bâle en Suisse et l’université de Strasbourg en France ; les étudiants suivent des cours dans les trois pays et sont quasiment trilingues en fin de cursus.
 |
Figure 3. L’équipe de l’ESBS Strasbourg. Étudiants : Martin Dressler, Manuel Gersbacher, Marius Müller, Sandra Dittmann, Michael Wild, Katja Karstens, Robin Sorg, Paul Bourgine. Encadrants : Mariel Donzeau, Jacques Haiech (absents de la photo). |
Fait peut-être unique parmi toutes les équipes participantes, c’est à la seule initiative des étudiants qu’a été prise la décision de participer aux iGEM 2008. Une conférence donnée par Randy Rettberg, directeur des iGEM, à l’Institut de génomique et de biologie moléculaire et cellulaire à côté de l’ESBS en septembre les encouragea à former une équipe et ils demandèrent le soutien de l’ESBS. Un soutien qui dépasse vite le cadre des iGEM quand l’ESBS décide de créer une nouvelle option de biologie synthétique au sein de son programme, sous la direction de Jacques Haiech, encadrant de l’équipe ; l’option inclut en « bonus » une participation facultative aux iGEM. Autre fruit de l’engagement des étudiants, une coopération plus que fructueuse établie avec une école de physique, initialement dans le but de pouvoir bénéficier des programmes de modélisation nécessaires au travail, mais qui a abouti, cerise sur le gâteau, à la soumission d’un article traitant de la comparaison de divers logiciels de modélisation. Quant au financement, la présentation du projet de recherche - et de ses retombées potentielles, notamment en cancérologie - aux représentants d’institutions de recherche et pharmacologiques de la région du Haut-Rhin a suffi pour s’assurer maints soutiens financiers.
Un compteur binaire de divisions cellulaires - le projet strasbourgeois
Fruit d’un brainstorming intense ayant débuté en janvier, le projet sélectionné par l’équipe est indépendant des sujets des laboratoires de l’ESBS et ne s’inspire d’aucun ouvrage connu.
L’idée est de développer des constructions génétiques permettant de compter les divisions cellulaires qui soient plus élégantes et efficaces que celles qui sont utilisées couramment. Il s’agit de fabriquer, dans la levure Saccharomyces cerevisiae, un interrupteur bidirectionnel couplé au cycle cellulaire (Figure 4). Au cœur du système se trouvent deux constructions similaires, dont l’une code pour un signal visible du type GFP (green fluorescent protein). À tout moment, seulement l’une des deux est active et inhibe l’expression de l’autre. L’élément répresseur des constructions comporte une séquence de dégradation par l’APC (anaphase promoting complex). Or l’APC, une ubiquitine-ligase, est active uniquement pendant l’anaphase et la phase G1 du cycle cellulaire, et va éteindre le répresseur de la construction active, ce qui permet de switcher sur l’expression de l’autre construction. Cette alternance d’activité des deux constructions (et donc d’expression de la GFP) permet de compter les divisions.
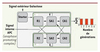 |
Figure 4. Un interrupteur génétique bidirectionnel régulé par le cycle cellulaire : le projet de l’équipe de Strasbourg. Les deux constructions A et B comportent chacune un auto-activateur (SA), un répresseur (R) ainsi qu’un activateur (CA) de l’autre gène. CA et R sont exprimés en parallèle, mais le répresseur l’emporte sur l’activateur. Les auto-activateurs et les répresseurs comportent un motif de dégradation par l’APC. Un signal extérieur (starter) active la construction A, maintenue ensuite par une boucle de rétrocontrôle positif via SA1. L’expression du répresseur R1 inhibe celle de la construction B et l’emporte sur l’activité de CA1. À la fin de la mitose, l’APC est exprimée et induit la dégradation de R1 et de SA1, permettant alors à CA1 d’activer la construction B tandis que A s’éteint. La disparition de l’APC en G1 permet l’expression de SA2, qui prend le relais pour maintenir l’activation de B, et de R2, qui réprime A. Lors de l’anaphase suivante, le système s’inverse de nouveau. En insérant le gène codant pour la GFP dans la construction A, les cellules exprimeront la fluorescence une génération sur deux, permettant de compter les divisions cellulaires. |
Les applications directes du système décrit seraient une détermination facile, voire automatisable, de l’âge d’une cellule ou d’une population de cellules, la détection d’une croissance anormale comme on l’observe dans le cas des cellules tumorales et finalement - tout à fait dans l’esprit des machines génétiques - la fabrication d’un programme nécessitant l’alternance répétée de l’expression de deux gènes lors d’un processus de synthèse. En d’autres termes : la création d’une nouvelle « brique biologique ».
La phase expérimentale avait été précédée d’une intense période de modélisation sélectionnant les composants génétiques les plus à même de fonctionner selon le modèle proposé. C’est ainsi par exemple que l’APC a été sélectionnée : son rôle bien défini dans le cycle cellulaire en fait la protéine endogène la mieux adaptée pour jouer le rôle d’interface avec les constructions introduites.
Même si le système final n’a pas pu être assemblé par manque de temps, l’équipe strasbourgeoise a enrichi le registre de composants des iGEM d’un total de 27 nouvelles constructions (biobricks) et défini une base théorique exhaustive qui ne demande qu’à être mise en pratique.
iGEM, une expérience de l’interdisciplinarité
Selon les Parisiens, le plus grand bénéfice de l’expérience iGEM est sûrement cet échange interdisciplinaire puisque tous les participants ont contribué aussi bien au travail expérimental qu’à la modélisation. « La biologie était pour moi un domaine peu connu, et c’était un défi pour moi de réussir à trouver ma place en tant qu’ingénieur et pas uniquement comme mathématicien ! » affirme Louis Hedde (École des Mines). Maintenant, les notions de plasmides, de GFP et de séquences d’activation sont familières à tous ; quant aux biologistes purs et durs, ils ont découvert l’univers de la modélisation grâce aux explications détaillées et patientes des étudiants ingénieurs, mathématiciens et informaticiens.
Mais l’expérience est également extrêmement intéressante d’un point de vue psychologique et se prêterait presque à une étude de comportement du groupe.
Samuel Bottani, l’un des encadrants, enseignant à l’université Paris Diderot, souligne le niveau exceptionnel de l’ensemble des participants : « Il est intéressant de remarquer qu’il n’y a pas eu la formation d’une dynamique du type leader/follower que l’on observe habituellement au sein d’un groupe, et même pendant les heures précédant la présentation, des différences d’opinion persistaient ».
Pourtant, les participants ont réussi à s’organiser, à planifier les différentes étapes du projet et à s’investir jusqu’à son terme avec une intervention minimale des encadrants. La réussite de l’équipe en l’absence totale d’obligations, d’horaires fixes, ou d’emploi du temps prédéfini contredit tout théorème disant que seul un guidage strict mène au succès. L’investissement était un choix personnel, poussé volontairement à l’extrême par certains ayant quasiment passé jour et nuit au CRI. D’autres ont « rapidement renoncé au stage “classique” de fin de L3 prévu » pour participer aux iGEM, comme le confirment Cyprien Maisonnier (École Normale Supérieure) et Hugo Raguet (École Centrale) et la plupart des participants ont surtout renoncé à leurs vacances, comme les étudiants de l’équipe de Strasbourg, qui avaient institué un système de rotation pour que le travail au laboratoire ne soit pas interrompu. Mais l’aspect d’organisation a été ressenti de façon très positive par l’ensemble des étudiants : tous reconnaissent que la prise de responsabilité dans un projet commun et l’organisation de manière autonome sans repères extérieurs - un défi majeur de cette compétition - ont représenté les principales leçons tirées de cette expérience.
L’autonomie se reflète également dans l’avancement du projet et des expériences qui repose sur le principe trial and error. « Nous les laissions volontairement faire des erreurs » explique Ariel Lindner, encadrant et coordinateur de l’équipe parisienne. « C’était de loin plus utile que de leur dire d’avance que la manipulation allait échouer. »
L’organisation du projet représente pour les sept étudiants de l’ESBS une expérience importante - ayant commencé très tôt à travailler sur le projet, ils ont quasiment passé une année entière restant très soudés dans les bons moments comme dans les plus difficiles. On lit cette phrase dans le rapport final des Strasbourgeois : « Souvent nous avions des avis très différents et quelques tensions se sont produites. Mais voici ce que nous avons appris : comment accepter la position de chacun et comment se motiver les uns les autres ». Les interactions avec divers spécialistes et laboratoires ont été perçues comme très fructueuses, sans parler des contacts durables qui se sont développés entre chercheurs et étudiants. L’apogée de l’expérience personnelle était bien évidemment la finale au MIT les 8 et 9 novembre 2008 - « un véritable régal » selon Jacques Haiech, encadrant des Strasbourgeois - permettant la rencontre avec les autres équipes venues du monde entier dans une ambiance chaleureuse et marquée par un véritable fairness, suivi d’un détour personnel de l’équipe parisienne à New York.
L’ère « post-iGEM »
Depuis, les cours, les stages ou le travail ont repris, mais les iGEM sont loin d’être terminés pour l’équipe BacteriO’Clock et les Strasbourgeois, car ils font désormais partie des « anciens » dont le devoir sera d’encadrer et de soutenir les équipes 2009. À Paris, l’idée d’une équipe commune avec l’université de Pékin (Chine) flotte dans l’air pendant que l’ESBS est en train de mettre en place un forum européen de la biologie synthétique et des iGEM afin de favoriser davantage la dimension internationale… Pour la suite, attendons Novembre !
À l’année prochaine !

Bio-bricks : en début d’été, chaque équipe participant aux iGEM reçoit un ensemble de standards de constructions biologiques du répertoire des composants biologiques standards (Registry of standard biological parts), comportant divers plasmides, des constructions de gènes rapporteurs variés, des enzymes de restriction et bien plus. Toute nouvelle construction développée par une équipe doit être mise à la disposition du répertoire, dans lequel elle sera éventuellement intégrée.
© 2009 médecine/sciences - Inserm / SRMS
Liste des figures
 |
Figure 1. L’équipe parisienne des iGEM 2008. Étudiants : Alexandra Bouziz, Pharmacochimie, Master SPGF, Université Paris 7 Diderot ; Philippe Bouziz, Physico-chimie, M2 Approches Interdisciplinaires du Vivant, Université Paris 5 Descartes ; Guillaume Bouchard, Lycée Jean-Perrin de Lyon ; Fanny Caffin, Biologie moléculaire et cellulaire, Master en cancérologie, Université Paris 11 Orsay ; Benoit D’Hayer, Pharmacie et Génétique, master Européen de Génétique, Université Paris Descartes ; Audrey Desgrange, Biologie, ICES ; Ana Jimenez, Biologie, master Européen de Génétique, Université Paris 7 Diderot ; Cyprien Maisonnier, Biologie, ENS ; Kok-Phen Yan, Biochimie, M2 Biologie Moléculaire et Génétique, Université Paris 6 Pierre et Marie Curie ; Felipe Golib, Mathématiques, Master Approches Interdisciplinaires du Vivant, Université Paris 5 Descartes ; Louis Hedde, Ingénieur, École des Mines ; Yann Le Cunff, Mathématiques appliquées, Supelec ; Hugo Raguet, Ingénieur, Ecole Centrale ; Romain Rousseau, Ingénieur Biotechnologie, ParisTech. Encadrants : Grégory Batt, INRIA Roquencourt ; David Bikard, Institut Pasteur ; Samuel Bottani, Université Paris 7 Denis Diderot ; Ariel Lindner, Université Paris-Descartes, Inserm (coordinateur). Conseillers : Aurélien Rizk, INRIA ; Gilles Vieira ; Genoscope Évry. Aide scientifique : Franck Delaplace, Université d’Évry ; Olivier Michel, Université de Créteil ; Jean Louis Giavitto, Université d’Évry ; Richard Emmanuel Eastes, ENS ; Anne Le Goff, Responsable de la collaboration iGEM/Science Académie. |
| Dans le texte | |
 |
Figure 2. Le système de contrôle temporel de l’expression génétique « BacteriO’clock » développé par l’équipe. Le but de la construction est de faire en sorte que les trois gènes contrôlés, représentés par les petites ampoules, s’allument ET s’éteignent successivement dans l’ordre 1, 2, 3 selon d’un principe de FIFO (first in, first out). Pour aboutir à ce fonctionnement séquentiel, les trois gènes sont tout d’abord placés sous le contrôle du gène régulateur A, dont le produit possède une affinité de liaison - et donc une force d’activation décroissante - pour les trois gènes : lorsque l’expression de A est déclenchée, celle du gène 1 est la première à être activée et il suffit d’une faible concentration de produit A en raison de la forte affinité ; le gène 3 s’allume en dernier en présence d’une forte concentration de A. Or, si le système en restait là, lors de l’extinction de A, l’ordre de désactivation se ferait alors dans l’ordre inverse 3, 2, 1. C’est là qu’entre en jeu le gène régulateur B, lui-même activé par A, d’où un certain décalage de l’accumulation du produit B par rapport à celui de A. Les affinités de liaison de B pour les trois gènes contrôlés sont, à l’inverse de A, croissantes dans l’ordre 1, 2, 3. En raison du décalage de la (dés)activation de B par rapport à A, l’influence de B joue surtout au moment de l’extinction du gène A en forçant les gènes à s’éteindre dans l’ordre 1, 2, 3. La désactivation de A est due à un rétrocontrôle négatif par le gène 3. Le concept de ce dernier composant a évolué au cours du projet ; son but est de faire osciller le système. À la place d’une inhibition directe de A par le gène 3 se substitue un mécanisme de quorum sensing : le gène 3 activé produit du HSL qui est sécrété dans le milieu externe et qui est simultanément reconnu à la surface des cellules et internalisé. Lorsqu’il est en quantité suffisante, le HSL endocyté active un gène inhibiteur de A et désactive l’expression de ce dernier jusqu’à ce que la dégradation progressive du HSL permette au système de redémarrer. Outre son rôle dans la boucle de rétrocontrôle, le passage par le milieu extérieur synchronise les oscillations de toute une population bactérienne transformée. |
| Dans le texte | |
 |
Figure 3. L’équipe de l’ESBS Strasbourg. Étudiants : Martin Dressler, Manuel Gersbacher, Marius Müller, Sandra Dittmann, Michael Wild, Katja Karstens, Robin Sorg, Paul Bourgine. Encadrants : Mariel Donzeau, Jacques Haiech (absents de la photo). |
| Dans le texte | |
 |
Figure 4. Un interrupteur génétique bidirectionnel régulé par le cycle cellulaire : le projet de l’équipe de Strasbourg. Les deux constructions A et B comportent chacune un auto-activateur (SA), un répresseur (R) ainsi qu’un activateur (CA) de l’autre gène. CA et R sont exprimés en parallèle, mais le répresseur l’emporte sur l’activateur. Les auto-activateurs et les répresseurs comportent un motif de dégradation par l’APC. Un signal extérieur (starter) active la construction A, maintenue ensuite par une boucle de rétrocontrôle positif via SA1. L’expression du répresseur R1 inhibe celle de la construction B et l’emporte sur l’activité de CA1. À la fin de la mitose, l’APC est exprimée et induit la dégradation de R1 et de SA1, permettant alors à CA1 d’activer la construction B tandis que A s’éteint. La disparition de l’APC en G1 permet l’expression de SA2, qui prend le relais pour maintenir l’activation de B, et de R2, qui réprime A. Lors de l’anaphase suivante, le système s’inverse de nouveau. En insérant le gène codant pour la GFP dans la construction A, les cellules exprimeront la fluorescence une génération sur deux, permettant de compter les divisions cellulaires. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




