| Numéro |
Med Sci (Paris)
Volume 30, Numéro 5, Mai 2014
|
|
|---|---|---|
| Page(s) | 558 - 566 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20143005020 | |
| Publié en ligne | 13 juin 2014 | |
Analyse in vivo de la signalisation tumorale induite par les tyrosine-kinases par protéomique quantitative
Analysis of oncogenic signaling induced by tyrosine kinases in tumors by SILAC-based quantitative proteomic approach
1
CNRS UMR5237, université de Montpellier 1 et 2, centre de recherche de biochimie macromoléculaire (CRBM), 34000
Montpellier, France
2
CNRS UMR5203, Inserm U661, université de Montpellier 1 et 2, institut de génomique fonctionnelle (IGF), plate-forme de protéomique fonctionnelle, 34000
Montpellier, France
Les tyrosine-kinases (TK) régulent la signalisation intracellulaire induite par de nombreux stimulus extracellulaires et conduisent à la croissance ou à l’adhésion cellulaires. La dérégulation de leur activité leur confère des propriétés oncogéniques qui contribuent à la formation de cancers chez l’homme. Cependant, les voies de signalisation impliquées ne sont que partiellement élucidées. Dans ce contexte, l’analyse par spectrométrie de masse de type SILAC (stable isotope labelling with amino acids in cell culture) permet de caractériser l’ensemble des substrats et la dynamique de phosphorylation activée par ces TK. Cette approche a ainsi mis en évidence une complexité inattendue de la signalisation oncogénique induite par la TK Src dans les cellules de cancer colorectal (CCR). Dans cette revue, nous décrivons une nouvelle méthode de type SILAC appliquée à des modèles in vivo de tumeurs humaines xénogreffées chez la souris immunodéprimée. L’application de cette méthode aux tumeurs colorectales a révélé des différences importantes dans la signalisation dépendante de Src in vivo et in vitro. Enfin, nous discutons l’intérêt du SILAC par rapport à d’autres méthodes de protéomique in vivo, ainsi que dans ses applications en cancérologie.
Abstract
Protein tyrosine kinases (TK) transmit intracellular signaling induced by many extracellular stimuli resulting in cell growth or adhesion. Deregulation of their activity leads to malignant cell transformation that plays an important role in human cancer. The signaling pathways involved in this oncogenic process are however only partially elucidated. Interestingly, SILAC-based quantitative proteomics allow the identification of the whole spectrum of TK substrates and the dynamic of phosphorylation state involved in oncogenic signaling. For example, this approach has highlighted the unsuspected complexity of the oncogenic signaling induced by the TK Src in colorectal cancer (CRC) cells. In this review, we describe a new SILAC-based technology applied to in vivo models of human tumors engrafted in nude mice. This method revealed significant differences between Src-oncogenic signaling of CRC cells in tumors and in culture. Finally, we discuss the interest of SILAC with recently described in vivo proteomic methods and in cancer, including the analysis of oncogenic signaling in tumor progression and the anti-tumoral activity of TK inhibitors in vivo.
© 2014 médecine/sciences – Inserm

Les tyrosine-kinases
La cellule normale reçoit et répond à des signaux extracellulaires provenant de son microenvironnement qui contrôlent sa croissance, sa morphologie, son métabolisme et sa motilité. L’exécution correcte de ces différents processus biologiques est assurée par l’activation de signaux chimiques intracellulaires complexes qui aboutissent à la réponse cellulaire adéquate. La phosphorylation des protéines joue un rôle crucial dans ces voies de transduction des signaux intracellulaires. Chez les métazoaires, la phosphorylation des protéines est effectuée pour plus de 99 % sur des résidus sérine (Ser) et thréonine (Thr), et pour seulement 0,8 % sur des résidus tyrosine (Tyr). Bien que très minoritaire, la phosphorylation sur Tyr (pTyr) constitue un mécanisme majeur dans la régulation de ces réponses cellulaires. Ce mécanisme a émergé avec l’apparition des organismes multicellulaires, sans doute pour coordonner la communication cellulaire au sein d’un organisme complexe [1]. Bien que très sophistiquée, la signalisation pTyr résulte de la combinaison de trois mécanismes moléculaires élémentaires que sont : (1) la phosphorylation du substrat, permettant l’activation du signal, (2) l’interaction avec une protéine effectrice contenant un domaine d’interaction protéine-protéine SH2, assurant la lecture du signal, et (3) la déphosphorylation du substrat, conduisant à l’arrêt du signal [2, 31]. Ce mécanisme de signalisation est extrêmement régulé afin d’éviter toute réponse cellulaire inappropriée, ce qui explique le taux cellulaire basal si faible de pTyr. En effet, la dérégulation d’une de ces trois actions conduit à de nombreuses pathologies humaines. Par exemple, les TK sont fréquemment dérégulées dans les cancers où elles jouent un rôle important dans la progression tumorale [3]. Ainsi, les TK sont devenues des cibles pharmacologiques attractives en thérapie anticancéreuse, et certains inhibiteurs de ces enzymes constituent des médicaments très efficaces [4, 5]. Citons, par exemple, le Gleevec® et le Tasigna® qui ciblent l’oncoprotéine BCR (breakpoint cluster region)-ABL, et qui sont maintenant utilisés en première ligne dans le traitement des patients atteints de leucémie myéloïde chronique [6].
La tyrosine-kinase Src
On dénombre 90 tyrosine-kinases chez l’homme, dont 58 récepteurs (RTK) et 32 TK cytoplasmiques. Les RTK sont, pour la plupart, des récepteurs de facteurs de croissance, ce qui confirme le rôle essentiel de la signalisation par pTyr dans la régulation de la croissance cellulaire. Cependant, la signalisation pTyr est également utilisée par des récepteurs dénués de séquence catalytique, comme les intégrines ou les récepteurs des cytokines, des hormones et des neurotransmetteurs. Dans ce contexte, ces récepteurs activent la TK Src, qui joue un rôle central dans ce type de réponse. Historiquement, Src a été la première TK identifiée grâce à la découverte du premier oncogène, le gène transformant v-Src du rétrovirus aviaire RSV (Figure 1) [7]. Cette découverte majeure démontrait ainsi le rôle essentiel des TK dans la tumorigenèse. Quelques années plus tard, son homologue cellulaire c-Src a été découvert dans les cellules non transformées, révélant l’existence des proto-oncogènes et un mécanisme fondamental de la tumorigenèse. Src se localise à la face interne de la membrane plasmique des cellules grâce à un site de myristylation présent dans sa séquence amino-terminale. Cette TK se compose d’un domaine kinase qui est responsable de son activité enzymatique, ainsi que des domaines non catalytiques d’interaction protéiques SH2 et SH3 qui lui permettent, à la fois, d’activer et de relayer le signal induit par pTyr. Cette double fonction, ainsi que sa capacité à phosphoryler un grand nombre de substrats (voir plus loin), expliquent le rôle central de cette TK dans les réponses cellulaires (Figure 2A). Par exemple, la liaison du facteur de croissance PDGF (plateled-derived growth factor) à son récepteur, le PDGFR, induit sa phosphorylation sur des résidus Tyr localisés dans sa partie intracellulaire, qui crée, alors, un site de liaison avec le domaine SH2 de Src [8]. La formation de ce complexe conduit à l’activation enzymatique de Src, la phosphorylation de substrats spécifiques et la transmission du signal. Par exemple, la phosphorylation de STAT3 (signal transducer and activator of transcription 3) par Src conduit à l’augmentation de l’expression du facteur de transcription Myc, un régulateur clé de l’induction de la synthèse d’ADN (Figure 2A).
 |
Figure 1. Principales découvertes scientifiques autour de la tyrosine kinase Src. |
 |
Figure 2. Signalisation cellulaire normale et tumorale régulée par la tyrosine kinase Src. Signalisations régulées par Src en réponse à l’activation des récepteurs à activité tyrosine kinase (RTK) et des intégrines dans les cellules normales A. Signalisations induites par la dérégulation de Src dans les cellules tumorales B. AKT : protéine kinase B ; FAK : focal adhesion kinase ; MAPK : mitogen activated protein kinase ; PI3K : phosphoinositide 3-kinase. |
Signalisation tumorale induite par la tyrosine-kinase Src
Compte tenu du rôle essentiel de la signalisation de pTyr dans le contrôle de la prolifération ou de la motilité cellulaires, il n’est pas surprenant que sa dérégulation joue un rôle important dans l’initiation et/ou la progression tumorales. En effet, une signalisation dépendante de pTyr aberrante conduit à la transformation cellulaire, qui se traduit par une augmentation de la prolifération cellulaire en l’absence de facteurs de croissance, une capacité accrue de la survie cellulaire ou encore l’induction de l’invasion et de la dissémination cellulaires nécessaire à la formation des métastases. La dérégulation de cette voie de signalisation est essentiellement attribuée à l’activation aberrante de TK (activation du signal). En effet, plus de 60 % des TK sont dérégulées dans les cancers avec des propriétés oncogéniques importantes [3]. Cependant, la dérégulation de cette voie peut être aussi induite par une activation anormale des substrats ou par un défaut de contrôle de l’arrêt du signal par l’inactivation des tyrosine-phosphatases. Bien que peu documentée, la dérégulation de ces étapes joue un rôle tout aussi important dans l’activation d’un signal oncogénique dépendant de pTyr.
La découverte de l’activité oncogénique de v-Src dans le sarcome du poulet a permis d’apporter des éléments importants dans la compréhension des mécanismes par lesquels les TK induisent la tumorigenèse. Ces travaux ont permis de conclure que l’activité oncogénique des TK est directement associée à la dérégulation de leur activité enzymatique. Ainsi, v-Src résulte de mutations activatrices qui lui confèrent une activité enzymatique totalement dérégulée. Cette activité aberrante conduit à la stimulation permanente des voies de signalisation cellulaire normalement induites par les facteurs de croissance. Suite à ces résultats, de nombreux laboratoires ont recherché un rôle semblable de Src dans les cancers chez l’homme. Toutefois, contrairement à v-Src ou à d’autres oncogènes cellulaires, tels que Ras, Src est très rarement muté. L’absence de mutation somatique a suggéré l’idée selon laquelle Src ne joue pas de rôle important dans les cancers chez l’homme. Malgré cela, Src est hyperactivée dans de nombreuses tumeurs humaines, en particulier celles d’origine épithéliale (Figure 1) [9]. Par exemple, Src est dérégulée dans la majorité des tumeurs colorectales, et son activité est 5 à 10 fois supérieure à celle qui est observée dans le tissu sain autologue. Les mécanismes moléculaires responsables de son activation sont encore mal connus. La dérégulation de Src est souvent associée à la surexpression de la protéine qui est due, en partie, à l’amplification du gène correspondant. Toutefois, cette surexpression ne peut être à elle seule responsable de sa dérégulation, car son activité enzymatique est extrêmement régulée. En particulier, Src est inhibée par la TK CSK (C-Src kinase), dont la phosphorylation stabilise l’enzyme en conformation inactive. CSK définit ainsi un mécanisme essentiel d’arrêt du signal activé par Src. Dans les cellules de cancers colorectaux, ce mécanisme est fortement atténué conduisant ainsi à une forte dérégulation de Src [10].
Le rôle clé de Src dans le cancer colorectal est soutenu par des données clinicobiologiques qui montrent que son niveau de dérégulation constitue un facteur de mauvais pronostic pour les patients. Depuis, plusieurs résultats expérimentaux définissent des rôles spécifiques de Src dans la tumorigenèse colorectale, dont l’augmentation de la prolifération et de la survie cellulaires, ainsi qu’une activation importante du processus d’angiogenèse (Figure 2B) [11]. Src joue également un rôle clé dans les étapes tardives de la progression tumorale, en particulier lors de l’invasion et de la dissémination des cellules tumorales conduisant à la formation de métastases1. Malgré ces données importantes, la fonction ainsi que la nature des voies de signalisation associées aux différentes étapes de la progression tumorale ne sont que partiellement élucidées. Dans ce contexte, l’identification et la quantification des signaux d’activation (substrats) sont essentielles pour mieux comprendre la fonction oncogénique de Src dans la progression tumorale.
Analyse phosphoprotéomique de la signalisation tumorale induite par les tyrosine-kinases in vitro
La phosphoprotéomique quantitative in vitro
L’analyse de la dynamique des modifications post-traductionnelles est un enjeu crucial pour une compréhension exhaustive des mécanismes de signalisation mis en jeu au cours d’une réponse cellulaire. Dans le cas des TK, une telle analyse est compliquée par la très faible abondance et la nature transitoire du signal pTyr. Le développement d’anticorps spécifiques des résidus pTyr s’est avéré être un outil très précieux dans le domaine, et il a permis de purifier les premiers substrats dont la phosphorylation était abondante et stable. Par exemple, ces anticorps ont permis d’identifier les premiers substrats de Src contrôlant l’adhésion à la matrice extracellulaire, tels que la cortactine, révélant ainsi un rôle important de cette enzyme dans ce processus cellulaire (Figure 1) [12]. Cependant, ces approches ne permettent pas d’identifier ni de quantifier les sites de phosphorylation, en particulier ceux dont la phosphorylation est peu abondante, mais souvent biologiquement importante. Dans ce contexte, le développement de la spectrométrie de masse (SM) a révolutionné les méthodes d’analyses dans ce domaine. En particulier, les approches de phosphoprotéomique quantitative permettent d’identifier et de donner une quantification relative de chaque site de phosphorylation dans un échantillon biologique donné [13]. Ces méthodes passent par une première étape biochimique d’enrichissement en protéines/peptides phosphorylés, utilisant différentes méthodes de chromatographie d’affinité. Pour les résidus pTyr, l’enrichissement est fait par immunoaffinité en utilisant des combinaisons d’anticorps spécifiques des résidus pTyr
La méthode SILAC
Actuellement, on distingue deux grands types de méthodes de quantification en protéomique : celles sans marquage, basées sur l’intensité du signal ou sur le nombre de spectres associés à un peptide, et celles basées sur le marquage isotopique différentiel, chimique (marquage de type iTRAQ [isobaric Tag for relative and absolute quantitation]) ou métabolique (marquage de type SILAC [stable isotope labelling with amino acids in cell culture]). La méthode SILAC a été décrite en 2002 par le laboratoire du Dr Mann [14]. Elle constitue, par la simplicité de sa mise en œuvre et sa précision d’analyse, une méthode de tout premier choix pour la réalisation d’analyses quantitatives robustes et reproductibles [15]. Son principe est basé sur le marquage métabolique du protéome cellulaire par incorporation d’acides aminés isotopiquement alourdis (non radioactifs) contenus dans le milieu de culture (Figure 3A). Les acides aminés classiquement utilisés sont la lysine et l’arginine. Ces acides aminés essentiel et semi-essentiel assurent un marquage constant de tous les peptides obtenus après digestion trypsique des protéines, puisque la trypsine clive spécifiquement les chaînes peptidiques après une lysine ou une arginine. Généralement, les protéines cellulaires ont incorporé les acides aminés lourds avec un taux ≥ 90 % après six à huit doublements cellulaires. Les protéines extraites des deux populations cellulaires à comparer sont ensuite mélangées en quantités égales pour être traitées dans des conditions équivalentes, limitant ainsi l’introduction de biais lors de la quantification. Lors de l’analyse en SM, le différentiel de masse connu entre les acides aminés légers et lourds permet de distinguer les peptides issus des deux échantillons (8-Da pour la lysine et 10-Da pour l’arginine dans des conditions de marquage classiques) et le rapport de l’intensité des deux signaux permet de mesurer l’abondance relative de chaque peptide. Malgré sa simplicité, une étape clé de cette approche est l’efficacité du marquage du protéome dans les conditions d’analyse (ici la culture cellulaire).
 |
Figure 3. Comparaison des méthodes SILAC in vitro et in vivo. La méthode SILAC sur cellules en culture ( A) : le marquage est effectué en cultivant les cellules dans un milieu contenant, soit des acides aminés naturels (condition « légère », en bleu), soit de la lysine et de l’arginine C13/N15 (condition « lourde », en rouge). La méthode SILAC sur tumeurs xénogreffées ( B) : après injection par voie sous-cutanée des cellules, les souris sont nourries, soit avec des granulés de nourriture standard (condition « légère », en bleu), soit avec de la nourriture contenant de la lysine C13 (condition « lourde », en rouge). Après marquage des cellules ou des tumeurs, les protéines totales sont extraites, mélangées en quantité égale (ratio 1:1), puis soumises à une immunopurification en utilisant des anticorps reconnaissant les résidus pTyr. Un fractionnement sur gel 1D est effectué afin d’augmenter la profondeur d’analyse. Les bandes sont ensuite découpées et les protéines sont digérées en utilisant de la trypsine pour le SILAC in vitro (A) ou de la Lys-C pour le SILAC in vivo (B). Les peptides obtenus sont analysés après séparation par chromatographie liquide en utilisant un spectromètre de masse de type LTQ-Orbitrap. Les données sont ensuite interprétées grâce au logiciel MaxQuant [29]. Le rapport d’intensité des signaux des peptides marqués et non marqués permet de calculer une abondance relative. Pour une protéine, le ratio sera déduit de l’ensemble des ratios obtenus pour chacun de ces peptides. |
Analyse SILAC de la signalisation tumorale régulée par Src in vitro
Bien que récentes, les approches de phosphoprotéomique quantitative de type SILAC ont permis la caractérisation détaillée de la signalisation oncogénique induite par de nombreuses TK, dont l’oncoprotéine cytoplasmique BCR-ABL et le récepteur à l’EGF (EGFR, epidermal growth factor receptor) [16, 17]. Ainsi, plusieurs laboratoires, dont le nôtre, ont utilisé cette méthode pour analyser la signalisation oncogénique dépendante de Src [18, 19]. Comme décrit plus haut, l’expression de Src augmente les propriétés transformantes des cellules tumorales de cancer colorectal qui métastasent, et cette réponse est associée à une forte augmentation du signal pTyr. Pour caractériser cette signalisation, nous avons analysé le phosphoprotéome associé à Src par la méthode SILAC. Cette analyse révèle que plus de 30 % des protéines phosphorylées sur Tyr présentent une augmentation de phosphorylation suite à l’expression de Src, suggérant ainsi un large spectre de substrats de cette kinase. De manière surprenante, une proportion importante des substrats identifiés correspondent à des substrats inconnus ou sans lien direct avec le cancer, soulignant notre connaissance encore limitée du rôle de Src dans la signalisation oncogénique. Enfin, cette analyse a permis de montrer que Src phosphoryle un réseau de substrats TK nécessaires à la transmission du signal oncogénique, ce qui suggère que certaines protéines identifiées par SILAC correspondent à des substrats indirects de Src [18]. En particulier, Src phosphoryle les RTK Met (hepatocyte growth factor receptor) et EphA2 (ephrin type-A receptor 2), qui jouent un rôle essentiel dans l’induction des propriétés transformantes des cellules tumorales (Figure 2B). Ces résultats mettent en évidence la complexité de la signalisation oncogénique induite par pTyr et pose la question du rôle des RTK dans la signalisation émanant d’une TK cytoplasmique. Ainsi, il est probable qu’une des fonctions oncogéniques de Src soit de modifier la réponse tumorale aux facteurs de leur micro-environnement via la phosphorylation de récepteurs spécifiques.
Analyse phosphoprotéomique de la signalisation tumorale induite par les tyrosine-kinases in vivo
La méthode SILAC appliquée aux tumeurs expérimentales
Si les modèles de culture de cellules tumorales ont permis de découvrir certains mécanismes fondamentaux de l’oncogenèse, ils ne permettent cependant pas d’appréhender toute la complexité environnementale des cellules tumorales au sein d’une tumeur. En particulier, les modèles de culture cellulaire ne permettent pas de résumer la géométrie, ni les contraintes mécaniques de la cellule tumorale au sein d’une tumeur, les conditions d’hypoxie, de nutriments ou la présence des cytokines inflammatoires sécrétées par le microenvironnement, ni les nombreuses interactions hétérotypiques avec les cellules du stroma, du système immunitaire ou du tissu endothélial2 [32, 33]. Dans ce contexte, la méthode SILAC ne semble pas appropriée pour réaliser de telles analyses à cause du marquage métabolique de la tumeur nécessaire à l’analyse protéomique quantitative. Toutefois, de nouvelles méthodes ont permis de contourner ce problème par l’addition d’acides aminés lourds dans l’environnement nutritionnel, permettant ainsi un marquage significatif de plusieurs organismes entiers, dont le poisson zèbre, le nématode, la drosophile, le triton et la souris [15, 20]. Par exemple, l’addition de [13C6]-lysine lourde dans la nourriture des rongeurs permet le marquage protéique complet de tous les organes de souris après deux générations. Ces nouvelles méthodes permettent ainsi l’analyse protéomique quantitative d’animaux entiers, d’organes ou de tissus, et de fluides biologiques spécifiques.
Si les modèles de souris transgéniques sont des outils très intéressants en cancérologie, la xénogreffe de cellules tumorales humaines chez la souris immunodéprimée représente un modèle de référence dans ce domaine. Par exemple, l’expression de Src dans des cellules de cancer colorectal issues de patients, puis leur injection chez les souris immunodéprimées, augmente d’environ trois fois la masse tumorale, démontrant ainsi l’activité oncogénique de Src in vivo [18, 21]. Cette réponse est associée à une augmentation de la prolifération, de l’angiogenèse et du taux de pTyr au sein de la tumeur [21]. Afin de caractériser la signalisation oncogénique impliquée, nous avons développé une nouvelle méthode permettant la quantification du phosphoprotéome associé à Src dans la tumeur xénogreffée. Cette méthode est basée sur le principe du SILAC appliqué à la souris xénogreffée par addition de [13C6]-lysine dans la nourriture (Figure 3B, Encadré 1) [21]. L’étape clé de cette méthode réside en l’obtention d’un marquage isotopique tumoral suffisant (≥ 90 %) pour permettre une analyse quantitative précise et robuste. Bien que le temps de marquage soit limité par le développement optimal de la tumeur, afin d’éviter une nécrose massive induite par une croissance trop importante, un marquage efficace et rapide de la tumeur est attendu en raison du fort taux de prolifération des cellules tumorales (temps de division cellulaire < 20 h). Ainsi, un taux de marquage tumoral de 90 % a pu être obtenu après un mois de ce régime alimentaire contenant la lysine lourde, alors qu’il atteint seulement 66 % dans le tissu musculaire de la souris adjacent à la tumeur [21]. De plus, nous observons une très faible variabilité du marquage entre les différentes souris, ce qui démontre la robustesse de la méthode d’incorporation. Si cette méthode est très semblable à celle du SILAC in vitro, elle n’utilise pas le marquage par l’arginine, du fait de la conversion en proline de cet acide aminé qui est incontrôlable in vivo et qui rend ensuite l’analyse très difficile. Ainsi, le marquage protéique s’effectue uniquement au niveau de la lysine et la digestion peptidique est assurée par l’endoprotéinase Lys-C qui clive uniquement en carboxy-terminal de ces résidus. La taille des peptides générés est un peu plus importante, ce qui peut réduire, dans certains cas, la sensibilité de l’analyse.
1 La méthode SILAC appliquée aux modèles de tumeurs expérimentales chez la souris xénogreffée
L’analyse protéomique SILAC sur les tumeurs xénogreffées chez la souris s’effectue de façon très similaire à une analyse classique de SILAC sur des cellules en culture. Brièvement, les protéines extraites des tumeurs qui sont phosphorylées sur Tyr, ou associées à ces protéines phosphorylées sur Tyr, sont purifiées sur colonne anti-phosphotyrosine. Après séparation sur gel SDS-PAGE, les protéines ainsi purifiées sont digérées avec l’endoprotéinase Lys-C et les peptides obtenus sont analysés par spectrométrie de masse couplée à une nano-chromatographie liquide haute performance (nano pour nano-débit : 300 nl x min-1). Les peptides sont séparés sur une colonne de type phase-inverse selon leur hydrophobicité et directement analysés au niveau du spectromètre de masse. Le spectromètre de masse, de type LTQ OBITRAP XL, fonctionne en alternant deux modes : (1) le mode MS, qui donne la masse des peptides à l’instant t, ainsi que l’intensité du signal pour chacun d’eux ; (2) le mode MS/MS, obtenu par fragmentation des peptides sélectionnés et qui permet d’avoir des informations sur la séquence en acides aminés des peptides par comparaison avec les bases de données. C’est le ratio de l’intensité du signal lourd/léger qui nous permet d’obtenir l’abondance relative des peptides (et donc des protéines) entre les conditions comparées. Les résultats ont été analysés avec le logiciel spécialisé MaxQuant. Cependant, les tumeurs xénogreffées étant un mélange complexe de cellules cancéreuses humaines et de cellules stromales de l’hôte souris (environ 20 % dans les cas des tumeurs colorectales), la base de séquence comporte à la fois les entrées humaines et murines. Pour identifier et quantifier la composante protéique murine et humaine de la tumeur, nous avons développé des outils spécifiques pour pouvoir bien différencier les espèces des protéines analysées [21]. Cette méthode SILAC in vivo a permis d’identifier et de quantifier plus de 1 600 protéines tumorales ainsi purifiées [21].
Analyse SILAC de la signalisation tumorale régulée par Src in vivo
L’analyse phosphoprotéomique quantitative de la croissance tumorale dépendante de Src s’est avérée très reproductible entre les différentes tumeurs et a apporté des renseignements importants sur la nature des voies de signalisation activées in vivo [21]. Tout d’abord, nos résultats montrent que Src présente une efficacité de phosphorylation plus restreinte in vivo, malgré une activité catalytique comparable. La raison de cette différence reste inconnue. Deuxièmement, l’identification de substrats murins suggère que Src envoie des signaux vers les cellules stromales qui composent environ 20 % de la masse tumorale. Enfin, la comparaison de l’analyse SILAC in vitro avec celle in vivo démontre une faible conservation du phosphoprotéome dépendant de Src (Figure 4). Ces résultats révèlent une variabilité de la signalisation activée par Src suivant les conditions d’analyse et mettent en évidence la limitation des modèles de culture cellulaire pour l’analyse de la signalisation tumorale. Malgré cela, on retrouve un nombre significatif de substrats communs, dont les protéines de signalisation et un réseau de substrats TK précédemment discuté. À l’opposé, on identifie un nombre plus restreint de protéines associées à la dynamique du cytosquelette in vivo. Cette différence peut être due aux contraintes biomécaniques plus faibles ou à la redondance de phosphorylation par d’autres TK activées au sein de la tumeur. De plus, on retrouve un nombre important de substrats qui contrôlent le trafic vésiculaire. Par exemple, l’analyse SILAC in vivo a permis la découverte d’une nouvelle fonction tumorale de la protéine TOM1L1 (target of myb1 [chicken]-like 1), qui est impliquée dans le trafic vésiculaire et représente un substrat de Src [21–23]. Enfin, notre analyse in vivo révèle un groupe de 17 substrats liant l’ARN, suggérant un rôle supplémentaire de Src sur la maturation et l’épissage des ARN au cours de la tumorigenèse. En conclusion, l’analyse SILAC appliquée aux modèles des tumeurs expérimentales chez la souris a mis en évidence la diversité des fonctions de Src dans la croissance tumorale et la phosphorylation d’un nombre important de substrats, dont la plupart n’ont pas encore de fonction établie dans la biologie du cancer. Ces résultats suggèrent également un rôle important du trafic vésiculaire et de la maturation des ARNm dans l’activité oncogénique de Src in vivo.
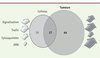 |
Figure 4. Comparaison de la signalisation Src identifiée par SILAC in vitro et in vivo. Ce diagramme de Venn représente le nombre de substrats de Src identifiés in vitro par SILAC sur cellules en culture (en gris clair) et in vivo par SILAC sur tumeurs xénogreffées (en gris foncé). Les substrats identifiés par les deux approches sont indiqués par la superposition des cercles. La taille des flèches est proportionnelle au nombre de substrats identifiés dans chacune des catégories indiquées (protéines de signalisation, de trafic, protéines associées à la dynamique du cytosquelette ou protéines liant l’ARN). |
Comparaison de la méthode SILAC in vivo avec les autres approches de protéomique quantitative
Nous décrivons dans cette revue une nouvelle application de la technologie SILAC développée sur un modèle de tumeurs expérimentales xénogreffées chez la souris. Celle-ci est basée sur un marquage rapide des protéines tumorales par addition de [13C6]-lysine dans la nourriture. Il existe déjà plusieurs méthodes de protéomique quantitative applicables à l’analyse de la signalisation tumorale in vivo qui présentent des propriétés complémentaires (Figure 5). Tout d’abord, l’analyse super-SILAC consiste en la comparaison d’un protéome tumoral à une référence isotopique « lourde » [24]. Cette référence est composée d’une combinaison de protéomes issus de lignées cellulaires tumorales cultivées en milieu lourd. La quantification précise ne sera possible que si le substrat de la tumeur est également présent dans l’échantillon de référence. Cette méthode ne prend pas en compte les substrats régulant l’environnement tumoral, à l’inverse du SILAC in vivo. Toutes les autres méthodes (avec ou sans marquage) impliquent un mélange plus tardif des échantillons à comparer, ce qui peut induire un biais dans la quantification. La méthode de marquage chimique de type iTRAQ permet de comparer jusqu’à huit échantillons en une seule expérience grâce au marquage chimique des peptides par un tag isobarique après la digestion trypsique3 [25]. Ce type de marquage a un inconvénient majeur : la quantification peptidique se fait à partir d’un seul spectre MS/MS, contrairement aux autres techniques où l’analyse est faite sur le signal issu de plusieurs spectres MS. Il existe d’autres techniques de marquage chimique, telles que la diméthylation [26], qui utilise des réactifs peu coûteux (formaldéhyde par exemple). Ces techniques présentent cependant le désavantage (outre le marquage au niveau peptidique) d’amener un marquage qui modifie légèrement le profil d’élution des peptides au niveau chromatographique (analyse en LC [liquid chromatography]-MS/MS) et complique ainsi la quantification. Enfin, la technologie label-free est une méthode quantitative à faible coût prenant de l’ampleur et qui ne nécessite aucun marquage préalable des échantillons. Cependant, elle reste encore à ce jour beaucoup moins précise que les autres méthodes. La méthode SILAC, appliquée à un modèle de tumeurs expérimentales xénogreffées chez la souris, permet une analyse plus complète et précise de la signalisation tumorale induite par les TK. Même si sa mise en œuvre reste coûteuse, elle est limitée par l’obtention d’un marquage isotopique suffisant de la tumeur.
 |
Figure 5. Comparaison des différentes approches de phosphoprotéomique quantitative applicables à l’analyse de tumeurs. Sur ce schéma sont représentés les échantillons à comparer (tumeurs ou cellules) marqués par isotope (en rouge) ou non marqués (en bleu foncé), ainsi que l’étape durant laquelle les échantillons sont mélangés (très précocement pour les marquages métaboliques comme le super-SILAC et le SILAC in vivo, après l’obtention des peptides pour le marquage chimique ou au cours de l’analyse de données pour les conditions sans marquage). L’analyse des données quantitatives est faite à partir des spectres MS1 (intensité du signal peptidique) ou des spectres MS2 (signal des rapporteurs libérés lors de la fragmentation des peptides marqués chimiquement). Cette représentation est adaptée de [30]. |
Conclusion
Les approches de phosphoprotéomique quantitatives in vivo, telles que le SILAC appliqué aux tumeurs expérimentales chez la souris xénogreffée, sont des outils puissants pour l’analyse de la signalisation oncogénique induite par les TK in vivo. En changeant le type d’enrichissement, cette méthode peut également s’appliquer à l’analyse d’autres processus post-translationnels au cours de la tumorigenèse, dont la phosphorylation sur sérine et thréonine, suite à un enrichissement des phosphopeptides sur billes de dioxyde de titane (pour revue voir [27]) ou l’ubiquitination après purification des peptides sur colonne greffée avec des anticorps anti-ubiquitine spécifiques [28]. Enfin, cette méthode devrait avoir de nombreuses applications en cancérologie, en particulier pour l’analyse de la dynamique de la signalisation dépendante de pTyr au cours de la progression tumorale, par exemple au cours du processus métastatique. Elle devrait également avoir un grand intérêt dans l’évaluation d’une molécule thérapeutique ciblant les TK sur la signalisation dépendante de pTyr in vivo et elle permettra de prédire les causes moléculaires de la résistance tumorale innée ou acquise vis-à-vis d’un tel inhibiteur.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Lim WA, Pawson T. Phosphotyrosine signaling: evolving a new cellular communication system. Cell 2010 ; 142 : 661–667. [CrossRef] [PubMed] [Google Scholar]
- Pawson T. Protein modules and signalling networks. Nature 1995 ; 373 : 573–580. [CrossRef] [PubMed] [Google Scholar]
- Blume-Jensen P, Hunter T. Oncogenic kinase signalling. Nature 2001 ; 411 : 355–365. [CrossRef] [PubMed] [Google Scholar]
- Hollande F, Pannequin J, Joubert D. The long road to colorectal cancer therapy: searching for the right signals. Drug Resist Updat 2010 ; 13 : 44–56. [CrossRef] [PubMed] [Google Scholar]
- Krause DS, Van Etten RA. Tyrosine kinases as targets for cancer therapy. N Engl J Med 2005 ; 353 : 172–187. [CrossRef] [PubMed] [Google Scholar]
- Weisberg E, Manley PW, Cowan-Jacob SW, et al. Second generation inhibitors of BCR-ABL for the treatment of imatinib-resistant chronic myeloid leukaemia. Nat Rev Cancer 2007 ; 7 : 345–356. [CrossRef] [PubMed] [Google Scholar]
- Hunter T, Sefton BM. Transforming gene product of Rous sarcoma virus phosphorylates tyrosine. Proc Natl Acad Sci USA 1980 ; 77 : 1311–1315. [CrossRef] [Google Scholar]
- Bromann PA, Korkaya H, Courtneidge SA. The interplay between Src family kinases and receptor tyrosine kinases. Oncogene 2004 ; 23 : 7957–7968. [CrossRef] [PubMed] [Google Scholar]
- Irby RB, Yeatman TJ. Role of Src expression and activation in human cancer. Oncogene 2000 ; 19 : 5636–5642. [CrossRef] [PubMed] [Google Scholar]
- Sirvent A, Benistant C, Pannequin J, et al. Src family tyrosine kinases-driven colon cancer cell invasion is induced by Csk membrane delocalization. Oncogene 2010 ; 29 : 1303–1315. [CrossRef] [PubMed] [Google Scholar]
- Sirvent A, Benistant C, Roche S. Oncogenic signaling by tyrosine kinases of the Src family in advanced colorectal cancer. Am J Cancer Res 2012 ; 2 : 357–371. [PubMed] [Google Scholar]
- Wu H, Parsons JT. Cortactin, an 80/85-kilodalton pp60src substrate, is a filamentous actin-binding protein enriched in the cell cortex. J Cell Biol 1993 ; 120 : 1417–1426. [CrossRef] [PubMed] [Google Scholar]
- Thingholm TE, Jensen ON, Larsen MR. Analytical strategies for phosphoproteomics. Proteomics 2009 ; 9 : 1451–1468. [CrossRef] [PubMed] [Google Scholar]
- Ong SE, Blagoev B, Kratchmarova I, et al. Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. Mol Cell Proteomics 2002 ; 1 : 376–386. [CrossRef] [PubMed] [Google Scholar]
- Emadali A, Gallagher-Gambarelli M. La protéomique quantitative par la méthode SILAC - Technique et perspectives. Med Sci (Paris) 2009 ; 25: 835–842 [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Guo A, Villen J, Kornhauser J, et al. Signaling networks assembled by oncogenic EGFR and c-Met. Proc Natl Acad Sci USA 2008 ; 105 : 692–697. [CrossRef] [Google Scholar]
- Liang X, Hajivandi M, Veach D, et al. Quantification of change in phosphorylation of BCR-ABL kinase and its substrates in response to Imatinib treatment in human chronic myelogenous leukemia cells. Proteomics 2006 ; 6 : 4554–4564. [CrossRef] [PubMed] [Google Scholar]
- Leroy C, Fialin C, Sirvent A, et al. Quantitative phosphoproteomics reveals a cluster of tyrosine kinases that mediates SRC invasive activity in advanced colon carcinoma cells. Cancer Res 2009 ; 69 : 2279–2286. [CrossRef] [PubMed] [Google Scholar]
- Rush J, Moritz A, Lee KA, et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nat Biotechnol 2005 ; 23 : 94–101. [CrossRef] [PubMed] [Google Scholar]
- Kruger M, Moser M, Ussar S, et al. SILAC mouse for quantitative proteomics uncovers kindlin-3 as an essential factor for red blood cell function. Cell 2008 ; 134 : 353–364. [CrossRef] [PubMed] [Google Scholar]
- Sirvent A, Vigy O, Orsetti B, et al. Analysis of SRC oncogenic signaling in colorectal cancer by stable isotope labeling with heavy amino acids in mouse xenografts. Mol Cell Proteomics 2012 ; 11 : 1937–1950. [CrossRef] [PubMed] [Google Scholar]
- Seykora JT, Mei L, Dotto GP, Stein PL. Srcasm: a novel Src activating and signaling molecule. J Biol Chem 2002 ; 277 : 2812–2822. [CrossRef] [PubMed] [Google Scholar]
- Franco M, Furstoss O, Simon V, et al. The adaptor protein Tom1L1 is a negative regulator of Src mitogenic signaling induced by growth factors. Mol Cell Biol 2006 ; 26 : 1932–1947. [CrossRef] [PubMed] [Google Scholar]
- Geiger T, Cox J, Ostasiewicz P, et al. Super-SILAC mix for quantitative proteomics of human tumor tissue. Nat Methods 2010 ; 7 : 383–385. [CrossRef] [PubMed] [Google Scholar]
- Ross PL, Huang YN, Marchese JN, et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Mol Cell Proteomics 2004 ; 3 : 1154–1169. [CrossRef] [PubMed] [Google Scholar]
- Hsu JL, Huang SY, Chow NH, Chen SH. Stable-isotope dimethyl labeling for quantitative proteomics. Anal Chem 2003 ; 75 : 6843–6852. [CrossRef] [PubMed] [Google Scholar]
- Engholm-Keller K, Larsen MR. Technologies and challenges in large-scale phosphoproteomics. Proteomics 2013 ; 13 : 910–931. [CrossRef] [PubMed] [Google Scholar]
- Kim W, Bennett EJ, Huttlin EL, et al. Systematic and quantitative assessment of the ubiquitin-modified proteome. Mol Cell 2011 ; 44 : 325–340. [CrossRef] [PubMed] [Google Scholar]
- Cox J, Mann M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol 2008 ; 26 : 1367–1372. [CrossRef] [PubMed] [Google Scholar]
- Ong SE, Mann M. Mass spectrometry-based proteomics turns quantitative. Nat Chem Biol 2005 ; 1 : 252–262. [CrossRef] [PubMed] [Google Scholar]
- Ashton-Beaucage D, Therrien M. La signalisation RTK/RAS/ERK élargie. Med Sci (Paris) 2010 ; 26 : 1067–1073. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Fridman WH, Sautès-Fridman C. Le microenvironnement tumoral : matrice nourricière, champ de bataille et cible thérapeutique des cancers. Med Sci (Paris) 2014 ; 30 : 359–365. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Provot S. Contrôle de la croissance et de la dissémination tumorales par le microenvironnement : certitudes et hypothèses émergentes. Med Sci (Paris) 2014 ; 30 : 366–371. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Principales découvertes scientifiques autour de la tyrosine kinase Src. |
| Dans le texte | |
 |
Figure 2. Signalisation cellulaire normale et tumorale régulée par la tyrosine kinase Src. Signalisations régulées par Src en réponse à l’activation des récepteurs à activité tyrosine kinase (RTK) et des intégrines dans les cellules normales A. Signalisations induites par la dérégulation de Src dans les cellules tumorales B. AKT : protéine kinase B ; FAK : focal adhesion kinase ; MAPK : mitogen activated protein kinase ; PI3K : phosphoinositide 3-kinase. |
| Dans le texte | |
 |
Figure 3. Comparaison des méthodes SILAC in vitro et in vivo. La méthode SILAC sur cellules en culture ( A) : le marquage est effectué en cultivant les cellules dans un milieu contenant, soit des acides aminés naturels (condition « légère », en bleu), soit de la lysine et de l’arginine C13/N15 (condition « lourde », en rouge). La méthode SILAC sur tumeurs xénogreffées ( B) : après injection par voie sous-cutanée des cellules, les souris sont nourries, soit avec des granulés de nourriture standard (condition « légère », en bleu), soit avec de la nourriture contenant de la lysine C13 (condition « lourde », en rouge). Après marquage des cellules ou des tumeurs, les protéines totales sont extraites, mélangées en quantité égale (ratio 1:1), puis soumises à une immunopurification en utilisant des anticorps reconnaissant les résidus pTyr. Un fractionnement sur gel 1D est effectué afin d’augmenter la profondeur d’analyse. Les bandes sont ensuite découpées et les protéines sont digérées en utilisant de la trypsine pour le SILAC in vitro (A) ou de la Lys-C pour le SILAC in vivo (B). Les peptides obtenus sont analysés après séparation par chromatographie liquide en utilisant un spectromètre de masse de type LTQ-Orbitrap. Les données sont ensuite interprétées grâce au logiciel MaxQuant [29]. Le rapport d’intensité des signaux des peptides marqués et non marqués permet de calculer une abondance relative. Pour une protéine, le ratio sera déduit de l’ensemble des ratios obtenus pour chacun de ces peptides. |
| Dans le texte | |
 |
Figure 4. Comparaison de la signalisation Src identifiée par SILAC in vitro et in vivo. Ce diagramme de Venn représente le nombre de substrats de Src identifiés in vitro par SILAC sur cellules en culture (en gris clair) et in vivo par SILAC sur tumeurs xénogreffées (en gris foncé). Les substrats identifiés par les deux approches sont indiqués par la superposition des cercles. La taille des flèches est proportionnelle au nombre de substrats identifiés dans chacune des catégories indiquées (protéines de signalisation, de trafic, protéines associées à la dynamique du cytosquelette ou protéines liant l’ARN). |
| Dans le texte | |
 |
Figure 5. Comparaison des différentes approches de phosphoprotéomique quantitative applicables à l’analyse de tumeurs. Sur ce schéma sont représentés les échantillons à comparer (tumeurs ou cellules) marqués par isotope (en rouge) ou non marqués (en bleu foncé), ainsi que l’étape durant laquelle les échantillons sont mélangés (très précocement pour les marquages métaboliques comme le super-SILAC et le SILAC in vivo, après l’obtention des peptides pour le marquage chimique ou au cours de l’analyse de données pour les conditions sans marquage). L’analyse des données quantitatives est faite à partir des spectres MS1 (intensité du signal peptidique) ou des spectres MS2 (signal des rapporteurs libérés lors de la fragmentation des peptides marqués chimiquement). Cette représentation est adaptée de [30]. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




