Figure 5.
Télécharger l'image originale
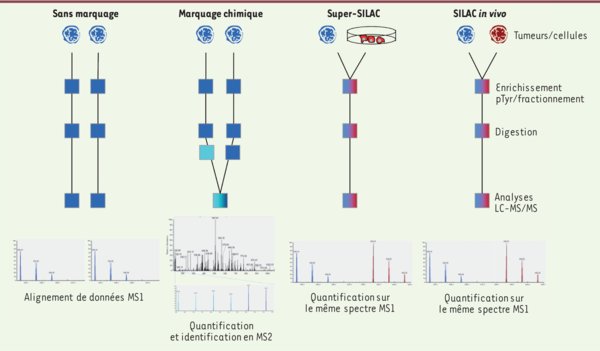
Comparaison des différentes approches de phosphoprotéomique quantitative applicables à l’analyse de tumeurs. Sur ce schéma sont représentés les échantillons à comparer (tumeurs ou cellules) marqués par isotope (en rouge) ou non marqués (en bleu foncé), ainsi que l’étape durant laquelle les échantillons sont mélangés (très précocement pour les marquages métaboliques comme le super-SILAC et le SILAC in vivo, après l’obtention des peptides pour le marquage chimique ou au cours de l’analyse de données pour les conditions sans marquage). L’analyse des données quantitatives est faite à partir des spectres MS1 (intensité du signal peptidique) ou des spectres MS2 (signal des rapporteurs libérés lors de la fragmentation des peptides marqués chimiquement). Cette représentation est adaptée de [30].
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




