Figure 3.
Télécharger l'image originale
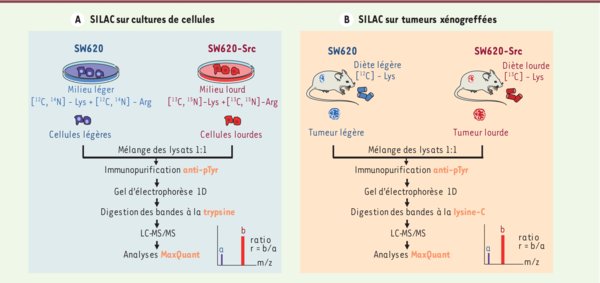
Comparaison des méthodes SILAC in vitro et in vivo. La méthode SILAC sur cellules en culture ( A) : le marquage est effectué en cultivant les cellules dans un milieu contenant, soit des acides aminés naturels (condition « légère », en bleu), soit de la lysine et de l’arginine C13/N15 (condition « lourde », en rouge). La méthode SILAC sur tumeurs xénogreffées ( B) : après injection par voie sous-cutanée des cellules, les souris sont nourries, soit avec des granulés de nourriture standard (condition « légère », en bleu), soit avec de la nourriture contenant de la lysine C13 (condition « lourde », en rouge). Après marquage des cellules ou des tumeurs, les protéines totales sont extraites, mélangées en quantité égale (ratio 1:1), puis soumises à une immunopurification en utilisant des anticorps reconnaissant les résidus pTyr. Un fractionnement sur gel 1D est effectué afin d’augmenter la profondeur d’analyse. Les bandes sont ensuite découpées et les protéines sont digérées en utilisant de la trypsine pour le SILAC in vitro (A) ou de la Lys-C pour le SILAC in vivo (B). Les peptides obtenus sont analysés après séparation par chromatographie liquide en utilisant un spectromètre de masse de type LTQ-Orbitrap. Les données sont ensuite interprétées grâce au logiciel MaxQuant [29]. Le rapport d’intensité des signaux des peptides marqués et non marqués permet de calculer une abondance relative. Pour une protéine, le ratio sera déduit de l’ensemble des ratios obtenus pour chacun de ces peptides.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




