Figure 1.
Télécharger l'image originale
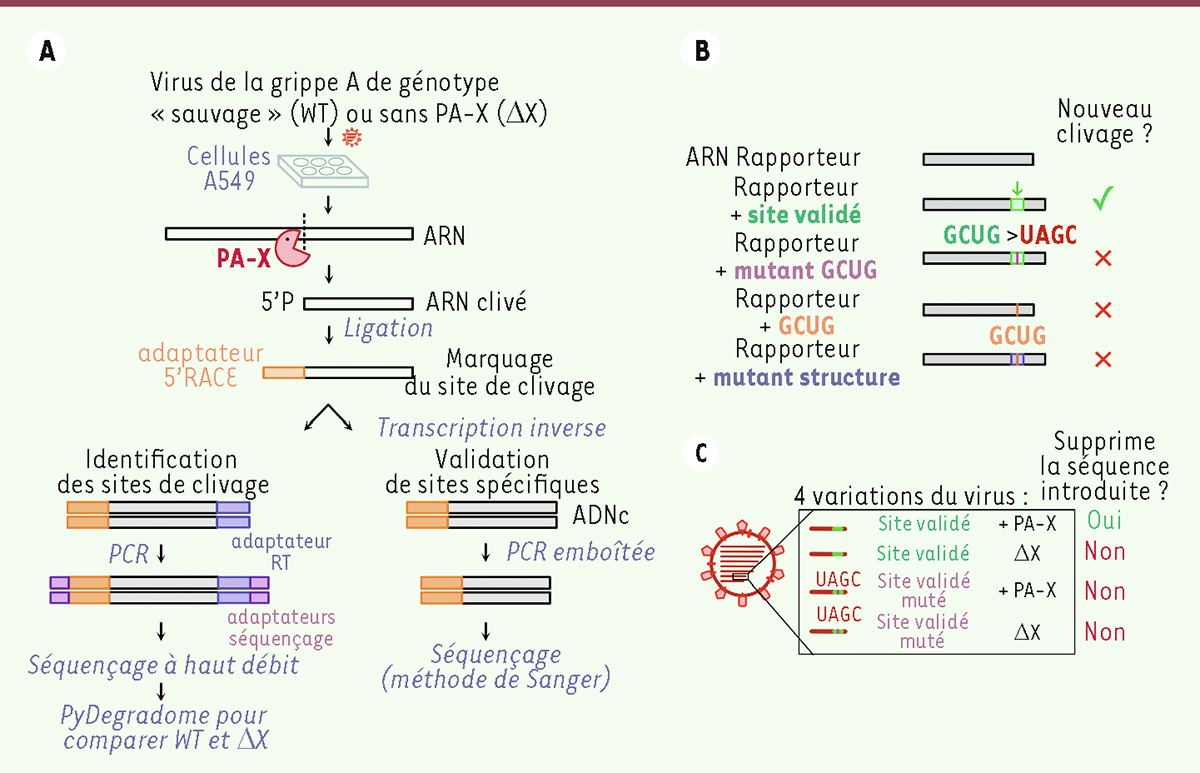
A. Stratégie expérimentale pour identifier, dans le transcriptome cellulaire, les sites de clivage de l’ARN par l’endoribonucléase virale PA-X. Nous avons infecté une lignée cellulaire de cellules épithéliales de poumon humain avec le virus de la grippe A de génotype « sauvage » (wild-type, WT) ou avec un virus muté pour ne plus exprimer la protéine PA-X (∆X), puis purifié l’ARN de chaque échantillon. Suivant le principe de la technique de 5’RACE-PCR, nous avons marqué le site de clivage en liant un adaptateur à l’extrémité 5’ de chaque fragment d’ARN clivé, transformé ces fragments d’ARN en ADNc par transcription inverse (reverse transcription, RT), puis ajouté les adaptateurs de séquençage aux deux extrémités de ces ADNc par PCR. Après l’étape de séquençage nucléotidique à haut débit, nous avons identifié les sites clivés enrichis en présence de PA-X (virus WT versus virus ∆X) grâce au programme PyDegradome. Pour valider les sites clivés identifiés, nous avons suivi le même protocole expérimental, mais en effectuant cette fois deux PCR emboîtées avec des amorces spécifiques du gène étudié. Nous avons ensuite visualisé les fragments amplifiés sur un gel d’agarose, et confirmé leur identité par purification sur gel et séquençage nucléotidique par la méthode de Sanger. B. Différentes versions de l’ARN rapporteur utilisées pour les expériences de clivage de l’ARN par PA-X. L’ajout de la séquence de sites de clivages identifiés fait apparaître un nouveau site de clivage du rapporteur à cet endroit. Ce site n’est plus clivé si le motif GCUG de sa séquence nucléotidique est remplacé par UAGC. De même, il n’est pas clivé si seul le motif GCUG est ajouté et pas le reste de la séquence du site de clivage, ou si cette séquence est mutée pour ne plus former une structure en épingle à cheveux. C. Différentes modifications expérimentales apportées au virus de la grippe A : ajout de la séquence d’un site de clivage validé en présence ou en absence de PA-X, et ajout de cette séquence en remplaçant le motif GCUG central en UAGC en présence ou en absence de PA-X. L’ajout de ces séquences dans le génome du virus n’est toléré qu’en l’absence de PA-X, ou si le motif GCUG central est remplacé par UAGC. Sinon, elles sont rapidement éliminées par mutation.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




