| Issue |
Med Sci (Paris)
Volume 40, Number 2, Février 2024
|
|
|---|---|---|
| Page(s) | 154 - 160 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2023218 | |
| Published online | 27 February 2024 | |
Lsr2 : protéine associée au nucléoïde (NAP) et facteur transcriptionnel chez les mycobactéries
Lsr2: A Nucleoid Associated Protein (NAP) and a transcription factor in mycobacteria
1
Université Paris-Saclay, UVSQ, Inserm, Infection et inflammation, 78180 Montigny-Le-Bretonneux, France
2
APHP, GHU Paris-Saclay,Hôpital Raymond Poincaré, Service de microbiologie, Garches, France
Lsr2, une petite protéine conservée chez les actinobactéries, joue un rôle crucial dans la virulence et l’adaptation des mycobactéries aux conditions environnementales. Membre de la superfamille des protéines associées au nucléoïde (NAP), Lsr2 influence l’organisation de l’ADN en facilitant la formation de boucle chromosomique in vitro, ce qui suggère qu’elle pourrait être un acteur majeur du repliement tridimensionnel du génome. Lsr2 agit également comme un facteur de transcription, régulant l’expression de nombreux gènes responsables de la coordination d’une multitude de processus cellulaires et moléculaires essentiels chez les actinobactéries. Tout comme la protéine H-NS, son orthologue chez les entérobactéries, son rôle de répresseur transcriptionnel repose probablement sur son oligomérisation conduisant à la rigidification de l’ADN et, dans certaines situations, sur le pontage de fragments génomiques distants. Ces mécanismes pourraient perturber le recrutement de l’ARN polymérase sur les promoteurs ainsi que l’élongation des transcrits.
Abstract
Lsr2, a small protein mainly found in actinobacteria, plays a crucial role in the virulence and adaptation of mycobacteria to environmental conditions. As a member of the nucleoid-associated protein (NAPs) superfamily, Lsr2 influences DNA organization by facilitating the formation of chromosomal loops in vitro and, therefore, may be a major player in the three-dimensional folding of the genome. Additionally, Lsr2 also acts as a transcription factor, regulating the expression of numerous genes responsible for coordinating a myriad of cellular and molecular processes essential for the actinobacteria. Similar to the H-NS protein, its ortholog in enterobacteria, its role in transcriptional repression likely relies on oligomerization, rigidifying, and bridging of DNA, thereby disrupting RNA polymerase recruitment as well as the elongation of RNA transcripts.
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.

Vignette (© Frédéric Crémazy).
Lsr2 : une protéine conservée chez les mycobactéries et les actinomycètes
Lsr2 (leprosy serum reactive clone 2) est une petite protéine de 12 à 15 kDa, identifiée pour la première fois en 1991 chez Mycobacterium leprae [1, 2]. Au cours des trois dernières décennies, cette protéine a été caractérisée chez de très nombreuses autres actinobactéries1, y compris les mycobactéries [3-5], les streptomycètes [6], les corynébactéries [7], ainsi que les actinobactériophages2 [8]. Une étude récente a ainsi révélé que, parmi les 2 951 génomes complets d’actinobactériophages analysés, la protéine Lsr2 était présente dans plus de 10 % de ces génomes, ce qui suggère l’implication de ces phages dans l’acquisition du gène lsr2 par leur espèce hôte bactérienne [9]. Le séquençage des génomes des mycobactéries a révélé que Lsr2 est une protéine hautement conservée, avec une identité de séquences d’acides aminés de plus de 85 %, suggérant une conservation importante de sa fonction à travers ce genre [4]. Contrairement aux mycobactéries qui ne possèdent qu’un gène, le génome des streptomycètes codent deux paralogues de lsr2 [6].
Lsr2 : un acteur majeur de la virulence des mycobactéries
Jouant un rôle crucial dans sa virulence ( Figure 1 ), Lsr2 a été identifiée comme un antigène immunodominant chez M. leprae [1]. Plusieurs études ont en effet mis en évidence une réponse lymphocytaire T dirigée contre cette protéine chez les patients souffrant de lèpre, caractérisée par une lymphoprolifération en réponse à de nombreux peptides de Lsr2 ainsi que par la présence d’anticorps spécifiques ciblant ces peptides [1, 10, 11]. Parallèlement, une forte induction et une importante sécrétion d’IFN-γ (interféron-γ) par les lymphocytes T CD3+ CD4+ ont été observées dans des cultures de cellules mononuclées du sang périphérique (PBMC) isolées de patients atteints de lèpre lépromateuse et stimulées par différents peptides de Lsr2 [11].
 |
Figure 1. Lsr2, déterminant physiologique important chez les mycobactéries. Illustration des principaux rôles décrits pour Lsr2 en tant que protéine associée au nucléoïde chez trois groupes de mycobactéries : les mycobactéries pathogènes strictes (Mycobacterium leprae et Mycobacterium tuberculosis) ; les mycobactéries opportunistes (Mycobacterium abscessus) ; et les mycobactéries saprophytes (Mycobacterium smegmatis). IFN-g : interféron gamma. |
Lsr2 joue un rôle majeur dans la morphologie et la composition de la membrane des mycobactéries. L’absence de Lsr2 chez M. smegmatis altère fortement la morphologie et la taille des colonies en milieux solides. Elle inhibe également la formation de biofilms, altère la biosynthèse de certains acides mycoliques, tels que les mycolyl-diacylglycérols qui forment la membrane mycobactérienne, et confère à la bactérie un phénotype hypermotile [12, 13]. Ces observations peuvent probablement être attribuées à un effet indirect de Lsr2, qui semble jouer un rôle dans la régulation de certaines enzymes liées au métabolisme des lipides de la paroi cellulaire [4]. Plusieurs études ont révélé le rôle de Lsr2 dans la régulation de gènes responsables de la synthèse et du transport des glycopeptidolipides (GPL), des lipides pariétaux complexes principalement retrouvés chez les mycobactéries non tuberculeuses (MNT). Ces composés lipidiques, en quantité importante chez les MNT, jouent un rôle majeur dans la structuration de leur paroi. La présence ou l’absence de GPL, qui conditionne respectivement l’existence de formes lisses ou rugueuses, affecte la motilité, la capacité de formation de biofilm, ainsi que la virulence des bactéries [14]. La délétion de lsr2 conduit à une augmentation de la production des GPL chez M. smegmatis [15].
Le rôle de Lsr2 dans la progression et la chronicité de l’infection a été démontré dans un modèle murin de tuberculose. Une absence de symptômes de la maladie et de lésions inflammatoires a été constatée dans le tissu pulmonaire de souris infectées par une souche dans laquelle Lsr2 avait été tronqué de son domaine C-terminal, même quatre semaines après l’infection [16]. Chez M. abscessus, un agent infectieux émergent dans la mucoviscidose, nous avons précédemment montré que l’expression accrue de lsr2 dans le morphotype rugueux, joue un rôle significatif dans sa virulence. Lsr2 joue un rôle physiologique important, tant dans les morphotypes lisses que rugueux de M. abscessus, et influence aussi la croissance intracellulaire de la bactérie au sein des macrophages et des amibes. Lsr2 contribue également à la virulence de la bactérie dans le modèle du poisson-zèbre et joue un rôle significatif dans sa persistance dans les poumons de souris infectées [5]. Chez M. abscessus et chez M. tuberculosis, Lsr2 occupe un rôle crucial dans la défense des bactéries contre les espèces réactives de l’oxygène, qui font partie des molécules anti-mycobactériennes les plus puissantes produites par l’hôte lors de l’infection intracellulaire des macrophages [5,16].
L’absence de viabilité in vitro des mutants de M. tuberculosis délétés de lsr2 a permis de montrer le caractère essentiel du gène lsr2 chez cette bactérie [17,18]. Comparativement, chez M. smegmatis [12] et M. abscessus [5], les souches dans lesquelles lsr2 est absent restent viables en culture planctonique3. Un aspect tout aussi significatif du rôle de Lsr2 dans le contrôle du cycle cellulaire a été rapporté [13] : pendant la phase de réplication de l’ADN, Lsr2 est localisée à proximité des réplisomes, suggérant son implication dans le bon déroulement de ce processus. Par la suite, Lsr2 migre vers les pôles cellulaires opposés de manière coordonnée avec les ségrosomes, démontrant l’existence d’un lien entre Lsr2 et le processus de ségrégation des chromosomes. L’absence de lsr2 a un impact à la fois sur le temps de réplication et sur la dynamique du réplisome.
L’expression de lsr2 est significative augmentée en réponse à divers types de stress, tels que la limitation des nutriments, les changements de température, ainsi que l’augmentation du fer et l’hypoxie, favorisant ainsi l’adaptation de la bactérie aux changements environnementaux [16, 19-22].
L’une des propriétés les plus intéressantes de Lsr2 est sa capacité à moduler la résistance aux antibiotiques [18]. Chez M. smegmatis, l’absence de Lsr2 induit une sensibilité accrue à l’acide nalidixique et à la rifampicine, ainsi qu’une augmentation de la perméabilité de la paroi bactérienne vis-à-vis de la vancomycine [22]. Récemment, notre équipe a observé une sensibilité plus marquée à l’amikacine liposomale et à la clarithromycine chez les souches de M. abscessus mutantes pour lsr2 lors d’infections de macrophages suivies de traitements à base de ces deux antibiotiques.
Finalement, outre l’implication de Lsr2 démontrée dans l’infection des cellules hôtes eucaryotes, Dulberger et al. [23] ont récemment mis en évidence le rôle de Lsr2 dans l’interaction entre mycobactéries et mycobactériophages. Cette étude a en effet révélé que Lsr2 joue un rôle crucial dans l’infection de la bactérie par le virus, en régulant la réplication des mycobactériophages dans les bactéries hôtes, et en établissant des zones de réplication de l’ADN viral. Bien que les mycobactériophages codent une protéine similaire à Lsr2, celle-ci ne semble cependant pas jouer de rôle dans ce processus.
Lsr2 : un facteur de transcription pléiotropiqueet un organisateur chromosomique
Lsr2 appartient à la superfamille des protéines associées au nucléoïde (ou NAP) [24]. Les NAP jouent un rôle essentiel dans la structure et la compaction des chromosomes, mais aussi dans la régulation génique au sein des cellules bactériennes [25, 26]. Parmi les protéines de cette superfamille, on peut citer H-NS (heat-stable nucleoid-structuring protein), Fis (factor for inversion stimulation), IHF (integration host factor), HU (heat unstable protein) et Dps (DNA-binding protein from starved cells) [27].
De nombreuses études ont mis en évidence le rôle des NAP dans la capacité de la bactérie à s’adapter à des conditions défavorables, en particulier en réponse au stress qu’elle rencontre lors de l’infection de son hôte. Ces protéines seraient ainsi responsables de l’induction de changements topologiques de l’ADN, afin de moduler le degré de condensation du chromosome ou de modifier le profil de transcription global de la bactérie [28, 29] ( Tableau I).
Les protéines associées au nucléoïde (NAP), facteurs de transcription impliqués dans la virulence des bactéries. Lsr2 : leprosy serum reactive clone 2 ; H-NS : heat-stable nucleoid-structuring protein ; Fis : factor for inversion stimulation ; IHF : integration host factor ; HU : heat unstable protein.
Lsr2 est composée de 110 à 120 acides aminés. Elle est riche en arginine, ce qui en fait une protéine hautement basique (pI : 10,69)4. Elle est principalement présente dans le cytosol des cellules mycobactériennes sous forme de dimères [4, 30-32]. Lsr2 se structure en deux domaines fonctionnels distincts, séparés par une région flexible : un domaine C-terminal, responsable de sa liaison à l’ADN, et un domaine N-terminal composé de deux sites de dimérisation qui lui permettent de s’oligomériser [31, 33]. Lsr2 est l’orthologue fonctionnel de la protéine H-NS, certainement une des NAP les plus étudiées que l’on retrouve exclusivement chez les entérobactéries [34, 35] (Figure 2). Des expériences de complémentation génétique ont montré que l’expression de lsr2 est capable de restaurer le phénotype lié à la mutation de hns chez E. coli. De même, chez les mutants de M. smegmatis dépourvus de lsr2, la complémentation de lsr2 par hns permet de rétablir la morphologie rugueuse des colonies altérée suite à la délétion du gène [35]. Bien que leurs séquences diffèrent fortement, ces protéines partagent d’importantes similitudes en ce qui concerne leur liaison à l’ADN, à la fois sur le plan structurel et sur le plan fonctionnel. Lsr2 et H-NS lient toutes deux préférentiellement les séquences riches en adénine et thymine (AT). Cette caractéristique suggère un rôle de ces protéines comme répresseurs xénogéniques de régions acquises par transfert horizontal, très souvent riches en AT [6, 7, 36, 37]. L’analyse structurale de Lsr2 a révélé une affinité plus marquée pour le sillon mineur de l’ADN5, impliquant un motif RGR (arginine-glycine-arginine) présent dans son domaine de liaison à l’ADN [37,38]. Une fois liée à l’ADN, Lsr2 peut recruter de nouveaux dimères via son domaine N-terminal, conduisant à la formation de filaments nucléoprotéiques rigides grâce à un mécanisme d’oligomérisation ( Figure 3 ). L’oligomérisation de Lsr2 a été démontrée à la fois in vitro, par des expériences de retard sur gel (electrophoretic mobility shift assay) [33], et in vivo par des expériences de ChIP-seq (chromatin immunoprecipitation with sequencing), réalisées chez M. tuberculosis [24] et, plus récemment, chez M. abscessus (résultats personnels). Si les conditions environnantes sont favorables, deux filaments nucléoprotéiques peuvent interagir ensemble et former un pont. Ce mécanisme de pontage peut non seulement connecter des fragments d’ADN proches, mais il pourrait également établir des connexions entre des régions plus distantes dans le même chromosome et contribuerait ainsi au repliement tridimensionnel du génome, notamment en facilitant la formation de boucles chromosomiques [37]. L’utilisation de la technique de microscopie à force atomique (AFM) a permis à Chen et al. de visualiser directement la formation de ces boucles résultant de l’interaction entre complexes Lsr2-ADN, tant à partir de matrices linéaires que de matrices circulaires [33]. Cependant l’existence de boucles dépendantes de Lsr2 dans le chromosome bactérien in vivo reste encore à démontrer.
 |
Figure 2. Modèles structuraux des homodimères de H-NS et de Lsr2. Les potentiels électriques de surface sont représentés sur chaque modèle structural de protomère à l’aide d’un gradient allant du rouge (charge négative) au bleu (charge positive). Les charges neutres sont représentées en blanc. Les « linkers », manquant dans le modèle de Lsr2, sont indiqués en bleu (chargés positivement) (figure adaptée de [38]). |
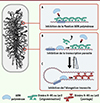 |
Figure 3. Mécanisme de répression des NAP de type H-NS (H-NS, Lsr2) dépendant de l’oligomérisation et du pontage d’ADN. Différents mécanismes ont été proposés pour expliquer le rôle de ces protéines dans la répression de l’expression des gènes. Le premier passe par la formation d’oligomères sur le promoteur, bloquant sa liaison à l’ARN polymérase (A). Le second dépend de la formation d’oligomères sur la région codante du gène cible, pouvant soit bloquer la production d’ARN parasites en empêchant la fixation d’ARN polymérases sur le corps du gène (B), soit bloquer l’élongation des transcrits du gène cible lors de la formation de ponts entre oligomères, se traduisant par un renforcement de leur liaison à l’ADN (C). |
En plus de leur rôle dans la conformation des chromosomes, les NAP sont également des facteurs de transcription intervenant dans la régulation des gènes impliqués dans divers processus cellulaires et moléculaires majeurs chez les bactéries [39, 40]. Lsr2 combine ces deux propriétés : celle d’une protéine associée au nucléoïde qui participe à l’organisation topologique du chromosome bactérien [41], et celle d’un régulateur pléiotrope agissant principalement comme un répresseur de la transcription [16]. Chez M. tuberculosis, Lsr2 module l’expression des protéines antigéniques et influence leurs interactions avec l’hôte, lui conférant ainsi un rôle dans la coordination des phases précoces d’infection latente et persistante [16]. Par ailleurs, Lsr2 joue un rôle crucial dans la modulation de l’expression d’une multitude de gènes liés à l’adaptation aux fluctuations des niveaux d’oxygène en se liant directement sur leur promoteur [22]. Récemment, nous avons montré, par une étude transcriptomique, le rôle régulateur de Lsr2 chez les deux morphotypes de M. abscessus, influençant la virulence, l’immunogénicité, le métabolisme énergétique et la résistance aux antibiotiques. Il a également été observé que la surexpression de lsr2 chez M. tuberculosis entraîne une répression très significative des gènes de virulence et induit des effets néfastes sur la capacité de la bactérie à déclencher une infection ou une maladie [42]. Ce rôle régulateur est également observé chez les streptomyces, tels que Streptomyces venezuelae, une bactérie connue pour sa production d’antibiotiques et d’autres composés bioactifs [6].
La distribution pangénomique de Lsr2 sur le chromosome des mycobactéries tend à confirmer son rôle en tant que facteur de transcription à effet pléiotropique. La technique de ChIP-seq a permis de montrer que Lsr2 est capable de lier une grande proportion des génomes de M. tuberculosis (21 %) et de M. smegmatis (13 %) [22, 24]. Chez M. tuberculosis, un enrichissement significatif de Lsr2 est observé pour environ 840 gènes. Ces gènes comprennent ceux codant les systèmes de sécrétion de type VII6 de la famille ESX (early antigenic secretion system), ceux impliqués dans la synthèse des lipides de la paroi cellulaire, ceux codant les protéines des familles PE/PPE7, ainsi que plusieurs facteurs de transcription [24]. Chez M. abscessus, Lsr2 se lie en particulier au niveau de la région promotrice du locus GPL, le locus rassemblant les gènes responsables de la synthèse et du transport des glycopeptidolipides [5].
Le mécanisme de répression transcriptionnelle exercée par Lsr2 chez les mycobactéries est encore loin d’être complètement élucidé. En revanche, le rôle de son orthologue H-NS dans la transcription ayant été plus largement étudié, il peut donc être utilisé comme modèle pour comprendre le mécanisme d’action de Lsr2 (Figure 3) [43].
Ce mécanisme repose sur la capacité d’oligomérisation de H-NS, en plus de sa fixation sur les régions promotrices de ses gènes cibles [26, 38]. H-NS exerce sa répression transcriptionnelle de diverses manières, étroitement liées à ses modes de liaison à l’ADN : rigidification et pontage. Son activité de répression intervient au moment de l’initiation de la transcription, les promoteurs liés à H-NS étant alors généralement rendus silencieux. Les oligomères présents sur ces séquences pourraient soit perturber la fixation de l’ARN polymérase, soit, au contraire, la piéger et ainsi inhiber l’initiation de la transcription. La présence d’oligomères de H-NS sur un nombre important de séquences codantes pourrait perturber l’élongation des transcrits in vitro, en réduisant la processivité ainsi qu’en augmentant les pauses de l’ARN polymérase. Il a été proposé que l’activité de pontage observée in vitro pour H-NS pourrait fortement renforcer ce mécanisme, suggérant ainsi que, en l’absence de pontage, le mouvement de l’ARN polymérase lors de l’élongation des transcrits suffirait à déplacer les dimères d’H-NS et ainsi désorganiser les oligomères. Cependant, ces derniers pourraient se reformer après son passage [43-45]. Ces oligomères empêcheraient en revanche la fixation de l’ARN polymérase sur des promoteurs cryptiques, responsables de la production de transcrits parasites et qui auraient un impact sur la croissance de la bactérie [46] ( Figure 3 ). H-NS peut aussi se lier, sous forme d’oligomères, au niveau des sites de terminaison de la transcription dépendante de Rho. Les interactions génétiques entre H-NS et Rho supposent que H-NS assisterait Rho dans la terminaison de la transcription [43, 47]. Les similitudes très importantes entre les modalités de liaison de H-NS et de Lsr2 suggèrent que les mécanismes de régulation exercés par ces deux protéines seraient analogues. De prochains résultats viendront certainement éclaircir cette hypothèse.
Modifications post-traductionnelles de Lsr2 et interactions avec d’autres NAP
Une seule étude a identifié une modification post-traductionnelle de Lsr2 chez M. tuberculosis : la phosphorylation de la thréonine 112 de son domaine C-terminal par la protéine kinase B [48]. La délétion du gène codant la protéine kinase B réduit significativement la phosphorylation de Lsr2 [49] et l’absence de phosphorylation entraîne une augmentation de la liaison de Lsr2 à l’ADN, conduisant à une perturbation de l’expression de nombreux gènes dont les produits participent à la croissance bactérienne, ainsi que de gènes impliqués dans l’adaptation de la bactérie à des conditions environnementales changeantes et défavorables.
L’interaction directe de la protéine Lsr2 avec d’autres NAP, ainsi que les effets de ces interactions sur sa liaison à l’ADN et sur la régulation de l’expression des gènes, ont été principalement étudiés dans le cas de la protéine HU, chez M. tuberculosis [50]. HU, grâce à son domaine C-terminal, interagit avec le domaine N-terminal de Lsr2 pour former des complexes nucléoprotéiques denses, observés lors d’expériences d’AFM.
Conclusion et perspectives
Lsr2 joue un rôle central dans la régulation de l’expression des gènes impliqués dans plusieurs processus cellulaires clés chez les mycobactéries, notamment la virulence, la réponse au stress, le métabolisme, la composition lipidique des membranes cellulaires et la résistance aux antibiotiques. En plus de son rôle de régulateur, la contribution possible de Lsr2 à la maintenance et à l’organisation hiérarchique des chromosomes en fait une cible prometteuse pour le développement de nouveaux agents antimicrobiens. À ce jour, seul le zafirlukast semble cibler directement Lsr2 [51]. Cette molécule thérapeutique, utilisée comme traitement prophylactique de l’asthme, perturbe l’interaction de Lsr2 avec l’ADN, ce qui impacte à la fois la transcription et la croissance de M. tuberculosis. Toutefois, d’autres études in vitro et in vivo seront nécessaires pour évaluer l’effet de ce médicament sur la protéine Lsr2 chez d’autres espèces de mycobactéries ainsi que pour révéler son potentiel thérapeutique dans la lutte contre les infections bactériennes. Des recherches supplémentaires visant à élucider le mécanisme par lequel Lsr2 agit en tant que répresseur transcriptionnel et à approfondir notre compréhension de ses interactions avec d’autres protéines régulatrices seront essentielles pour obtenir une vision complète de son impact sur la régulation génique chez les mycobactéries.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
La famille des actinobactéries ou actinomycètes est constituée d’une quarantaine de genres qui se distinguent les uns des autres essentiellement sur base de leur morphologie générale qui peut être extrêmement diversifiée. Ce sont des bactérie Gram-positives formant généralement des hyphes ramifiés et des spores asexuées.
Remerciements
Elias Gerges est bénéficiaire d’une bourse de thèse de Vaincre la Mucoviscidose et de l’Association Grégory Lemarchal (RF20200502727/2/1/53).
Références
- Laal S, Sharma YD, Prasad HK, et al. Recombinant fusion protein identified by lepromatous sera mimics native Mycobacterium leprae in T-cell responses across the leprosy spectrum. Proc Natl Acad Sci USA 1991 ; 88 : 1054–1058. [CrossRef] [PubMed] [Google Scholar]
- Sela S, Thole JE, Ottenhoff TH, et al. Identification of Mycobacterium leprae antigens from a cosmid library : characterization of a 15-kilodalton antigen that is recognized by both the humoral and cellular immune systems in leprosy patients. Infect Immun 1991 ; 59 : 4117–4124. [CrossRef] [PubMed] [Google Scholar]
- Oftung F, Mustafa AS, Wiker HGExtensive sequence homology between the mycobacterium leprae LSR (12 kDa) antigen and its Mycobacterium tuberculosis counterpart. FEMS Immunol Med Microbiol 2000 ; 27 : 87–89. [CrossRef] [PubMed] [Google Scholar]
- Chen JM, German GJ, Alexander DC, et al. Roles of Lsr2 in colony morphology and biofilm formation of Mycobacterium smegmatis. J Bacteriol 2006 ; 188 : 633–641. [CrossRef] [PubMed] [Google Scholar]
- Le Moigne V, Bernut A, Cortès M, et al. Lsr2 Is an Important Determinant of Intracellular Growth and Virulence in Mycobacterium abscessus. Front Microbiol 2019 ; 10 : 905. [CrossRef] [PubMed] [Google Scholar]
- Gehrke EJ, Zhang X, Pimentel-Elardo SM, et al. Silencing cryptic specialized metabolism in Streptomyces by the nucleoid-associated protein Lsr2. Elife 2019 ; 8 : e47691. [CrossRef] [PubMed] [Google Scholar]
- Wiechert J, Filipchyk A, Hünnefeld M, et al. Deciphering the Rules Underlying Xenogeneic Silencing and Counter-Silencing of Lsr2-like Proteins Using CgpS of Corynebacterium glutamicum as a Model. mBio 2020; 11 : e02273–19. [CrossRef] [PubMed] [Google Scholar]
- Pfeifer E, Hünnefeld M, Popa O, et al. Impact of Xenogeneic Silencing on Phage-Host Interactions. J Mol Biol 2019 ; 431 : 4670–4683. [CrossRef] [PubMed] [Google Scholar]
- Sharma V, Hardy A, Luthe T, et al. Phylogenetic Distribution of WhiB- and Lsr2-Type Regulators in Actinobacteriophage Genomes. Microbiol Spectr 2021; 9 : e0072721. [CrossRef] [PubMed] [Google Scholar]
- Singh S, Jenner PJ, Narayan NP, et al. Critical residues of the Mycobacterium leprae LSR recombinant protein discriminate clinical activity in erythema nodosum leprosum reactions. Infect Immun 1994 ; 62 : 5702–5705. [CrossRef] [PubMed] [Google Scholar]
- Saini C, Prasad HK, Rani R, et al. Lsr2 of Mycobacterium leprae and its synthetic peptides elicit restitution of T cell responses in erythema nodosum leprosum and reversal reactions in patients with lepromatous leprosy. Clin Vaccine Immunol 2013 ; 20 : 673–682. [CrossRef] [PubMed] [Google Scholar]
- Arora K, Whiteford DC, Lau-Bonilla D, et al. Inactivation of lsr2 results in a hypermotile phenotype in Mycobacterium smegmatis. J Bacteriol 2008 ; 190 : 4291–4300. [CrossRef] [PubMed] [Google Scholar]
- Kołodziej M, Trojanowski D, Bury K, et al. Lsr2, a nucleoid-associated protein influencing mycobacterial cell cycle. Sci Rep 2021; 11 : 2910. [CrossRef] [PubMed] [Google Scholar]
- Gutiérrez AV, Viljoen A, Ghigo E, et al. Glycopeptidolipids, a Double-Edged Sword of the Mycobacterium abscessus Complex. Front Microbiol 2018 ; 9 : 1145. [CrossRef] [PubMed] [Google Scholar]
- Kocíncová D, Singh AK, Beretti J-L, et al. Spontaneous transposition of IS1096 or ISMsm3 leads to glycopeptidolipid overproduction and affects surface properties in Mycobacterium smegmatis. Tuberculosis (Edinb) 2008 ; 88 : 390–398. [CrossRef] [PubMed] [Google Scholar]
- Bartek IL, Woolhiser LK, Baughn AD, et al. Mycobacterium tuberculosis Lsr2 is a global transcriptional regulator required for adaptation to changing oxygen levels and virulence. mBio 2014; 5 : e01106–01114. [CrossRef] [PubMed] [Google Scholar]
- Sassetti CM, Boyd DH, Rubin EJGenes required for mycobacterial growth defined by high density mutagenesis. Mol Microbiol 2003 ; 48 : 77–84. [CrossRef] [PubMed] [Google Scholar]
- Colangeli R, Helb D, Vilchèze C, et al. Transcriptional regulation of multi-drug tolerance and antibiotic-induced responses by the histone-like protein Lsr2 in M. tuberculosis. PLoS Pathog 2007 ; 3 : e87. [CrossRef] [PubMed] [Google Scholar]
- Wong DK, Lee BY, Horwitz MA, et al. Identification of fur, aconitase, and other proteins expressed by Mycobacterium tuberculosis under conditions of low and high concentrations of iron by combined two-dimensional gel electrophoresis and mass spectrometry. Infect Immun 1999 ; 67 : 327–336. [CrossRef] [PubMed] [Google Scholar]
- Betts JC, Lukey PT, Robb LC, et al. Evaluation of a nutrient starvation model of Mycobacterium tuberculosis persistence by gene and protein expression profiling. Mol Microbiol 2002 ; 43 : 717–731. [CrossRef] [PubMed] [Google Scholar]
- Stewart GR, Wernisch L, Stabler R, et al. Dissection of the heat-shock response in Mycobacterium tuberculosis using mutants and microarrays. Microbiology (Reading) 2002 ; 148 : 3129–3138. [CrossRef] [PubMed] [Google Scholar]
- Kołodziej M, Łebkowski T, Płocin’ ski P, et al. Lsr2 and Its Novel Paralogue Mediate the Adjustment of Mycobacterium smegmatis to Unfavorable Environmental Conditions. mSphere 2021; 6 : e00290–21. [PubMed] [Google Scholar]
- Dulberger CL, Guerrero-Bustamante CA, Owen SV, et al. Mycobacterial nucleoid-associated protein Lsr2 is required for productive mycobacteriophage infection. Nat Microbiol 2023; 8 : 695–710. [CrossRef] [PubMed] [Google Scholar]
- Gordon BRG, Li Y, Wang L, et al. Lsr2 is a nucleoid-associated protein that targets AT-rich sequences and virulence genes in Mycobacterium tuberculosis. Proc Natl Acad Sci USA 2010 ; 107 : 5154–5159. [CrossRef] [PubMed] [Google Scholar]
- Dame RTThe role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin. Mol Microbiol 2005 ; 56 : 858–870. [CrossRef] [PubMed] [Google Scholar]
- Dame RT, Rashid F-ZM, Grainger DC. Chromosome organization in bacteria : mechanistic insights into genome structure and function. Nat Rev Genet 2020; 21 : 227–42. [CrossRef] [PubMed] [Google Scholar]
- Dillon SC, Dorman CJBacterial nucleoid-associated proteins, nucleoid structure and gene expression. Nat Rev Microbiol 2010 ; 8 : 185–195. [CrossRef] [PubMed] [Google Scholar]
- Hołówka J, Zakrzewska-Czerwin’ska J. Nucleoid Associated Proteins : The Small Organizers That Help to Cope With Stress. Front Microbiol 2020; 11 : 590. [CrossRef] [PubMed] [Google Scholar]
- Rajapaksha N, Soldano A, Yao H, et al. Pseudomonas aeruginosa Dps (PA0962) Functions in H2O2 Mediated Oxidative Stress Defense and Exhibits In Vitro DNA Cleaving Activity. Int J Mol Sci 2023; 24 : 4669. [CrossRef] [PubMed] [Google Scholar]
- Marques MAM, Espinosa BJ, Xavier da Silveira EK, et al. Continued proteomic analysis of Mycobacterium leprae subcellular fractions. Proteomics 2004 ; 4 : 2942–2953. [CrossRef] [PubMed] [Google Scholar]
- Summers EL, Meindl K, Usón I, et al. The structure of the oligomerization domain of Lsr2 from Mycobacterium tuberculosis reveals a mechanism for chromosome organization and protection. PLoS One 2012 ; 7 : e38542. [CrossRef] [PubMed] [Google Scholar]
- Qu Y, Lim CJ, Whang YR, et al. Mechanism of DNA organization by Mycobacterium tuberculosis protein Lsr2. Nucleic Acids Res 2013 ; 41 : 5263–5272. [CrossRef] [PubMed] [Google Scholar]
- Chen JM, Ren H, Shaw JE, et al. Lsr2 of Mycobacterium tuberculosis is a DNA-bridging protein. Nucleic Acids Res 2008 ; 36 : 2123–2135. [CrossRef] [PubMed] [Google Scholar]
- Dorman CJH-NS : a universal regulator for a dynamic genome. Nat Rev Microbiol 2004 ; 2 : 391–400. [CrossRef] [PubMed] [Google Scholar]
- Gordon BRG, Imperial R, Wang L, et al. Lsr2 of Mycobacterium represents a novel class of H-NS-like proteins. J Bacteriol 2008 ; 190 : 7052–7059. [CrossRef] [PubMed] [Google Scholar]
- Navarre WW, McClelland M, Libby SJ, et al. Silencing of xenogeneic DNA by H-NS-facilitation of lateral gene transfer in bacteria by a defense system that recognizes foreign DNA. Genes Dev 2007 ; 21 : 1456–1471. [CrossRef] [PubMed] [Google Scholar]
- Gordon BRG, Li Y, Cote A, et al. Structural basis for recognition of AT-rich DNA by unrelated xenogeneic silencing proteins. Proc Natl Acad Sci USA 2011 ; 108 : 10690–10695. [CrossRef] [PubMed] [Google Scholar]
- Qin L, Erkelens AM, Ben Bdira F, et al. The architects of bacterial DNA bridges : a structurally and functionally conserved family of proteins. Open Biol 2019 ; 9 : 190223. [CrossRef] [PubMed] [Google Scholar]
- Dorman CJ. Function of nucleoid-associated proteins in chromosome structuring and transcriptional regulation. J Mol Microbiol Biotechnol 2014 ; 24 : 316–331. [PubMed] [Google Scholar]
- Amemiya HM, Schroeder J, Freddolino PL. Nucleoid-associated proteins shape chromatin structure and transcriptional regulation across the bacterial kingdom. Transcription 2021; 12 : 182–218. [CrossRef] [PubMed] [Google Scholar]
- Kriel NL, Gallant J, van Wyk N, et al. Mycobacterial nucleoid associated proteins : An added dimension in gene regulation. Tuberculosis (Edinb) 2018 ; 108 : 169–177. [CrossRef] [PubMed] [Google Scholar]
- Liu J, Gordon BRGTargeting the global regulator Lsr2 as a novel approach for anti-tuberculosis drug development. Expert Rev Anti Infect Ther 2012 ; 10 : 1049–1053. [CrossRef] [PubMed] [Google Scholar]
- Kotlajich MV, Hron DR, Boudreau BA, et al. Bridged filaments of histone-like nucleoid structuring protein pause RNA polymerase and aid termination in bacteria. Elife 2015 ; 4 : e04970. [CrossRef] [PubMed] [Google Scholar]
- Boudreau BA, Hron DR, Qin L, et al. StpA and Hha stimulate pausing by RNA polymerase by promoting DNA-DNA bridging of H-NS filaments. Nucleic Acids Res 2018 ; 46 : 5525–5546. [CrossRef] [PubMed] [Google Scholar]
- Wade JT, Grainger DCWaking the neighbours : disruption of H-NS repression by overlapping transcription. Mol Microbiol 2018 ; 108 : 221–225. [CrossRef] [PubMed] [Google Scholar]
- Singh SS, Singh N, Bonocora RP, et al. Widespread suppression of intragenic transcription initiation by H-NS. Genes Dev 2014 ; 28 : 214–219. [CrossRef] [PubMed] [Google Scholar]
- Peters JM, Mooney RA, Grass JA, et al. Rho and NusG suppress pervasive antisense transcription in Escherichia coli. Genes Dev 2012 ; 26 : 2621–2633. [CrossRef] [PubMed] [Google Scholar]
- Alqaseer K, Turapov O, Barthe P, et al. Protein kinase B controls Mycobacterium tuberculosis growth via phosphorylation of the transcriptional regulator Lsr2 at threonine 112. Mol Microbiol 2019 ; 112 : 1847–1862. [CrossRef] [PubMed] [Google Scholar]
- Turapov O, Forti F, Kadhim B, et al. Two Faces of CwlM, an Essential PknB Substrate, in Mycobacterium tuberculosis. Cell Reports 2018 ; 25 : 57. [CrossRef] [PubMed] [Google Scholar]
- Datta C, Jha RK, Ahmed W, et al. Physical and functional interaction between nucleoid-associated proteins HU and Lsr2 of Mycobacterium tuberculosis : altered DNA binding and gene regulation. Mol Microbiol 2019 ; 111 : 981–994. [CrossRef] [PubMed] [Google Scholar]
- Pinault L, Han J-S, Kang C-M, et al. Zafirlukast inhibits complexation of Lsr2 with DNA and growth of Mycobacterium tuberculosis. Antimicrob Agents Chemother 2013 ; 57 : 2134–2140. [CrossRef] [PubMed] [Google Scholar]
- Ares MA, Fernández-Vázquez JL, Rosales-Reyes R, et al. H-NS Nucleoid Protein Controls Virulence Features of Klebsiella pneumoniae by Regulating the Expression of Type 3 Pili and the Capsule Polysaccharide. Front Cell Infect Microbiol 2016 ; 6 : 13. [PubMed] [Google Scholar]
- Deng X, Li M, Pan X, et al. Fis Regulates Type III Secretion System by Influencing the Transcription of exsA in Pseudomonas aeruginosa Strain PA14. Front Microbiol 2017 ; 8 : 669. [PubMed] [Google Scholar]
- Leite B, Werle CH, Carmo CP do, et al. Integration host factor is important for biofilm formation by Salmonella enterica Enteritidis. Pathog Dis 2017; 75. doi : 10.1093/femspd/ftx074. [Google Scholar]
- Jurcisek JA, Brockman KL, Novotny LA, et al. Nontypeable Haemophilus influenzae releases DNA and DNABII proteins via a T4SS-like complex and ComE of the type IV pilus machinery. Proc Natl Acad Sci USA 2017 ; 114 : E6632–E6641. [CrossRef] [PubMed] [Google Scholar]
Liste des tableaux
Les protéines associées au nucléoïde (NAP), facteurs de transcription impliqués dans la virulence des bactéries. Lsr2 : leprosy serum reactive clone 2 ; H-NS : heat-stable nucleoid-structuring protein ; Fis : factor for inversion stimulation ; IHF : integration host factor ; HU : heat unstable protein.
Liste des figures
 |
Figure 1. Lsr2, déterminant physiologique important chez les mycobactéries. Illustration des principaux rôles décrits pour Lsr2 en tant que protéine associée au nucléoïde chez trois groupes de mycobactéries : les mycobactéries pathogènes strictes (Mycobacterium leprae et Mycobacterium tuberculosis) ; les mycobactéries opportunistes (Mycobacterium abscessus) ; et les mycobactéries saprophytes (Mycobacterium smegmatis). IFN-g : interféron gamma. |
| Dans le texte | |
 |
Figure 2. Modèles structuraux des homodimères de H-NS et de Lsr2. Les potentiels électriques de surface sont représentés sur chaque modèle structural de protomère à l’aide d’un gradient allant du rouge (charge négative) au bleu (charge positive). Les charges neutres sont représentées en blanc. Les « linkers », manquant dans le modèle de Lsr2, sont indiqués en bleu (chargés positivement) (figure adaptée de [38]). |
| Dans le texte | |
 |
Figure 3. Mécanisme de répression des NAP de type H-NS (H-NS, Lsr2) dépendant de l’oligomérisation et du pontage d’ADN. Différents mécanismes ont été proposés pour expliquer le rôle de ces protéines dans la répression de l’expression des gènes. Le premier passe par la formation d’oligomères sur le promoteur, bloquant sa liaison à l’ARN polymérase (A). Le second dépend de la formation d’oligomères sur la région codante du gène cible, pouvant soit bloquer la production d’ARN parasites en empêchant la fixation d’ARN polymérases sur le corps du gène (B), soit bloquer l’élongation des transcrits du gène cible lors de la formation de ponts entre oligomères, se traduisant par un renforcement de leur liaison à l’ADN (C). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




