| Issue |
Med Sci (Paris)
Volume 39, Number 4, Avril 2023
|
|
|---|---|---|
| Page(s) | 331 - 333 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2023050 | |
| Published online | 24 April 2023 | |
Peste noire, sélection naturelle et susceptibilité aux maladies auto-immunes ou auto-inflammatoires
The Black Death, natural selection and susceptibility to auto-immune disorders
1
Unité de recherche Yersinia, Institut Pasteur, université Paris Cité, CNRS UMR6047, Paris, France
2
McMaster Ancient DNA Centre, Departments of anthropology, biology and biochemistry, université McMaster, Hamilton, Ontario, Canada
3
Michael G. DeGroote Institute of infectious disease research, université McMaster, Hamilton, Ontario, Canada
4
Section of genetic medicine, Department of medicine, université de Chicago, Chicago, États-Unis
5
Department of human genetics, université de Chicago, Chicago, États-Unis
« Le fléau n’est pas à la mesure de l’homme, on se dit donc que le fléau est irréel, c’est un mauvais rêve qui va passer. Mais il ne passe pas toujours et, de mauvais rêve en mauvais rêve, ce sont les hommes qui passent… Ils se croyaient libres et personne ne sera jamais libre tant qu’il y aura des fléaux »
Albert Camus (La Peste)
Pour les êtres vivants, survivre dans la nature n’est pas seulement un enjeu individuel, mais c’est aussi, pour la survie de l’espèce, une question de transmission du patrimoine génétique. L’environnement exerce sur les individus des pressions de sélection face auxquelles ils sont inégalement dotés. Porter le variant allélique avantageux d’un gène déterminant peut alors devenir une condition nécessaire pour atteindre l’âge de se reproduire et transmettre son patrimoine génétique à sa descendance. Au fil du temps, la fréquence des allèles désavantageux diminuerait donc jusqu’à leur éventuelle disparition, tandis que les allèles avantageux prédomineraient dans la population.
Les maladies infectieuses font partie de ces pressions de sélection soupçonnées d’avoir façonné le patrimoine génétique de l’humanité, de manière insidieuse sur le long terme, ou plus brutalement lors de pandémies, en empêchant certains individus de procréer. Parmi elles, peu ont frappé l’humanité aussi violemment et sur de brèves périodes que les pandémies de peste, et notamment la grande peste du moyen-âge, qui tua jusqu’à la moitié de la population européenne. La peste aurait-elle effectué une sélection positive d’allèles pouvant être considérés comme protecteurs parce que portés plus souvent par les individus survivants que par ceux qui ont succombé à la peste ? C’est la question à laquelle nous avons tenté de répondre dans un article récent [1], en utilisant une approche de paléogénomique1 [2] (→).
(→) Voir le Repères de E. Heyer, m/s n° 2, février 2023, page 181
Fondée sur l’étude de l’ADN ancien, cette nouvelle science donne accès à des génomes datant parfois de plusieurs milliers d’années, ce qui permet de mettre en évidence l’adaptation du génome humain consécutive à des évènements aussi anciens.
Historiquement bien documentés et datés, les cimetières anciens de Londres ou du Danemark recèlent des squelettes de personnes ayant vécu juste avant, pendant, ou juste après la peste noire (en anglais, « black death »), une pandémie survenue au Moyen-Âge, entre les années 1347 et 1352 [12] (→).
(→) Voir la Chronique génomique de B. Jordan, page 381 de ce numéro
Nous avons ainsi pu reconstituer les génomes de ces individus afin d’analyser la séquence nucléotidique des gènes du système immunitaire, déterminants pour l’issue de l’infection. Cette approche a permis, par la détection des différences alléliques entre ces génomes, d’identifier plusieurs gènes potentiellement associés à la résistance à la peste. Le plus fort signal de sélection positive est apparu pour une copie fonctionnelle du gène codant la protéine ERAP2 (endoplasmic reticulum-associated aminopeptidase 2), une enzyme protéolytique présente dans la plupart des cellules immunitaires, active dans la présentation antigénique aux lymphocytes T, et au cours de la signalisation entre cellules [3] (Figure 1). Ce gène est présent dans les cellules d’hommes et de femmes vivant actuellement, sous la forme de différents variants alléliques, fonctionnels ou non. En infectant des cellules possédant ou non un allèle fonctionnel du gène ERAP2 avec le bacille de la peste (Yersinia pestis), nous avons confirmé que la présence de la protéine ERAP2 augmentait la capacité de ces cellules à tuer cette bactérie et contrôlait la production de plusieurs molécules qui facilitent l’activation du système immunitaire. Posséder « la bonne version » de ce gène avait donc vraisemblablement été un atout pour la survie des Londoniens et des Danois du xiv e siècle confrontés à l’épidémie de peste. Selon notre estimation (en utilisant un modèle de Markov caché)2, un individu possédant deux allèles fonctionnels de ERAP2 aurait eu environ 40 % de chances supplémentaires de survivre à la peste noire par rapport à un individu possédant deux allèles non fonctionnels de ce gène.
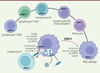 |
Figure 1. Les cellules du système immunitaire produisant la protéine ERAP2, et son rôle dans la réponse immunitaire. La protéase ERAP2 contribue à découper les protéines exogènes internalisées par la cellule immunocompétente en peptides courts susceptibles d’être présentés aux lymphocytes T en association avec protéines du complexe majeur d’histocompatibilité de classe I (CMH-I), ce qui induit l’activation de ces lymphocytes, importante pour la réponse immunitaire adaptative. La protéine ERAP2 sécrétée par les macrophages a également la capacité d’activer d’autres macrophages [11]. Figure réalisée avec le logiciel BioRender. |
Ce n’est pas la première fois qu’est émise l’hypothèse selon laquelle la peste noire aurait été une cause importante de sélection pour le système immunitaire. Les gènes codant des variants du récepteur FPR1 (un « récepteur » pour Yersinia pestis) ou de la pyrine [4, 5] auraient eux aussi été soumis à cette pression. Toutefois, ces travaux sont fondés au mieux sur des populations humaines contemporaines, et l’évolution de ces gènes a pu être influencée par diverses autres pressions de sélection (autres maladies infectieuses, famines, guerres, changements climatiques), qui ont pu agir et se combiner au cours des siècles. Dans notre étude [1], cette objection peut quasiment être écartée du fait de l’intervalle temporel très court séparant les sujets ayant vécu avant et ceux ayant vécu après l’épidémie de peste noire. Ainsi, cette étude paléogénomique montre que la sélection naturelle, plutôt lente dans l’espèce humaine du fait de la longueur de son temps de génération et de sa population réduite par rapport à celles d’autres espèces, peut agir sur l’humanité en un temps court. La paléogénomique a d’ailleurs permis d’identifier d’autres épidémies ayant laissé leur empreinte sur le génome humain [6]. De tels travaux de recherche peuvent donc renseigner sur certains mécanismes qui ont façonné le système immunitaire humain au fil du temps.
Cependant, la présence d’un gène ERAP2 fonctionnel chez un individu est aussi associée à un risque accru de maladie de Crohn (une maladie inflammatoire chronique de l’intestin), tout comme celle d’un autre gène identifié dans notre étude, CTLA4 (Cytotoxic T-lymphocyte-associated protein 4), qui code un récepteur présent à la surface de certains lymphocytes T, est associée au risque d’arthrite rhumatoïde et de lupus érythémateux disséminé. Ainsi, des allèles qui ont pu procurer à l’individu une certaine protection contre la peste pendant des siècles s’avèrent aujourd’hui être liés à des dysfonctionnements du système immunitaire responsables d’auto-immunité et d’inflammation chronique. Les autres gènes dont certains allèles ont potentiellement été sélectionnés par la peste noire semblent répondre à la même logique : ainsi, la présence d’un gène FPR1 (formyl peptide receptor 1) fonctionnel est un facteur de risque pour certains cancers [4], et des mutations de MEFV (mediterranean fever), le gène codant la pyrine, sont responsables de la fièvre méditerranéenne familiale, une maladie auto-inflammatoire caractérisée par des accès fébriles récurrents [5]. Un système immunitaire « hyperactif » a peut-être été un outil très utile dans le passé pour lutter contre la bactérie Yersinia pestis, mais en l’absence de la menace infectieuse, il peut s’avérer nuisible. La prévalence accrue de ces maladies auto-immunes ou auto-inflammatoires pourrait représenter le prix que les populations humaines ont payé pour survivre à la peste.
Que peut-on espérer de l’identification de gènes comme ERAP2 en matière de santé publique ? Le génotypage par la technique de PCR (polymerase chain reaction) permet d’identifier les allèles de ce gène, fonctionnels ou défectueux, qui sont présents chez un individu, et pourrait renseigner non seulement sur le risque de maladie de Crohn, mais aussi sur une sensibilité accrue à certaines maladies infectieuses. En effet, les résultats d’une étude récente ont confirmé que l’expression de ERAP2 est associée à une protection contre plusieurs maladies infectieuses, mais qu’elle augmente également le risque de maladie auto-immune [7].
Grâce à l’étude de l’ADN ancien, la paléogénomique est en train d’opérer une révolution dans notre compréhension de l’évolution du monde vivant. La reconstitution de génomes a ainsi permis de montrer par exemple que la bactérie Yersinia pestis était bien la cause des trois pandémies historiques de peste [8], que l’homme de Néandertal nous a transmis environ 3 % de son génome [9] (→) ou que la majeure partie de l’adaptation génétique humaine s’est produite après le début de l’âge du bronze, il y a moins de 4 500 ans, et est associée à un risque accru de maladies inflammatoires [10]. Cette étude sur la peste noire soulève également une question d’actualité : le coronavirus SARS-CoV-2 est-il en train de sélectionner une sous-population humaine ? C’est en réalité peu probable, car la mortalité de la pandémie actuelle de Covid-19 n’est pas comparable à celle des pandémies de peste. Le SARS-CoV-2 a surtout causé la mort de personnes âgées, alors que la peste noire a eu un effet bien plus dévastateur sur la population de jeunes individus, les empêchant ainsi de transmettre leurs gènes à une descendance.
(→) Voir la Synthèse de M. Deschamps et L. Quintana-Murci, m/s n° 12, décembre 2016, page 1079
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
La paléogénomique est une discipline biologique récente qui a pour objectifs la reconstitution et l’analyse des génomes anciens : animaux, végétaux, ou microbiens. Son précurseur, Sven Pääbo, a reçu le Prix Nobel de physiologie ou médecine en 2022 [2].
Modèle statistique dans lequel le système modélisé est supposé être un processus markovien de paramètres inconnus. Contrairement à une chaîne de Markov classique, où les transitions prises sont inconnues de l’utilisateur mais où les états d’une exécution sont connus, dans un modèle de Markov caché, les états d’une exécution sont inconnus de l’utilisateur.
Références
- Klunk J, Vilgalys TP, Demeure CE, et al. Evolution of immune genes is associated with the Black Death. Nature 2022; 611 : 312–9. [CrossRef] [PubMed] [Google Scholar]
- Heyer E. Prix Nobel de physiologie ou médecine 2022 : Svante Pääbo, précurseur de la paléogénomique. Med Sci (Paris) 2023; 39 : 181–3. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Saulle I, Vicentini C, Clerici M, Biasin M. An overview on ERAP roles in infectious diseases. Cells 2020; 9 : 720. [CrossRef] [PubMed] [Google Scholar]
- Osei-Owusu P, Charlton TM, Kim HK, et al. FPR1 is the plague receptor on host immune cells. Nature 2019 ; 574 : 57–62. [CrossRef] [PubMed] [Google Scholar]
- Park YH, Remmers EF, Lee W, et al. Ancient familial Mediterranean fever mutations in human pyrin and resistance to Yersinia pestis. Nat Immunol 2020; 21 : 857–67. [CrossRef] [PubMed] [Google Scholar]
- Souilmi Y, Lauterbur ME, Tobler R, et al. An ancient viral epidemic involving host coronavirus interacting genes more than 20,000 years ago in East Asia. Curr Biol 2021; 31 : 3504–14.e9. [CrossRef] [PubMed] [Google Scholar]
- Hamilton F, Mentzer A, Parks T, et al. Variation in ERAP2 has opposing effects on severe respiratory infection and autoimmune disease. medBioRXiv 2022; doi : 10.1101/2022.11.04.22281942. [Google Scholar]
- Wagner DM, Klunk J, Harbeck M, et al. Yersinia pestis and the plague of Justinian 541–3 AD: a genomic analysis. Lancet Infect Dis 2014 ; 14 : 319–326. [CrossRef] [PubMed] [Google Scholar]
- Deschamps M, Quintana-Murci L. Immunité innée et maladies chez l’homme : de l’introgression archaïque à la sélection naturelle. Med Sci (Paris) 2016 ; 32 : 1079–1086. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Kerner G, Neehus AL, Philippot Q, et al. Genetic adaptation to pathogens and increased risk of inflammatory disorders in post-Neolithic Europe. Cell Genomics 2023; doi : 10.1016/j.xgen.2022.100248. [PubMed] [Google Scholar]
- Saulle I, Marventano I, Saresella M, et al. ERAPs reduce in vitro HIV infection by activating innate immune response. J Immunol 2021; 206 : 1609–17. [CrossRef] [PubMed] [Google Scholar]
- Jordan B. Les traces génétiques de la peste noire. Med Sci (Paris) 2023; 39 : 381–3. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
© 2023 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Les cellules du système immunitaire produisant la protéine ERAP2, et son rôle dans la réponse immunitaire. La protéase ERAP2 contribue à découper les protéines exogènes internalisées par la cellule immunocompétente en peptides courts susceptibles d’être présentés aux lymphocytes T en association avec protéines du complexe majeur d’histocompatibilité de classe I (CMH-I), ce qui induit l’activation de ces lymphocytes, importante pour la réponse immunitaire adaptative. La protéine ERAP2 sécrétée par les macrophages a également la capacité d’activer d’autres macrophages [11]. Figure réalisée avec le logiciel BioRender. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




