| Issue |
Med Sci (Paris)
Volume 39, Number 4, Avril 2023
|
|
|---|---|---|
| Page(s) | 351 - 358 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2023052 | |
| Published online | 24 April 2023 | |
L’organisation native de la cellule révélée grâce à la cryo-microscopie à expansion
Closer to the native architecture of the cell using Cryo-ExM
Department of Molecular and Cellular Biology, Université de Genève, 30 quai Ernest Ansermet, 1211 Genève, Suisse
* Co-auteurs correspondants: virginie.hamel@unige.ch
** paul.guichard@unige.ch
La plupart des techniques d’imagerie cellulaire, telles que la microscopie photonique ou la microscopie électronique, nécessitent que l’échantillon biologique soit préalablement fixé par des agents chimiques, une étape qui est connue pour endommager l’organisation sub-cellulaire. Pour pallier à ce problème, la cryo-fixation, inventée il y a plus de 40 ans, consiste à vitrifier les échantillons biologiques afin de préserver leur état natif. Cette méthode n’avait cependant été que très peu utilisée en microscopie photonique. Dans cette revue, nous présentons en détail la microscopie d’expansion, une technique de super-résolution développée récemment et qui, couplée à la cryo-fixation, permet de visualiser l’architecture cellulaire au plus près de son état natif.
Abstract
Most cellular imaging techniques, such as light or electron microscopy, require that the biological sample is first fixed by chemical cross-linking agents. This necessary step is also known to damage molecular nanostructures or even sub-cellular organization. To overcome this problem, another fixation approach was invented more than 40 years ago, which consists in cryo-fixing biological samples, thus allowing to preserve their native state. However, this method has been scarcely used in light microscopy due to the complexity of its implementation. In this review, we present a recently developed super-resolution method called expansion microscopy, which, when coupled with cryo-fixation, allows to visualize at a nanometric resolution the cell architecture as close as possible to its native state.
© 2023 médecine/sciences – Inserm
Vignette (© Guichard/Hamel Lab, Université de Genève).
L’imagerie cellulaire en biologie
Deux types d’imagerie sont couramment utilisés dans les laboratoires de biologie cellulaire et moléculaire pour observer les cellules : la microscopie photonique et la microscopie électronique. Comme leurs noms l’indiquent, il s’agit de méthodes qui utilisent des sources de différentes longueurs d’ondes, les photons ou les électrons. En microscopie photonique, les longueurs d’ondes monochromatiques utilisées correspondent aux longueurs d’ondes qui composent la lumière blanche (de 400 nm à 800 nm). Quand ces ondes passent à travers une ouverture, celles-ci vont diffuser et créer une tache lumineuse, appelée « tache d’Airy », spécifique pour chacune des longueurs d’ondes utilisées. Ce phénomène de diffraction limite le pouvoir de résolution car deux taches trop proches deviennent indiscernables. L’observation de structures sub-cellulaires est ainsi limitée par la diffraction, définie par la formule d’Ernst Abbe (1840-1905) [1]. En simplifiant cette formule, il est possible de déterminer la résolution atteignable en divisant la longueur d’onde par l’ouverture numérique (ON), ce qui donne une résolution maximale de 160 à 200 nm selon les paramètres optiques utilisés. Par ailleurs, il faut rappeler que la microscopie photonique repose majoritairement sur l’utilisation d’anticorps spécifiques de protéines d’intérêt, couplés à un fluorochrome, qui, excité à une longueur d’onde donnée, émet un spectre lumineux capté par des détecteurs. S’ajoute donc aux limites de résolution une perte de précision, de l’ordre de 15 à 20 nm, qui correspond à la taille des anticorps couplés aux fluorochromes (aussi appelée linkage error). En raison de ces limitations, il est donc difficile de visualiser une grande majorité des structures cellulaires dont la taille se situent en dessous de la limite de résolution de la microscopie photonique, telles que les organites (moins de 500 nm) ou les protéines (environ 10 nm).
La microscopie électronique permet de contourner cette limite. Elle repose sur l’utilisation d’une source d’électrons dont la longueur d’onde peut atteindre quelques picomètres (pm). Néanmoins, la microscopie électronique requiert que l’échantillon soit fixé chimiquement, enrobé dans un polymère résistant aux électrons, puis coupé en fines sections, et, enfin, coloré avec des colorants aspécifiques, tels que le sel d’uranium et l’osmium. Dans ces conditions de préparation, qui peuvent affecter l’organisation nanométrique des biomolécules, la résolution effective de la microscopie électronique cellulaire est d’environ 5 à 10 nm.
Dès lors, nous disposons de deux méthodes complémentaires : la microscopie photonique, qui permet la localisation spécifique de biomolécules, mais avec une résolution limitée ; et la microscopie électronique, 20 à 40 fois plus résolutive, mais qui révèle essentiellement l’ultrastructure sans marquage spécifique. Il existe donc une « zone grise » du vivant difficilement visualisable qui correspond à l’organisation nanométrique de protéines spécifiques dans une structure sub-cellulaire. Afin de palier à ce manque, une nouvelle forme de microscopie à fluorescence est apparue il y quelques années : la microscopie à fluorescence à super-résolution, aussi appelée nanoscopie [2]. Cette méthode regroupe principalement trois approches de microscopies photoniques capables de franchir la barrière de la diffraction de la lumière : le SIM (structured illumination microscopy), le STED (stimulated emission depletion) et le SMLM (single molecule light microscopy) [3]. En 2014, Eric Betzig, William Moerner, et Stefan Hell ont été récompensé par le prix Nobel de chimie pour la mise au point de cette technique de fluorescence à super-résolution. Ces approches, qui reposent sur le développement de microscopes de pointe et d’algorithmes de reconstructions d’images, ont des résolutions comprises entre 10 et 120 nm. Elles ont permis de révolutionner nos connaissances en biologie cellulaire, en microbiologie et en neurobiologie [4]. Il est en effet à présent possible de localiser à l’échelle nanométrique des constituants sub-cellulaires [5]. Cependant, leur utilisation nécessite une expertise et des microscopes spécifiques, limitant leur exploitation à certains laboratoires de biologie.
La microscopie à expansion : principe et développement
La méthode de microscopie à expansion (ou ExM) [6], développée en 2015 dans le laboratoire de Edward S. Boyden, professeur de neurotechnologie au Massachusetts Institute of Technology (MIT) à Cambridge (États-Unis), est une nouvelle technique de microscopie photonique. Cette méthode appartient également à la classe des microscopies à super-résolution [3] mais contrairement aux autres techniques que nous avons présentées, elle consiste à enrober les échantillons biologiques dans un polymère capable de s’expandre de façon isotropique (dans toutes les directions). Elle permet ainsi d’augmenter physiquement la taille des échantillons d’un facteur quatre, ou plus, améliorant la résolution et donc leur observation en utilisant des microscopes photoniques classiques. L’utilisation de microscopes conventionnels est l’un des avantages les plus importants de la microscopie à expansion. En effet, contrairement aux autres techniques de super-résolution, l’observation des échantillons ne nécessite pas de microscopes spécifiques, ce qui facilite fortement la mise en place de cette technique dans les laboratoires. La microscopie à expansion a été adaptée à de nombreux échantillons (des cellules, des parasites, des tissus) et a prouvé son utilité dans tous ces derniers. Un autre avantage de cette technique de microscopie réside dans le fait que, dans la majorité des cas, les anticorps utilisés classiquement en microscopie par immunofluorescence fonctionnent parfaitement avec ce type d’échantillons. Néanmoins, cette méthode présente des inconvénients, tels que la nécessité d’utiliser une quantité importante d’anticorps, ce qui peut être coûteux si la technique est utilisée en routine, ainsi que l’augmentation de l’épaisseur de l’échantillon, qui peut affecter la visualisation en profondeur de certains objets biologiques, non compatible avec la limite de distance de travail des objectifs utilisés.
Depuis son invention, la microscopie à expansion n’a cessé d’évoluer, soit dans le but d’en améliorer la résolution [7, 8], soit dans un but de simplification [9]. À ce jour, il existe deux grandes classes de protocoles [10] : le protocole « pré-expansion » et le protocole « post-expansion », selon le moment où l’immunomarquage des échantillons biologiques est réalisé (Figure 1).
 |
Figure 1. Comparaison des protocoles « pré-expansion » et « post-expansion ». Dans le protocole « pré-expansion », les cellules sont fixées puis immunomarquées avant d’être enrobées dans un polymère expansible. Après digestion totale du protéome par la protéinase K, les fluorochromes associés au polymère sont soumis à l’expansion par ajout d’eau. Dans le protocole « post-expansion », les cellules sont directement enrobées dans un polymère expansible. Après dénaturation partielle du protéome, les protéines ancrées au polymère sont soumises à l’expansion par ajout d’eau puis immunomarquées. |
Dans le protocole « pré-expansion », les échantillons biologiques sont fixés, puis marqués par des anticorps couplés à des fluorochromes, et incubés en présence d’agents chimiques permettant d’ancrer ces fluorochromes au futur polymère. Les échantillons sont ensuite enrobés dans le polymère composé, entre autres, d’acrylate de sodium. L’acrylate de sodium est l’élément responsable de l’expansion : en présence d’eau, il change de conformation, entraînant une dilatation du polymère. Après polymérisation, les échantillons sont intégralement digérés par une protéase, la protéinase K, laissant essentiellement l’empreinte des fluorochromes attachés au polymère (Figure 1). L’ajout d’eau pure permet ensuite l’expansion du polymère, ce qui espace d’autant les fluorochromes ancrés qui témoignent de la position initiale des protéines d’intérêt. Ils sont alors visualisés avec une résolution améliorée (Figure 1) [6].
Dans le deuxième protocole, l’approche de « post-expansion », l’immunomarquage des protéines s’effectue après l’expansion. Dans ce protocole, initialement appelé MAP pour magnified analysis of the proteome [11], les échantillons biologiques sont fixés puis directement incubés en présence d’agents chimiques permettant d’ancrer toutes les protéines au futur polymère. Les échantillons sont ensuite enrobés dans un mélange d’acrylamide et d’acrylate de sodium, puis le gel est polymérisé. À l’inverse de la protéolyse complète réalisée dans le protocole « pré-expansion », le protéome est conservé et dénaturé afin de perturber les interactions entre protéines sans en impacter la séquence primaire. Au cours de l’expansion du gel dans l’eau pure, la distance entre les protéines est dès lors augmentée d’un facteur quatre. Après expansion, les protéines d’intérêt sont directement marquées dans le gel avec des anticorps spécifiques (Figure 1).
Ces deux protocoles présentent chacun leurs avantages et leurs inconvénients. La méthode « pré-expansion » permet l’immunomarquage des échantillons dans leur état non dénaturé, et elle ne nécessite que peu d’anticorps. En revanche, elle s’accompagne de quatre inconvénients : la dilution volumétrique des fluorochromes dans le gel en expansion [8], l’introduction d’un linkage error, qui correspond à la distance entre le fluorochrome couplé à l’anticorps et l’épitope reconnu [10, 12], la destruction de certains fluorophores [13], et la non accessibilité aux épitopes qui sont masqués [11, 14]. Il en résulte que l’intensité globale de la fluorescence finale est souvent faible. La méthode « post-expansion », du fait de l’ajout des anticorps portant un fluorochrome après la dilution volumétrique, résulte en un signal plus intense et le linkage error diminue de façon proportionnelle au facteur d’expansion. En effet, l’échantillon biologique étant quatre fois plus large, la taille relative des anticorps et donc la distance entre fluorochrome et épitope sont réduites, améliorant ainsi la précision des observations et des mesures qui en découlent [10]. Ce protocole se révèle particulièrement intéressant pour les structures compactes dans lesquelles certains épitopes sont naturellement masqués. En effectuant l’immunomarquage après l’expansion des échantillons, de nouveaux épitopes deviennent accessibles et peuvent donc être révélés [14]. En revanche, ce protocole a le désavantage, outre le fait de consommer plus d’anticorps, de perdre les conformations tridimensionnelles de certains épitopes lors du processus de dénaturation, comme c’est le cas, par exemple, avec la technique d’analyse de protéines par électrophorèse suivie de western blot. Récemment, notre laboratoire a développé une variante du protocole MAP de « post-expansion » [11]. Cette méthode, appelée ultrastructure expansion microscopy (U-ExM) [14, 15], dans laquelle les conditions d’ancrage des protéines ont été optimisées, permet à la fois une expansion et une préservation optimales de l’organisation ultrastructurale des organites cellulaires, donnant accès à leur architecture moléculaire.
En utilisant les centrioles de l’algue verte Chlamydomonas reinhadrtii comme référence moléculaire, nous avons pu démontrer que l’organisation de cet organite cylindrique, aux dimensions stéréotypiques de 500 nm de long sur 200 nm de large, était intacte après expansion [14]. Le protocole d’U-ExM a également permis de révéler, pour la première fois, l’organisation spatiale des protéines qui composent le centriole à l’échelle nanométrique (voir plus loin) [16–21]. D’autres variantes de ce protocole existent, comme le eMAP (epitope-preserving-MAP) [22], le cMAP (centriole-MAP) [23] et le Cell-MAP [22].
Les gels d’expansion peuvent être conservés plusieurs jours à 4 °C. Mais au-delà d’une semaine, le signal de fluorescence commence à se dégrader, ce qui peut poser un problème si l’on souhaite conserver des expériences pour des études ultérieures. Les gels d’U-ExM peuvent être plongés dans du glycérol (50 % en eau distillée) et congelés à – 20 °C. Dans ces conditions, ils peuvent être gardés plusieurs mois et être de nouveau imagés ou marqués avec des anticorps après élimination du glycérol.
Cryo-fixation et microscopie à expansion
Chacune des méthodes d’imagerie (microscopie électronique, microscopie photonique, microscopie super-résolution, microscopie à expansion) nécessite la fixation préalable des échantillons biologiques. Cette fixation, fondée généralement sur l’utilisation d’aldéhydes, crée une réticulation entre les protéines qui est indispensable au maintien de l’organisation cellulaire au cours des traitements nécessaires à la microscopie photonique ou électronique. Mais elle entraîne la formation d’artéfacts et des changements morphologiques de la cellule [24]. Pour la microscopie électronique et en raison de sa haute résolution, les chercheurs ont œuvré pendant des années pour développer une méthode optimale permettant de fixer les structures subcellulaires ou macromoléculaires tout en préservant leur état natif. En 1981, Jacques Dubochet, biophysicien et universitaire suisse, prix Nobel de chimie en 2017, et son équipe, découvrent ainsi la cryo-fixation en eau vitreuse [25]. Cette méthode consiste à refroidir instantanément, à – 180 °C, les échantillons biologiques. Elle permet ainsi d’immobiliser les molécules d’eau sans laisser le temps d’une cristallisation (ce que l’on nomme l’état vitreux). Cette formation d’eau vitreuse « fige » alors l’échantillon biologique dans son état natif. Alors qu’il est possible d’observer en cryo-microscopie électronique les molécules purifiées directement en eau vitreuse, les cellules, quant à elles, doivent, la plupart du temps, être enrobées dans une résine afin d’en réaliser des sections fines. Aussi, afin de préserver et stabiliser au maximum l’échantillon dans son état le plus natif possible, les chercheurs ont développé une étape supplémentaire appelée freeze substitution [26]. Au cours de ce processus, les molécules de glace, en retournant à l’état liquide, seront instantanément substituées par de l’acétone, dont le coefficient de diffusion est supérieur à celui de l’eau. Lors du retrait des molécules d’eau, les molécules d’intérêt peuvent s’agréger et s’effondrer, comme c’est le cas lors de la fixation par réticulation chimique [24]. Afin de limiter cette agrégation, la freeze substitution est réalisée à basse température, ce qui conduit à la formation d’agrégats de taille réduite [26]. En effet, grâce à l’abaissement de la température, la viscosité intracellulaire augmente, limitant ainsi la formation de gros agrégats et préservant l’ultrastructure cellulaire au plus proche de son état natif.
En microscopie photonique, les artéfacts ultrastructuraux liés à la fixation chimique n’étaient pas jusqu’à présent un facteur limitant, la résolution de la méthode étant réduite. Depuis l’invention des méthodes de microscopie à super-résolution, il est cependant devenu important que l’architecture cellulaire soit mieux préservée. C’est dans cette optique que nous avons développé une méthode qui couple la cryo-fixation à la microscopie à expansion (appelée cryo-ExM) [27]. Sur le plan expérimental, la première étape consiste à cryo-congeler les échantillons biologiques. Il existe deux méthodes de cryo-fixation : la congélation à haute pression et le plunging. Ces deux méthodes nécessitent des équipements particuliers qui sont généralement présents dans les laboratoires ou plateformes de microscopie électronique.
Dans le plunging, les échantillons biologiques sont fixés à une lamelle de verre qui est ensuite rapidement plongée, via un bras mécanique, dans un puits d’éthane liquide refroidi à – 180 °C par de l’azote liquide, ce qui entraîne la vitrification de l’eau contenue dans les échantillons1. S’en suit l’étape de freeze substitution. Au cours de cette étape, qui ne nécessite pas d’équipements particuliers et qui est facilement réalisable dans des laboratoires de biologie, les lamelles sont transférées rapidement dans des tubes (de 5 mL) contenant de l’acétone pur (1 mL) préalablement congelé dans l’azote liquide. Les tubes sont ensuite réchauffés lentement, de – 180 °C à + 4 °C. Les molécules d’eau, en retournant à l’état liquide, sont ainsi instantanément substituées par l’acétone, dont le coefficient de diffusion est supérieur à celui de l’eau. Après une étape de réhydratation par des incubations dans des bains contenant une concentration croissante d’eau, les échantillons seront prêts pour l’expansion avec le protocole U-ExM [14]. Les lamelles sont tout d’abord incubées en présence d’un mélange de formaldéhyde et d’excès d’acrylamide : le formaldéhyde lie les groupements amines libres des protéines à l’acrylamide. L’acrylamide en excès permet de limiter la réticulation protéique normalement provoquée par le formaldéhyde et d’ancrer les protéines au futur polymère d’expansion. Ce polymère est constitué d’acrylamide/bis-acrylamide (utilisé en biochimie pour les gels d’électrophorèse) et d’acrylate de sodium (qui permet l’expansion). Afin de permettre une expansion isotropique des protéines dans le gel, les échantillons sont dénaturés, ce qui provoque une rupture des liaisons entre protéines. Sous l’effet de l’ajout de l’eau, le gel se dilate, ce qui induit l’éloignement des protéines à une distance égale à celle de l’expansion du gel. Les gels sont ensuite immunomarqués et examinés avec des microscopes à épifluorescence ou par microscopie confocale (Figure 2).
 |
Figure 2. Étapes de la cryo-microscopie à expansion. Les échantillons vivants (A) sont plongés rapidement dans de l’éthane liquide refroidi à – 180 °C (B) puis directement placés en présence d’acétone congelé et incubés en carboglace (C). Après une étape de réhydratation, les échantillons sont prêts pour l’expansion. Le protéome des échantillons est ensuite associé à un polymère extensible via des ancres moléculaires (formaldéhyde-acrylamide) (D, E). Les protéines sont dénaturées (F) et, après une expansion partielle, immunomarquées (G). L’expansion finale du gel est réalisée par ajout d’eau distillée (H). FA : formaldéhyde ; AA : acrylamide ; BIS : bisacrylamide ; SA : acrylate de sodium ; TEMED/APS : 1,2-Bis(diméthylamino)éthane/persulphate d’ammonium ; SDS : sodium dodécyl sulfate. |
« Voir c’est savoir » : application de la cryo-ExM
La microscopie à expansion a d’ores et déjà fait ses preuves à différentes échelles [6, 7, 13, 28, 29]. Au niveau tissulaire, son utilisation a, par exemple, permis de révéler avec précision l’organisation du système nerveux central en conditions saine et pathologique [30], ou de mieux comprendre le mécanisme de la rétinite pigmentaire (retinitis pigmentosa), une maladie dégénérative de la rétine [31]. À l’échelle de la cellule, l’U-ExM, en particulier, a considérablement accru nos connaissances sur l’organisation et la fonction de certains organelles, notamment des centrioles [14, 16–21, 32–36]. Comme pour le centriole, l’étude de l’organisation de certains parasites est particulièrement difficile en raison de leur très petite taille et, parfois, de l’impossibilité d’utiliser la nanoscopie. Elle repose ainsi principalement sur l’utilisation de la microscopie électronique. L’U-ExM a néanmoins permis, pour la première fois, de révéler chez Plasmodium, le parasite responsable du paludisme, la présence d’une structure, appelée apical tubulin ring, jusqu’alors uniquement observée chez d’autres parasites de la famille des apicomplexes, comme Toxoplasma gondii [37]. La microscopie à expansion a également révélé l’organisation du cytosquelette de Toxoplasma gondii et d’un autre parasite, Trypanosoma brucei, responsable de la maladie du sommeil [38, 39]. Au-delà de la puissance descriptive et quantitative apportée par la microscopie à expansion, le domaine de la parasitologie bénéficie aujourd’hui d’un nouvel outil de pointe permettant d’appréhender des questions biologiques, telles que les mécanismes sous-jacents à certaines fonctions parasitaires [38–46].
Bien que ces nombreux travaux démontrent la puissance de la microscopie à expansion pour étudier l’organisation tissulaire et cellulaire et, en particulier, pour l’étude du cytosquelette, il existe peu d’études approfondies dans le cadre de l’observation d’organelles membranaires. La préservation des membranes cellulaires requiert en effet des connaissances méthodologiques particulières, notamment pour la fixation des échantillons. Les méthodes de fixation permettant de préserver les membranes ne sont par ailleurs pas nécessairement compatibles avec l’observation des éléments du cytosquelette, ni avec le processus d’expansion lui-même. En effet, le glutaraldéhyde, l’aldéhyde nécessaire au maintien de l’ultrastructure des membranes, empêche la bonne expansion des échantillons en « post-fixation » [14], probablement à cause des ponts qui sont formés par les aldéhydes entre les protéines. Pour pallier à ces contraintes, il est nécessaire de combiner une méthode de fixation qui permet de préserver l’ultrastructure des échantillons (membrane et cytosquelette, par exemple) et qui n’altère pas l’expansion. L’application de la cryo-fixation à l’expansion remplit ces critères et permet de préserver et d’observer des structures cellulaires de différents types avec l’approche d’immunomarquage « post-expansion ». Il est ainsi possible d’observer à la fois des éléments du cytosquelette (d’actine ou de microtubules) et des organites membranaires (mitochondries, réticulum endoplasmique) dans leur état quasi-natif et avec une haute résolution. La cryo-ExM a permis de capturer des évènements dynamiques, tels que l’interaction du réticulum endoplasmique ou des mitochondries avec les microtubules, deux phénomènes transitoires et, jusqu’alors, principalement observés en vidéo-nanoscopie sur des cellules vivantes [27]. En ajoutant une très faible concentration de glutaraldéhyde et de formaldéhyde lors de la freeze substitution [47], il est possible de visualiser directement les structures membranaires en utilisant le fluorophore BODIPY (bore-dipyrrométhene) après l’étape d’expansion (Figure 3A). Ce colorant lipidique permet d’observer l’organisation fine des membranes plasmiques et endosomales, comme avec la microscopie électronique.
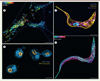 |
Figure 3. Divers exemples de l’utilisation de la cryo-ExM. A. Image confocale d’une cellule de sarcome (U2OS) dont les lipides membranaires ont été marqués en utilisant le BODIPY après cryo-fixation et expansion. Le code couleur représente la position de chaque image à travers la cellule (13 images sur 2 µm d’épaisseur). Barre d’échelle : 2,5 µm. B. Image confocale de levures (Saccaromyces cerevisiae) immunomarquées pour la détection de l’ATPase vacuolaire VPH1 (orange) et du protéome total en utilisant le NHS-ester (bleu) après cryo-fixation et expansion. Ces marquages permettent de visualiser distinctement différents organites : la vacuole (V), les mitochondries (m) et le noyau (N). Barre d’échelle : 2,5 µm. C. Image confocale du parasite Trypanosoma Brucei dont la tubuline (jaune), l’ADN (bleu) et les mitochondries (magenta) ont été immunomarqués après cryo-fixation et expansion. Ces marquages permettent de distinguer les microtubules individuels (jaune) ainsi que le réseau unique de mitochondries (magenta). Enfin, le marquage de l’ADN avec le Hoechst (bleu) met en évidence l’ADN nucléaire et l’ADN mitochondrial, dans le kinétoplaste, une structure spécifique qui se trouve à la base du flagelle, lui-même attaché le long du corps cellulaire. Barre d’échelle : 2,5 µm. D. Image confocale du parasite Trypanosoma Brucei dont le protéome total a été marqué avec le NHS-ester après cryo-fixation et expansion. Le code couleur représente la position de chaque image à travers le parasite (13 images sur 2 µm d’épaisseur). Barre d’échelle : 2,5 µm. |
La cryo-fixation permet également de contourner d’autres artéfacts induits par les fixations chimiques, tels que la délocalisation de certaines protéines dynamiques. La localisation du facteur de transcription Sox2 au niveau des chromosomes, au cours de cycle cellulaire, qui est perdue après fixation des échantillons par des aldéhydes, est en effet conservée avec la cryo-ExM, dans des cellules cryo-fixées [27].
En plus de la conservation des structures membranaires et de la localisation de certaines protéines dynamiques, la cryo-ExM préserve les organisations moléculaires de type liquid-liquid phase transition, sensibles aux fixations chimiques [27]. Nous avons montré qu’il était possible d’imager le pyrénoïde, une structure de stockage du carbone présente chez l’algue verte Chlamydomonas reinardthii. Dans cet organite, la Rubisco, une enzyme clé de la photosynthèse, s’organise par séparation de phase. Grâce à la cryo-ExM, il a été possible de visualiser cet organite avec un niveau de détail se rapprochant de celui obtenu avec la cryo-microscopie électronique [27].
Finalement, la cryo-ExM peut s’appliquer à différents modèles cellulaires pour lesquels la microscopie à fluorescence à super-résolution est très peu utilisée, comme chez le parasite Trypanosoma brucei, mais également chez la levure en ajoutant une étape de digestion enzymatique après la freeze-substitution, pour digérer la paroi cellulaire qui entrave l’expansion isotropique (Figure 3B–D) [48]. En couplant l’utilisation de différents anticorps avec le marquage d’ester de N-hydroxysuccinimide2 qui permet de visualiser l’intégralité du protéome cellulaire [29], il est possible à présent de préserver, de localiser et de visualiser distinctement les différents organites dans leur contexte cellulaire.
Conclusion
Inventée en 2015, la microscopie à expansion, grâce à sa simplicité, a démocratisé la microscopie à super-résolution. Le développement de l’ultrastructure expansion microscopy (U-ExM) et sa combinaison avec la cryo-fixation (cryo-ExM), permet désormais de visualiser l’architecture cellulaire native d’échantillons biologiques, en contournant l’utilisation autrefois nécessaire des fixateurs chimiques. Néanmoins la résolution de la microscopie à expansion reste dépendante de la capacité résolutive du microscope utilisé, rapportée au facteur d’expansion. En pratique, la résolution est de l’ordre de 70 nm, avec un facteur d’expansion de quatre [6]. Afin de d’augmenter la résolution, il est possible de coupler ces approches avec la nanoscopie. Le couplage de l’U-ExM à la nanoscopie a déjà montré sa puissance à l’échelle du centriole, pour lequel il a permis de révéler certaines caractéristiques qui n’avaient jamais été observées en microscopie photonique [14, 36]. De nouvelles approches d’expansion de 10x, 16x ou même 20x, permettent désormais d’observer des structures moléculaires avec une résolution nanométrique inédite [29, 47, 49, 50]. En couplant toutes ces approches de microscopie à expansion avec la cryo-fixation, il sera possible de visualiser la structure native des bio-molécules avec un minimum d’artéfact, et, ainsi, de repousser d’avantage les limites de la microscopie photonique.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Nous remercions le Fonds National Suisse pour la recherche (SNSF subside n° 310030_205087 attribué à Paul Guichard et Virginie Hamel) ainsi que la bourse EMBO Long Term fellowship n° 284-2019 pour le financement d’Eloïse Bertiaux. Nous remercions Mélanie Bonhivers pour avoir donné la souche sauvage de Trypanosoma brucei.
À partir de ce moment, la lamelle de verre doit être conservée à - 180 °C, en la transférant rapidement de l'éthane liquide à l'azote liquide qui entoure le puits. Si la température de la lamelle augmente, au-dessus de - 140 °C, l'état vitreux sera perdu et la structure cellulaire sera fortement affectée.
Références
- Verdaasdonk JS, Stephens AD, Haase J, et al. Bending the Rules: Widefield Microscopy and the Abbe Limit of Resolution. J Cell Physiol 2014 ; 229 : 132–138. [CrossRef] [PubMed] [Google Scholar]
- Sahl SJ, Hell SW, Jakobs S. Fluorescence nanoscopy in cell biology. Nat Rev Mol Cell Biol 2017 ; 18 : 685–701. [CrossRef] [PubMed] [Google Scholar]
- Jacquemet G, Carisey AF, Hamidi H, et al. Cell science at a glance. The cell biologist’s guide to super-resolution microscopy. J Cell Sci 2020; 133 : jcs240713. [CrossRef] [PubMed] [Google Scholar]
- Schermelleh L, Ferrand A, Huser T, et al. Super-resolution microscopy demystified. Nat Cell Biol 2019 ; 21 : 72–84. [CrossRef] [PubMed] [Google Scholar]
- Sieben C, Douglass KM, Guichard P, et al. Super-resolution microscopy to decipher multi-molecular assemblies. Curr Opin Struct Biol 2018 ; 49 : 169–176. [CrossRef] [PubMed] [Google Scholar]
- Chen F, Tillberg PW, Boyden ES. Optical imaging. Expansion microscopy. Science 2015 ; 347 : 543–548. [CrossRef] [PubMed] [Google Scholar]
- Gao R, Yu CJ, Gao L, et al. A highly homogeneous polymer composed of tetrahedron-like monomers for high-isotropy expansion microscopy. Nat Nanotechnol 2021; 16 : 698–707. [CrossRef] [PubMed] [Google Scholar]
- Damstra HG, Mohar B, Eddison M, et al. Visualizing cellular and tissue ultrastructure using Ten-fold Robust Expansion Microscopy (TREx). eLife 2022; 11. [Google Scholar]
- Chozinski TJ, Halpern AR, Okawa H, et al. Expansion microscopy with conventional antibodies and fluorescent proteins. Nat Methods 2016 ; 13 : 485–488. [CrossRef] [PubMed] [Google Scholar]
- Hamel V, Guichard P. Improving the resolution of fluorescence nanoscopy using post-expansion labeling microscopy. Methods Cell Biol 2021; 161 : 297–315. [CrossRef] [PubMed] [Google Scholar]
- Ku T, Swaney J, Park JY, et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nat Biotechnol 2016 ; 34 : 973–981. [CrossRef] [PubMed] [Google Scholar]
- Zwettler FU, Reinhard S, Gambarotto D, et al. Molecular resolution imaging by post-labeling expansion single-molecule localization microscopy (Ex-SMLM). Nat Commun 2020; 11 : 3388. [CrossRef] [PubMed] [Google Scholar]
- Wassie AT, Zhao Y, Boyden ES. Expansion microscopy: principles and uses in biological research. Nat Methods 2019 ; 16 : 33–41. [CrossRef] [PubMed] [Google Scholar]
- Gambarotto D, Zwettler FU, Le Guennec M, et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nat Methods 2019 ; 16 : 71–74. [CrossRef] [PubMed] [Google Scholar]
- Gambarotto D, Hamel V, Guichard P. Ultrastructure expansion microscopy (U-ExM). Methods Cell Biol 2021; 161 : 57–81. [CrossRef] [PubMed] [Google Scholar]
- Steib E, Laporte MH, Gambarotto D, et al. Wdr90 is a centriolar microtubule wall protein important for centriole architecture integrity. eLife 2020; 9 : 1–28. [CrossRef] [Google Scholar]
- Le Guennec M, Klena N, Gambarotto D, et al. A helical inner scaffold provides a structural basis for centriole cohesion. Sci Adv 2020; 6 : eaaz4137. [CrossRef] [PubMed] [Google Scholar]
- Laporte MH, Bouhlel IB, Bertiaux E, et al. Human SFI1 and Centrin form a complex critical for centriole architecture and ciliogenesis. EMBO J 2022; 41 : e112107. [CrossRef] [PubMed] [Google Scholar]
- Karasu OR, Neuner A, Atorino ES, et al. The central scaffold protein CEP350 coordinates centriole length, stability, and maturation. J Cell Biol 2022; 221 : e202203081. [CrossRef] [Google Scholar]
- van den Hoek H, Klena N, Jordan MA, et al. In situ architecture of the ciliary base reveals the stepwise assembly of intraflagellar transport trains. Science 2022; 377 : 543–8. [CrossRef] [PubMed] [Google Scholar]
- Le Borgne P, Greibill L, Laporte MH, et al. The evolutionary conserved proteins CEP90, FOPNL, and OFD1 recruit centriolar distal appendage proteins to initiate their assembly. PLOS Biol 2022; 20 : e3001782. [CrossRef] [PubMed] [Google Scholar]
- Park J, Khan S, Yun DH, et al. Epitope-preserving magnified analysis of proteome (eMAP). Sci Adv 2021; 7 : eabf6589. [CrossRef] [PubMed] [Google Scholar]
- Sahabandu N, Kong D, Magidson V, et al. Expansion microscopy for the analysis of centrioles and cilia. J. Microsc. 2019 ; 276 : 145–159. [CrossRef] [PubMed] [Google Scholar]
- Kellenberger E, Johansen R, Maeder M, et al. Artefacts and morphological changes during chemical fixation. J Microsc 1992 ; 168 : 181–201. [CrossRef] [PubMed] [Google Scholar]
- Dubochet J, McDowall AW. Vitrification of pure water for electron microscopy. J Microsc 1981; 124 : RP3–RP4. [CrossRef] [Google Scholar]
- Kellenberger E. The potential of cryofixation and freeze substitution: observations and theoretical considerations. J Microsc 1991 ; 161 : 183–203. [CrossRef] [PubMed] [Google Scholar]
- Laporte MH, Klena N, Hamel V, et al. Visualizing the native cellular organization by coupling cryofixation with expansion microscopy (Cryo-ExM). Nat Methods 2022; 19 : 216–22. [CrossRef] [PubMed] [Google Scholar]
- Yu C-C (Jay), Barry NC, Wassie AT, et al. Expansion microscopy of C. elegans. eLife 2020; 9. [Google Scholar]
- M’Saad O, Bewersdorf J. Light microscopy of proteins in their ultrastructural context. Nat Commun 2020; 11 : 3850. [CrossRef] [PubMed] [Google Scholar]
- Sarkar D, Kang J, Wassie AT, et al. Revealing nanostructures in brain tissue via protein decrowding by iterative expansion microscopy. Nat Biomed Eng 2022; 6 : 1057–73. [CrossRef] [PubMed] [Google Scholar]
- Mercey O, Kostic C, Bertiaux E, et al. The connecting cilium inner scaffold provides a structural foundation that protects against retinal degeneration. PLOS Biol 2022; 20 : e3001649. [CrossRef] [PubMed] [Google Scholar]
- Mahecic D, Gambarotto D, Douglass KM, et al. Homogeneous multifocal excitation for high-throughput super-resolution imaging. Nat Methods 2020; 17 : 726–33. [CrossRef] [PubMed] [Google Scholar]
- Vásquez-Limeta A, Lukasik K, Kong D, et al. CPAP insufficiency leads to incomplete centrioles that duplicate but fragment. J Cell Biol 2022; 221 : e202108018. [CrossRef] [PubMed] [Google Scholar]
- Kong D, Sahabandu N, Sullenberger C, et al. Prolonged mitosis results in structurally aberrant and over-elongated centrioles. J Cell Biol 2020; 219 : e201910019. [CrossRef] [PubMed] [Google Scholar]
- Mahen R. cNap1 bridges centriole contact sites to maintain centrosome cohesion. PLOS Biol 2022; 20 : e3001854. [CrossRef] [PubMed] [Google Scholar]
- Woglar A, Pierron M, Schneider FZ, et al. Molecular architecture of the C. elegans centriole. PLOS Biol 2022; 20 : e3001784. [CrossRef] [PubMed] [Google Scholar]
- Bertiaux E, Balestra AC, Bournonville L, et al. Expansion microscopy provides new insights into the cytoskeleton of malaria parasites including the conservation of a conoid. PLOS Biol 2021; 19 : e3001020. [CrossRef] [PubMed] [Google Scholar]
- Tosetti N, Dos Santos Pacheco N, Bertiaux E, et al. Essential function of the alveolin network in the subpellicular microtubules and conoid assembly in Toxoplasma gondii. eLife 2020; 9. [CrossRef] [PubMed] [Google Scholar]
- Amodeo S, Kalichava A, Fradera-Sola A, et al. Characterization of the novel mitochondrial genome segregation factor TAP110 in Trypanosoma brucei. J Cell Sci 2021; 134 : jcs254300. [CrossRef] [PubMed] [Google Scholar]
- Dos Santos Pacheco N, Tosetti N, Krishnan A, et al. Revisiting the Role of Toxoplasma gondii ERK7 in the Maintenance and Stability of the Apical Complex. mBio 2021; 12 : e0205721. [CrossRef] [PubMed] [Google Scholar]
- Liffner B, Absalon S. Expansion Microscopy Reveals Plasmodium falciparum Blood-Stage Parasites Undergo Anaphase with A Chromatin Bridge in the Absence of Mini-Chromosome Maintenance Complex Binding Protein. Microorganisms 2021; 9 : 2306. [CrossRef] [PubMed] [Google Scholar]
- Lentini G, Ben Chaabene R, Vadas O, et al. Structural insights into an atypical secretory pathway kinase crucial for Toxoplasma gondii invasion. Nat Commun 2021; 12 : 3788. [CrossRef] [PubMed] [Google Scholar]
- Alonso VL. Ultrastructure Expansion Microscopy (U-ExM) in Trypanosoma cruzi: localization of tubulin isoforms and isotypes. Parasitol Res 2022; 121 : 3019–24. [CrossRef] [PubMed] [Google Scholar]
- Vigetti L, Labouré T, Roumégous C, et al. The BCC7 Protein Contributes to the Toxoplasma Basal Pole by Interfacing between the MyoC Motor and the IMC Membrane Network. Int J Mol Sci 2022; 23 : 5995. [CrossRef] [PubMed] [Google Scholar]
- Pavlou G, Touquet B, Vigetti L, et al. Coupling Polar Adhesion with Traction, Spring, and Torque Forces Allows High-Speed Helical Migration of the Protozoan Parasite Toxoplasma. ACS Nano 2020; 14 : 7121–39. [CrossRef] [PubMed] [Google Scholar]
- Tomasina R, Gonzalez FC, Martins-Duarte ÉS, et al. Separate To Operate: the Centriole-Free Inner Core of the Centrosome Regulates the Assembly of the Intranuclear Spindle in Toxoplasma gondii. mBio 2022; 13 : e0185922. [CrossRef] [PubMed] [Google Scholar]
- Louvel V, Haase R, Mercey O, et al. Nanoscopy of organelles and tissues with iterative ultrastructure expansion microscopy (iU-ExM). bioRxiv 2022; 2022.11.14.516383. [Google Scholar]
- Hinterndorfer K, Laporte MH, Mikus F, et al. Ultrastructure Expansion Microscopy reveals the nanoscale cellular architecture of budding and fission yeast. J Cell Sci 2022; 135 : jcs260240. [CrossRef] [PubMed] [Google Scholar]
- Damstra HG, Mohar B, Eddison M, et al. Visualizing cellular and tissue ultrastructure using Ten-fold Robust Expansion Microscopy (TREx). eLife 2022; 11. [Google Scholar]
- Chang JB, Chen F, Yoon YG, et al. Iterative expansion microscopy. Nat Methods 2017; 14 : 593–9. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Comparaison des protocoles « pré-expansion » et « post-expansion ». Dans le protocole « pré-expansion », les cellules sont fixées puis immunomarquées avant d’être enrobées dans un polymère expansible. Après digestion totale du protéome par la protéinase K, les fluorochromes associés au polymère sont soumis à l’expansion par ajout d’eau. Dans le protocole « post-expansion », les cellules sont directement enrobées dans un polymère expansible. Après dénaturation partielle du protéome, les protéines ancrées au polymère sont soumises à l’expansion par ajout d’eau puis immunomarquées. |
| Dans le texte | |
 |
Figure 2. Étapes de la cryo-microscopie à expansion. Les échantillons vivants (A) sont plongés rapidement dans de l’éthane liquide refroidi à – 180 °C (B) puis directement placés en présence d’acétone congelé et incubés en carboglace (C). Après une étape de réhydratation, les échantillons sont prêts pour l’expansion. Le protéome des échantillons est ensuite associé à un polymère extensible via des ancres moléculaires (formaldéhyde-acrylamide) (D, E). Les protéines sont dénaturées (F) et, après une expansion partielle, immunomarquées (G). L’expansion finale du gel est réalisée par ajout d’eau distillée (H). FA : formaldéhyde ; AA : acrylamide ; BIS : bisacrylamide ; SA : acrylate de sodium ; TEMED/APS : 1,2-Bis(diméthylamino)éthane/persulphate d’ammonium ; SDS : sodium dodécyl sulfate. |
| Dans le texte | |
 |
Figure 3. Divers exemples de l’utilisation de la cryo-ExM. A. Image confocale d’une cellule de sarcome (U2OS) dont les lipides membranaires ont été marqués en utilisant le BODIPY après cryo-fixation et expansion. Le code couleur représente la position de chaque image à travers la cellule (13 images sur 2 µm d’épaisseur). Barre d’échelle : 2,5 µm. B. Image confocale de levures (Saccaromyces cerevisiae) immunomarquées pour la détection de l’ATPase vacuolaire VPH1 (orange) et du protéome total en utilisant le NHS-ester (bleu) après cryo-fixation et expansion. Ces marquages permettent de visualiser distinctement différents organites : la vacuole (V), les mitochondries (m) et le noyau (N). Barre d’échelle : 2,5 µm. C. Image confocale du parasite Trypanosoma Brucei dont la tubuline (jaune), l’ADN (bleu) et les mitochondries (magenta) ont été immunomarqués après cryo-fixation et expansion. Ces marquages permettent de distinguer les microtubules individuels (jaune) ainsi que le réseau unique de mitochondries (magenta). Enfin, le marquage de l’ADN avec le Hoechst (bleu) met en évidence l’ADN nucléaire et l’ADN mitochondrial, dans le kinétoplaste, une structure spécifique qui se trouve à la base du flagelle, lui-même attaché le long du corps cellulaire. Barre d’échelle : 2,5 µm. D. Image confocale du parasite Trypanosoma Brucei dont le protéome total a été marqué avec le NHS-ester après cryo-fixation et expansion. Le code couleur représente la position de chaque image à travers le parasite (13 images sur 2 µm d’épaisseur). Barre d’échelle : 2,5 µm. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




