| Issue |
Med Sci (Paris)
Volume 38, Number 5, Mai 2022
Nos jeunes pousses ont du talent !
|
|
|---|---|---|
| Page(s) | 487 - 492 | |
| Section | Partenariat médecine/sciences - Écoles doctorales - Masters | |
| DOI | https://doi.org/10.1051/medsci/2022059 | |
| Published online | 24 May 2022 | |
Du microbiote humain à l’antibiotique de demain
The human gut microbiota to find new antibiotics
1
Master 2 Microbiologie intégrative et fondamentale, Aix Marseille Université, Marseille, France
2
Aix Marseille Université, CNRS, LCB UMR, 7283, IMM, Marseille, France
a camille.herrou@etu.univ-amu.fr
b jeremy.mom@etu.univ-amu.fr
c ihsane.nasri@etu.univ-amu.fr
d elsa.turrini@etu.univ-amu.fr
e aussel@imm.cnrs.fr
Dans le cadre de l’unité d’enseignement « Rédiger en sciences » proposée par Aix-Marseille Université, les étudiants du Master 2 Microbiologie Intégrative et Fondamentale (MIF) - en partenariat avec l’Institut de Microbiologie, Bioénergies et Biotechnologie (IM2B) - ont été confrontés aux exigences de l’écriture scientifique. Trois thématiques leur ont été proposées en relation étroite avec les laboratoires de recherche du tissu local : les bactériophages, la biologie des mycobactéries et les bactériocines. Les étudiants ont ainsi rédigé une nouvelle soulignant les résultats majeurs et l’originalité des articles étudiés. Complétée par un entretien avec leurs auteurs, l’ensemble offre un éclairage original sur la compréhension du vivant dans le domaine de la microbiologie et de la santé.
© 2022 médecine/sciences – Inserm
L’actualité scientifique vue par les étudiants du Master 2 Microbiologie d’Aix-Marseille Université (AMU), Parcours Microbiologie Intégrative et Fondamentale (MIF), Unité d’Enseignement « Rédiger en sciences »

Responsable de l’Unité d’Enseignement
Laurent Aussel
Équipe pédagogique
Laurent Aussel - Professeur, Aix-Marseille Université - aussel@imm.cnrs.fr - Amel Latifi - Professeur, Aix-Marseille Université - latifi@imm.cnrs.fr
Site web https://sciences.univ-amu.fr/fr/formation/masters/master-microbiologie
Série coordonnée par Sophie Sibéril.
Le microbiote intestinal est le lieu où coexistent et évoluent près de 100 000 milliards de microorganismes dont 800 à 1 000 espèces bactériennes différentes, mais également des archées1, des eucaryotes2 et des virus. Chez les mammifères, il assure des fonctions nutritionnelles, détoxifiantes et de support au développement du système immunitaire, essentielles au maintien de l’homéostasie intestinale. Ces fonctions lui confèrent une importance comparable à celle d’un organe.
Apparu comme « l’empreinte digitale » de chaque individu, le microbiote intestinal est cependant composé d’espèces bactériennes dominantes communes à tous les êtres humains et appartenant à deux phylums majoritaires : les bacteroidetes et les firmicutes [1]. Parmi les firmicutes, les ruminocoques sont très étudiés car certaines souches possèdent l’avantage d’être cultivables en laboratoire, contrairement à la majorité des bactéries du microbiote. Les ruminocoques sont retrouvés chez 90 % des individus sains et sont connus pour leur intérêt nutritionnel et protecteur [2]. De plus, la mise en évidence d’une activité antibactérienne de certaines souches de cette famille contre des bactéries pathogènes suscite un fort intérêt dans le domaine de la recherche clinique, dans un contexte où l’émergence des résistances aux antibiotiques constitue un problème de santé publique majeur. Ainsi, les composés à l’origine de cette activité antibactérienne pourraient s’ajouter à d’autres alternatives aux antibiotiques comme la phagothérapie. Cette nouvelle reprend quelques-uns des travaux qui ont permis la caractérisation de ces molécules chez Ruminococcus gnavus.
Découverte d’une activité antibactérienne chez R. gnavus E1
L’omniprésence de R. gnavus dans la majorité des microbiomes humains en fait un microorganisme d’intérêt dans la recherche clinique. La souche E1 de R. gnavus, qui peut être isolée à partir de fèces humaines, établit une relation de symbiose avec le tube digestif. Du côté de l’hôte, la bactérie présente un intérêt nutritionnel via la production d’enzymes essentielles au métabolisme alimentaire [2]. Elle possède également un intérêt protecteur et assure la maturation du mucus intestinal en dégradant les glycoprotéines qui le composent. Du côté de la bactérie, la dégradation des glycoprotéines et éléments nutritifs présents dans l’intestin de l’hôte favorise sa capacité à coloniser le milieu.
Par ailleurs, dans les années 1990, une activité anti-Clostridium perfringens (anti-Cp) fut détectée dans les fèces de rats mono-colonisés avec R. gnavus E1 [3]. Étonnament, cette activité était absente lorsque ces mêmes bactéries étaient cultivées seules in vitro. L’ajout de trypsine pancréatique dans le milieu de culture permit de rétablir cette activité antibactérienne, suggérant une relation hôte-symbionte (Figure 1). Pour appuyer ce résultat, des expériences in vivo utilisant des rats mono-colonisés avec la souche E1 en présence de trypsine exogène ont alors été réalisées. Celles-ci ont permis de confirmer la nécessité de la trypsine pour l’activité antibactérienne de R. gnavus E1 contre C. perfringens [3]. Les molécules responsables de cette activité anti-Clostridium ont par la suite été purifiées et caractérisées.
Caractérisation des ruminococcines
En effet, les progrès en matière de séquençage, de clonage et de purification de protéines ont permis l’identification et la caractérisation des molécules à l’origine de cette activité antibactérienne. Il s’agit de bactériocines, peptides antimicrobiens ayant la particularité d’être synthétisés par voie ribosomique et non par des complexes multienzymatiques.
La ruminococcine A (RumA) est la première des bactériocines sécrétées par R. gnavus E1 à avoir été mise en évidence, une dizaine d’années après la découverte de l’activité anti-Cp [4]. RumA a été purifiée à partir de cultures in vitro de R. gnavus E1 par des techniques de chromatographie visant à séparer les constituants d’un mélange en fonction de leur polarité. La séquence peptidique de RumA a été caractérisée, permettant l’identification d’acides aminés Dhb (déhydrobutyrine) et Lan (lanthionine), caractéristiques de la classe des lantibiotiques3. Ces derniers, avec les labyrinthopeptines4 et les sactipeptides5, constituent la classe I des bactériocines ayant pour cible principale les bactéries à Gram positif. Plus particulièrement, RumA possède une activité antibactérienne contre les souches phylogénétiquement proches de R. gnavus et également contre différentes espèces du genre Clostridium. Elle ne présente, en revanche, aucune activité contre des bactéries à Gram négatif [4]. Cette activité est dépendante de la trypsine. Un modèle de régulation propose qu’un peptide inducteur sécrété par R. gnavus soit clivé par la trypsine pour être activé. Ce peptide inducteur active alors une cascade de régulation menant à l’activation de la transcription du gène rumA. [5] (Figure 1). Il a ensuite fallu attendre plus de dix ans et le développement de la production hétérologue (production chez Escherichia coli) de RumA pour analyser précisément sa structure et montrer la présence dans le peptide de trois ponts thioéther caractéristiques des ruminococcines et essentiels à son bon repliement [6].
 |
Figure 1. Modèle de synthèse et de maturation de RumA et RumC. Les ruminococcines RumA et RumC sont synthétisées et subissent une maturation selon deux mécanismes distincts. Les différentes étapes de synthèse et de maturation de RumA (en rouge) et RumC (en bleu) sont indiquées par des numéros. La transcription de rumA est activée par un peptide inducteur (PI) après clivage de ce dernier par la trypsine présente dans le milieu extracellulaire (produite par l’hôte). RumC est produite dans la cellule sous forme inactive, puis subit une première étape de clivage du peptide signal par RumPc. Une fois sécrétés, les 5 résidus en N-terminal de RumC sont clivés par la trypsine présente dans le milieu extracellulaire, ce qui produit la forme active du sactipeptide RumC (figure créée avec BioRender). |
La caractérisation de RumA et la mise en évidence de son rôle de peptide antimicrobien, associées aux progrès des techniques de purification, ont facilité la caractérisation d’autres bactériocines produites par R. gnavus E1. En effet, des études génomiques ont montré que le contenu des fèces provenant de rats mono-colonisés par R. gnavus E1 présentait une activité anti-C. perfringens directement corrélée à une forte expression du régulon rumC 6. La ruminococcine C (RumC) étant produite uniquement in vivo, dans l’intestin de l’hôte, il a fallu une dizaine d’années pour déterminer les conditions de production de cette bactériocine et procéder à sa purification à partir de contenus cæcaux humains : après plusieurs étapes de chromatographie, les fractions possédant une activité anti-Cp ont été analysées par spectrométrie de masse, révélant la présence de cinq isoformes de RumC possédant toutes quatre ponts thioéther, caractéristiques du groupe des sactipetides [7, 8].
Par la suite, afin de mieux caractériser le processus de biosynthèse ainsi que la structure de l’une de ces isoformes, RumC1, un système d’expression hétérologue chez Escherichia coli, a été développé. La mutagenèse dirigée de RumC1, ainsi que l’utilisation de la chromatographie en phase liquide associée à la spectrométrie de masse, ont permis d’identifier les acides aminés impliqués dans la formation des ponts thioéther : Ala12, Asn16, Arg34, et Lys42 (Figures 1, 2) [8, 9]. L’obtention de la forme mature de RumC1, contenant quatre ponts thioéther essentiels à l’activité antibactérienne, dépend de l’activité d’une SAM-sactisynthase7, RumMc1. Cette enzyme permet la formation d’une structure en double épingle à cheveux particulière à ce sactipeptide (Figure 2). De plus, la maturation de RumC1 conduisant à la production de la forme active implique deux clivages protéolytiques successifs : un premier clivage du peptide signal par la métallopeptidase RumPc, puis un second clivage des cinq résidus en N-terminal par la trypsine présente dans le milieu extracellulaire (Figure 1). Ce dernier clivage est indispensable à l’activité anti-Cp de RumC1 [8].
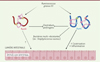 |
Figure 2. Relations entre Ruminococcus gnavus E1 et l’hôte dans le cadre de la production de ruminococcines. Ruminococcus gnavus E1 produit et sécrète plusieurs bactériocines, dont RumA et RumC. Ces deux peptides subissent des étapes de maturation intracellulaires qui permettent de former des ponts thioéther dans leur structure tridimensionnelle. Les résidus cystéine, donneurs de soufre pour les liaisons thioéther, sont représentés en rose. Les autres résidus impliqués dans la formation des liaisons covalentes au sein des formes matures de RumA et RumC sont indiqués en couleur. Ces deux bactériocines possèdent une activité anti-bactérienne contre différentes espèces dont certaines bactéries multi-résistantes aux antibiotiques. Les mécanismes qui restent à démontrer sont représentés par les flèches en traits pointillés (figure créée avec BioRender). |
Malgré un mécanisme d’action encore mal compris, l’analyse du spectre d’activité de RumC1 a permis de mettre en lumière une efficacité contre les espèces pathogènes du genre Clostridum et plus particulièrement Clostridium perfringens, mais aussi contre d’autres bactéries Gram positives telles que Staphylococcus aureus, bactérie multirésistante aux antibiotiques [8] (Figure 2). Il a également été montré in vitro que RumC1 pouvait générer une activité antifongique contre certains Basidiomycètes [10]. Par ailleurs, des tests de cytotoxicité sur des cellules intestinales et des explants iléocæcaux humains ont montré que RumC1 n’affectait pas la viabilité des cellules et n’induisait pas d’hémolyse des érythrocytes.
Ainsi, l’activité antimicrobienne exercée par RumC1 et l’absence d’effet néfaste sur les cellules eucaryotes font de ce sactipeptide un candidat intéressant pour une utilisation en tant qu’agent thérapeutique. En effet, RumC1, naturellement produit au sein du microbiote humain, pourrait constituer une alternative aux antibiotiques utilisés pour traiter les infections gastro-intestinales. La production de la protéine RumC1 recombinante fonctionnelle en laboratoire constitue l’un des enjeux importants pour une future production industrielle de cette bactériocine. Cependant, ce projet pourrait être confronté à de nombreuses limites liées notamment aux coûts de production de ce composé qui sont à ce jour très élevés par rapport aux antibiotiques disponibles sur le marché. Néanmoins, la simplification du processus de maturation de ce peptide, via la production de mimétiques de synthèse par exemple (voir entretien avec Michael Lafond), pourrait faciliter le développement de RumC1 comme nouvel antibiotique issu du microbiote intestinal.
Les ruminococcines, de nouveaux agents thérapeutiques
Trente années d’étude des ruminococcines, bactériocines produites par R. gnavus, ont permis la découverte de leur activité anti-Cp et leur caractérisation structurale et fonctionnelle. Ces connaissances ont été permises grâce à des avancées techniques considérables, notamment en matière de production et d’expression hétérologue des protéines, mais également en matière de purification et de résolution structurale. Ces bactériocines possèdent une activité antibactérienne contre des bactéries pathogènes à Gram positif, dont certaines sont résistantes aux antibiotiques telles que Staphylococcus aureus. De plus, les premiers travaux utilisant des cultures bactériennes de C. perfringens exposées de façon itérative à ces ruminococcines n’ont, jusqu’ici, pas mis en évidence de mécanismes de résistance qui seraient développés par les bactéries cibles. L’ensemble de ces données font des ruminococcines des candidats prometteurs pour le développement de nouveaux antibiotiques. En outre, au-delà de l’activité antibactérienne et l’intérêt des ruminococcines en tant qu’antibiotique, ces peptides pourraient participer de manière bénéfique aux interactions hôte-microbiote. En effet, il a été montré que RumC1 jouait un rôle multifonctionnel dans un contexte commensal (Figure 1), agissant aussi bien comme une molécule favorisant la régénération des tissus que comme un anti-inflammatoire [10]. Ce peptide joue également le rôle d’inhibiteur de la formation des biofilms, un mode de croissance qui favorise l’apparition d’infections bactériennes. Ces effets multifonctionnels de RumC1 sur l’hôte soulignent l’importance des ruminococcines et suggèrent une multitude d’applications cliniques possibles pour cette famille de composés.
Entretien avec Mickaël Lafond

Mickaël Lafond est maître de conférences à l’Université d’Aix-Marseille au sein de l’Institut des Sciences Moléculaires de Marseille (iSm2) dans l’équipe BiosCiences. Après un doctorat en Biochimie et Nutrition à l’Université Paul Cézanne (Aix-Marseille III), il a consacré l’essentiel de sa carrière à l’étude du microbiote. Il est actuellement responsable scientifique d’un projet de recherche qui porte sur le développement des nouvelles alternatives aux antibiotiques : les bactériocines de la classe des sactipeptides, plus précisément les ruminococcines.
Comment avez-vous découvert la thématique des bactériocines et pour quelle raison avez-vous décidé de travailler sur ce sujet ?
J’ai découvert la thématique des bactériocines durant ma thèse. A l’époque, Michel Fons s’intéressait à cette thématique et alors que le projet avait été mis en stand-by, j’ai repris ce sujet qui m’intéressait et j’ai relancé la machine en collaboration avec des partenaires à Grenoble notamment.
Quel est selon vous le plus des bactériocines par rapport aux autres alternatives aux antibiotiques ?
Je ne suis pas sûr qu’on puisse classer les bactériocines devant une autre stratégie alternative aux antibiotiques puisqu’il y en a un certain nombre qui sont explorées à l’heure actuelle, dont la phagothérapie. Cependant, l’avantage des bactériocines, c’est qu’elles sont produites généralement par des bactéries qui sont commensales de l’homme, ce qui en fait l’intérêt. On s’attend ainsi à une absence d’effets indésirables lors d'une utilisation thérapeutique.
Pourquoi avoir choisi de travailler sur RumC et pas de continuer les travaux sur RumA qui semble aussi posséder des propriétés antimicrobiennes intéressantes ?
Pour être honnête, la ruminococcine A, à l’époque où j’ai redémarré le projet, avait été relativement bien caractérisée. Elle avait été prédite comme appartenant à la famille des lantibiotiques de classe II, qui était une famille relativement bien connue. À l’inverse, la ruminococcine C n’était pas du tout connue, offrant un fort potentiel de découverte. Néanmoins, il reste une caractérisation fonctionnelle poussée à réaliser sur la ruminococcine A puisque les travaux qui ont été développés par des collaborateurs allemands ont porté essentiellement sur la résolution structurale de la bactériocine, pas forcément sur ses caractéristiques fonctionnelles.
Les ruminococcines possédant toutes une activité anti-Clostridia, quel est selon vous l’intérêt d’une telle redondance fonctionnelle chez Ruminococcus gnavus ?
La caractérisation fonctionnelle de la ruminococcine A n’a pas été très détaillée à ce jour et le spectre d’activité de cette ruminococcine n’a pas été complètement défini. On ne sait donc pas actuellement si les ruminoccoccines A et C ont le même spectre d’activité. De ce fait, on ne peut pas réellement affirmer qu’il y ait une vraie redondance. En revanche, on sait que les ruminococcines C sont uniquement produites in vivo, ce qui n’a jamais été montré pour la ruminococcine A.
Plusieurs études montrent un lien entre la présence abondante de R. gnavus dans le microbiote et la survenue de certaines maladies inflammatoires. Pensez-vous que cela puisse constituer un obstacle au développement des ruminococcines comme agent thérapeutique ?
La ruminococcine est un peptide antimicrobien. Il n’y a donc pas de lien de cause à effet direct entre la ruminococcine et les maladies inflammatoires chroniques de l’intestin. Le fait que R. gnavus soit surreprésenté dans le cadre de ce type de maladies inflammatoires intestinales peut être simplement lié au fait que durant l’inflammation, il y a des niches écologiques qui se libèrent. Cela laisse la place à d’autres commensaux ou symbiontes tels que R. gnavus. Donc, nous n’avons pas de preuve à l’heure actuelle d’une implication de R. gnavus dans ce type de maladies.
Les travaux portant sur le microbiote intestinal sont en plein essor mais peu de gens connaissent réellement ce sujet et son intérêt. Cela n’a pas été un frein pour vous au départ ?
Absolument pas car dès que j’ai débuté ma carrière dans la recherche, j’ai suivi le développement des travaux sur le microbiote, notamment les travaux de Joel Doré et de Jeffrey Gordon. J’ai également suivi les travaux de Michel Fons à l’époque où il venait de rejoindre le laboratoire, puisqu’il travaillait depuis des années sur cet aspect-là. C’est donc quelque chose qui m’a intéressé dès le départ. Par ailleurs, j’ai travaillé sur des aspects bénéfiques du microbiote, notamment sur la dégradation de la biomasse végétale par des bactéries du microbiote intestinal. Donc non, cela n’a pas été un frein au départ car j’avais un minimum de culture sur ce microbiote et l’intérêt qu’il peut apporter à l’hôte.
L’évolution des techniques utilisées dans l’étude des bactériocines fait apparaitre un aspect pluridisciplinaire (génomique, biologie moléculaire, biochimie et même biophysique). Pensez-vous que le microbiologiste d’aujourd’hui doit maitriser un panel de techniques plus vaste que le microbiologiste d’hier ?
Complétement ! Il est vrai qu’il y a 15-20 ans les chercheurs étaient essentiellement spécialisés dans l’étude d’une famille de protéines ou même une voie métabolique, donc quelque chose de bien précis. À l’heure actuelle, avec le développement du « haut-débit », nous sommes forcément contraints d’augmenter notre spectre de compétences techniques. De plus, je pense qu’aujourd’hui, nous sommes amenés à développer plus de collaborations pour mener à bien un projet. Les projets sont par ailleurs plus ambitieux et sont programmés pour des durées plus courtes, car ils sont financés sur des périodes de 3 à 5 ans. Généralement, les organismes financeurs nous demandent justement d’avoir des projets qui soient ambitieux et pour cela, il faut couvrir un maximum de techniques. Bien évidemment, on ne peut pas tout couvrir. Il faut donc établir des collaborations avec des experts du domaine.
Sans rentrer dans les détails confidentiels, en quoi consiste la suite de vos travaux sur RumC1 ?
Au départ du projet, nous n’aurions jamais imaginé que les ruminococcines C puissent avoir un intérêt thérapeutique. Aujourd’hui, nous en sommes conscients. La suite du projet serait de décrire le mécanisme d’action de la ruminococcine C afin de répondre aux prérequis pour rentrer en phase clinique. Cela est indispensable pour des questions de maitrise de la pharmacologie et de la pharmacocinétique. C’est ce qui est demandé : comprendre comment fonctionne une molécule pour maitriser ces aspects-là. Nous avons débuté cela il y a plus d’un an en collaboration avec le LMGM (Laboratoire de microbiologie et génétique moléculaire) de Toulouse.
Un second aspect, qui fait aussi référence au développement clinique, c’est l’aspect industriel. En ce qui concerne la production de RumC, c’est un peptide qui est modifié, qui subit une maturation, et qui a donc un coût élevé de production à l’heure actuelle. Pour rejoindre une échelle industrielle, il va falloir optimiser cette production. Cela peut notamment passer par la production de mimétiques de synthèse de RumC et c’est ce que nous essayons de faire. Cela peut aussi passer par la recherche de RumC-like naturels, qui soient plus simples à produire et donc moins coûteux.
À l’avenir, si RumC1 est utilisé comme agent thérapeutique, seriez-vous prêt à travailler avec l’industrie pharmaceutique pour produire l’antibiotique de demain ?
Notre cœur de métier, c’est le laboratoire et je ne suis pas spécialisé dans le développement de process industriels. En revanche, l’idée serait de passer le relais à un industriel plutôt que d’essayer de travailler avec ce dernier. À l’heure actuelle, on a besoin de projets de recherche purement fondamentale pour comprendre les mécanismes moléculaires et cellulaires afin de répondre à des maladies. Quand on monte un projet de recherche de ce type, il devient évident de viser une application. Nous, nous n’en sommes pas loin, mais il nous reste du chemin à parcourir.
Si vous deviez changer de thématique de recherche vers laquelle vous orienteriez-vous ?
Pourquoi changer de thématique de recherche quand on répond à une thématique de recherche prioritaire à l’échelle mondiale ! De mon point de vue, je trouve cette thématique plaisante et intéressante, notamment ce qui concerne les objectifs thérapeutiques pour l’homme. Je vous avoue ne m’être jamais posé la question.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Mariat D, Firmesse O, Levenez F, et al. The Firmicutes/Bacteroidetes ratio of the human microbiota changes with age. BMC microbiol 2009 ; 9 : 123. [CrossRef] [PubMed] [Google Scholar]
- Lafond M, Tauzin A, Bruel L, et al. α-Galactosidase and Sucrose-Kinase Relationships in a Bi-functional AgaSK Enzyme Produced by the Human Gut Symbiont Ruminococcus gnavus E1. Front Microbiol 2020; 11 : 579521. [CrossRef] [PubMed] [Google Scholar]
- Ramare F, Nicoli J, Dabard J, et al. Trypsin-dependent production of an antibacterial substance by a human Peptostreptococcus strain in gnotobiotic rats and in vitro. Appl Environ Microbiol 1993 ; 59 : 2876–2883. [CrossRef] [PubMed] [Google Scholar]
- Dabard J, Bridonneau C, Phillipe C, et al. Ruminococcin A, a new lantibiotic produced by a Ruminococcus gnavus strain isolated from human feces. Appl environ microbiol 2001 ; 67 : 4111–4118. [CrossRef] [PubMed] [Google Scholar]
- Gomez A, Ladiré M, Marcille F, et al. Trypsin mediates growth phase-dependent transcriptional regulation of genes involved in biosynthesis of ruminococcin A, a lantibiotic produced by a Ruminococcus gnavus strain from a human intestinal microbiota. J Bacteriol 2002 ; 184 : 18–28. [CrossRef] [PubMed] [Google Scholar]
- Ongey E.L., Giessmann R.T., Fons M, et al. Heterologous biosynthesis, modifications and structural characterization of Ruminococcin-A, a lanthipeptide from the gut bacterium Ruminococcus gnavus E1, in Escherichia coli. Front Microbiol 2018 ; 9 : 1688. [CrossRef] [PubMed] [Google Scholar]
- Pujol A, Crost EH, Simon G, et al. Characterization and distribution of the gene cluster encoding RumC, an anti-C lostridium perfringens bacteriocin produced in the gut. FEMS Microbiol Ecol 2011 ; 78 : 405–415. [CrossRef] [PubMed] [Google Scholar]
- Chiumento S, Roblin C, Kieffer-Jaquinod S, et al. Ruminococcin C, a promising antibiotic produced by a human gut symbiont. Sci. Adv. 2019; 5 : eaaw9969. [CrossRef] [Google Scholar]
- Roblin C, Chiumento S, Bornet O, et al. The unusual structure of Ruminococcin C1 antimicrobial peptide confers clinical properties. Proc Natl Acad Sci USA 2020; 117 : 19168–77. [CrossRef] [PubMed] [Google Scholar]
- Roblin C, Chiumento S, Jacqueline C, et al. The Multifunctional Sactipeptide Ruminococcin C1 Displays Potent Antibacterial Activity In Vivo as Well as Other Beneficial Properties for Human Health. Int J Mol Sci 2021; 22 : 3253. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Modèle de synthèse et de maturation de RumA et RumC. Les ruminococcines RumA et RumC sont synthétisées et subissent une maturation selon deux mécanismes distincts. Les différentes étapes de synthèse et de maturation de RumA (en rouge) et RumC (en bleu) sont indiquées par des numéros. La transcription de rumA est activée par un peptide inducteur (PI) après clivage de ce dernier par la trypsine présente dans le milieu extracellulaire (produite par l’hôte). RumC est produite dans la cellule sous forme inactive, puis subit une première étape de clivage du peptide signal par RumPc. Une fois sécrétés, les 5 résidus en N-terminal de RumC sont clivés par la trypsine présente dans le milieu extracellulaire, ce qui produit la forme active du sactipeptide RumC (figure créée avec BioRender). |
| Dans le texte | |
 |
Figure 2. Relations entre Ruminococcus gnavus E1 et l’hôte dans le cadre de la production de ruminococcines. Ruminococcus gnavus E1 produit et sécrète plusieurs bactériocines, dont RumA et RumC. Ces deux peptides subissent des étapes de maturation intracellulaires qui permettent de former des ponts thioéther dans leur structure tridimensionnelle. Les résidus cystéine, donneurs de soufre pour les liaisons thioéther, sont représentés en rose. Les autres résidus impliqués dans la formation des liaisons covalentes au sein des formes matures de RumA et RumC sont indiqués en couleur. Ces deux bactériocines possèdent une activité anti-bactérienne contre différentes espèces dont certaines bactéries multi-résistantes aux antibiotiques. Les mécanismes qui restent à démontrer sont représentés par les flèches en traits pointillés (figure créée avec BioRender). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




