Figure 1.
Télécharger l'image originale
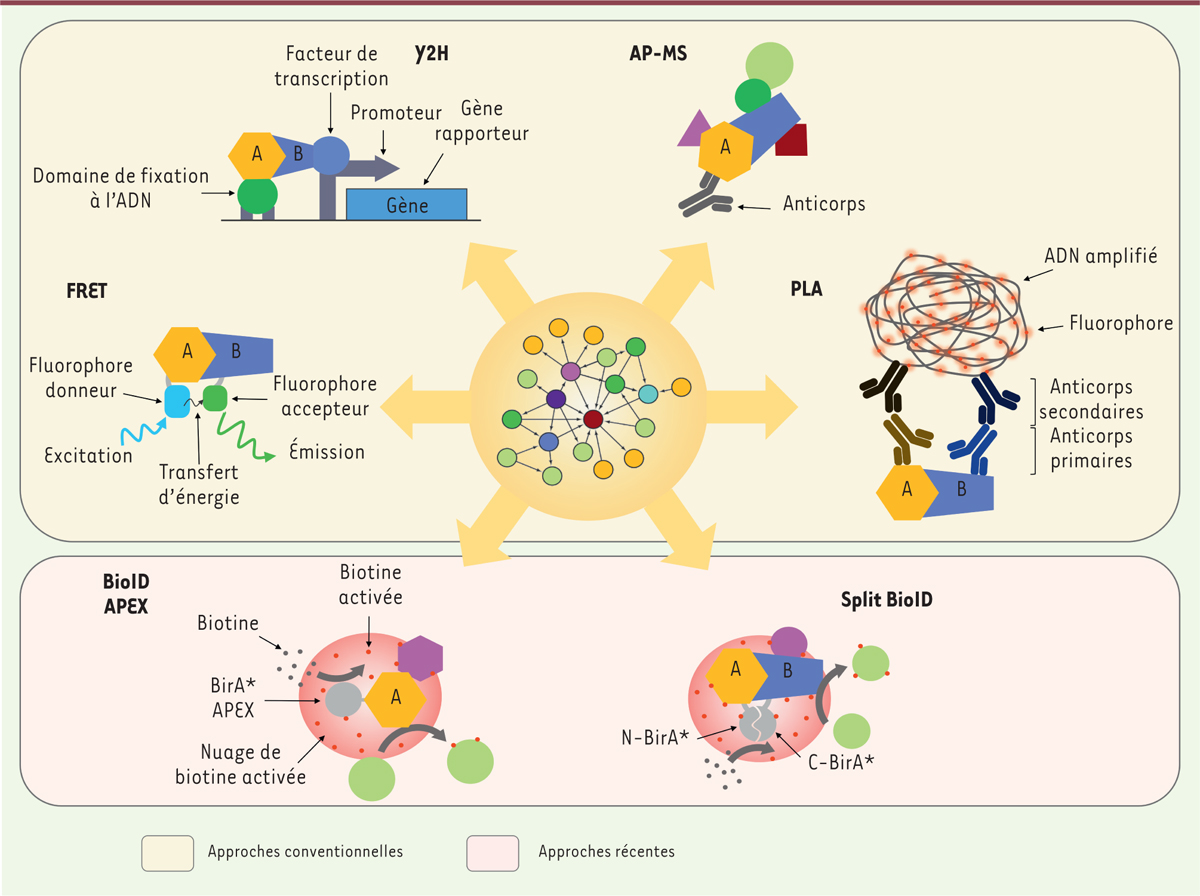
Vue d’ensemble des techniques d’identification des interactions protéine-protéine. Le réseau d’interaction, au centre du disque orange, peut être construit au moyen d’approches conventionnelles (Y2H, AP-MS, FRET, PLA) ou d’approches récentes (APEX, BioID, Split-BioID). Les techniques d’APEX, BioID et AP-MS sont principalement utilisées pour caractériser le réseau d’interactions d’une protéine « A » sans a priori d’interactions. À l’inverse, les techniques de Y2H, FRET et PLA sont principalement utilisées pour valider une interaction supposée entre une protéine « A » et une protéine « B ». Les techniques présentées ne sont pas exhaustives. AP-MS : affinity purification-mass spectrometry ; APEX : engineered ascorbate peroxidase ; BioID : proximity-dependent biotinylation identification ; FRET : Förster resonance energy transfer ; PLA : proximity ligation assay ; Y2H : yeast-two-hybrid.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




