| Issue |
Med Sci (Paris)
Volume 33, Number 2, Février 2017
|
|
|---|---|---|
| Page(s) | 159 - 168 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20173302011 | |
| Published online | 27 February 2017 | |
Application à la biologie synthétique des méthodes et outils de CAO de la microélectronique
Application of microelectronics CAD tools to synthetic biology
Laboratoire des sciences de l’ingénieur, de l’informatique et de l’imagerie (ICube), 300, boulevard Sébastien Brandt, 67412
Illkirch, France
La biologie synthétique est une science émergente dont l’objectif est de créer de nouvelles fonctions biologiques, encore inexistantes dans la nature, à partir des connaissances sur le vivant acquises ce dernier siècle. Jusqu'à peu, les systèmes biologiques synthétiques artificiels étaient de complexité raisonnable. Ce n’est plus le cas aujourd’hui, ce qui a conduit au développement de nombreux logiciels de conception assistée par ordinateur. Une des voies permettant la mise au point rapide et efficace de ces logiciels est l’adaptation d’outils existant déjà dans différents domaines de l’ingénierie, et en particulier la microélectronique.
Abstract
Synthetic biology is an emerging science that aims to create new biological functions that do not exist in nature, based on the knowledge acquired in life science over the last century. Since the beginning of this century, several projects in synthetic biology have emerged. The complexity of the developed artificial bio-functions is relatively low so that empirical design methods could be used for the design process. Nevertheless, with the increasing complexity of biological circuits, this is no longer the case and a large number of computer aided design softwares have been developed in the past few years. These tools include languages for the behavioral description and the mathematical modelling of biological systems, simulators at different levels of abstraction, libraries of biological devices and circuit design automation algorithms. All of these tools already exist in other fields of engineering sciences, particularly in microelectronics. This is the approach that is put forward in this paper.
© 2017 médecine/sciences – Inserm

Ces quinze dernières années, la biologie synthétique a connu un essor important. Cette nouvelle science, à l’interface entre les biotechnologies et les sciences pour l’ingénieur, correspond à la transition d’une biologie purement expérimentale et descriptive à une biologie plus computationnelle et prédictive. Ainsi, l’objet de la biologie synthétique est de réinvestir les connaissances du vivant acquises au cours de ces dernières décennies afin de créer de nouvelles fonctions biologiques artificielles [1, 2, 51, 52] (→) à l’image de ce qui est réalisé dans d’autres disciplines de l’ingénierie comme le développement logiciel ou l’électronique.
(→) Voir la Synthèse de F. Képès, m/s hors série n° 2, mai 2009, page 39, et la Synthèse de J. Haiech, m/s hors série n° 2, mai 2013, page 43
Le champ d’application de la biologie synthétique est très vaste. Il inclut la mise au point de nouvelles thérapies [3] et de thérapies ciblées [4], la réduction des coûts de synthèse de certains médicaments [5, 6], le diagnostic in vivo [7, 8], la conception de nouvelles fonctions traitant directement des signaux biologiques [9–12], la mise au point de biocapteurs [3, 9, 13–15], l’agriculture et l’agronomie [16] ou encore l’environnement [17, 18].
Parmi les applications les plus marquantes de ces dernières années, nous pouvons citer les travaux de Keasling sur un nouveau procédé de fabrication de l’artémisinine, molécule à la base des traitements anti-paludéens que l’on extrait classiquement de l’armoise annuelle [6, 53] (→).
(→) Voir l’article Nobel de D. Mazier et M. Thellier, m/s n° 1, janvier 2016, page 106
Ce nouveau procédé utilise une levure classique (Saccharomyces cerevisae) reprogrammée avec 12 gènes artificiels (néo-synthétisés) qui la rendent capable de produire de l’acide artémisinique (un précurseur de l’artémisinine) directement à partir du glucose. Les levures permettent d’atteindre des rendements exceptionnels, réduisant ainsi considérablement le coût de production du médicament. Il s’agit là de la première application commerciale connue issue de la biologie synthétique puisque ces travaux ont donné lieu à la création de l’entreprise Amyris et que le procédé, décrit ci-dessus, est exploité depuis 2013 par la société Sanofi pour la synthèse de médicaments anti-paludéens. Cet exemple, comme beaucoup d’autres, illustre le fort potentiel applicatif de la biologie synthétique.
Jusqu’à présent, les circuits biologiques artificiels ont été conçus à la main. Cette démarche est de plus en plus délicate au fur et à mesure de l’augmentation de la complexité des fonctions réalisées. L’utilisation d’outils d’aide à la conception (CAO) dédiés à la biologie de synthèse devient donc indispensable. Jusqu’alors, ces outils étaient développés dans un contexte très spécifique, ce qui en limite leur application à d’autres utilisations. C’est ici que les principes de l’ingénierie des systèmes, et en particulier de l’électronique, entrent en jeu.
Dès le début des années 2000, l’idée est née de développer des gènes artificiels standardisés, caractérisés, réutilisables dans des contextes divers et dont le comportement soit complètement découplé de leur environnement. Cette manière de penser a conduit à la notion de composants biologiques (les « parts » ou composants-modèles) ou de BioBricks (voir Glossaire) introduites entre autres par T. Knight, D. Endy et C. Voigt [19, 21]. L’analogie entre ces parts et les composants d’un circuit électronique est assez naturelle [19]. Aujourd’hui, des bibliothèques existent. Elles sont riches en nombre de parts, mais elles restent malheureusement difficiles à exploiter dans un contexte de conception, principalement par manque de standardisation et de caractérisation.
Les systèmes biologiques
Au-delà des analogies que nous pouvons trouver dans les approches de conception, l’électronique et la biologie se rapprochent également dans la manière de décrire les systèmes. À haut niveau d’abstraction1, un système biologique peut être représenté comme un réseau d’interactions où la production, ou non, de chaque molécule chimique peut être contrôlée par la présence, ou l’absence, d’autres molécules. Un exemple simple est donné dans la Figure 1A . Le système décrit est un réseau de régulation génétique (ou RGN pour gene regulatory network), une mémoire à deux états qui correspond à l’un des premiers circuits biologiques artificiels réalisés en 2000 par Gardner [22]. Le fonctionnement du système peut être résumé par les propositions logiques suivantes :
-
La GFP (green fluorescent protein) n’est synthétisée que si le promoteur P1 est actif.
-
Le facteur de transcription R2 n’est synthétisé que si le promoteur P1 est actif.
-
Le facteur de transcription R1 n’est synthétisé que si le promoteur P2 est actif.
-
Le promoteur P1 est actif si le facteur de transcription (répresseur) R1 est absent ou s’il est présent mais lui-même inhibé par I1.
-
Le promoteur P2 est actif si le facteur de transcription (répresseur) R2 est absent ou s’il est présent mais lui-même inhibé par I2.
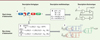 |
Figure 1. Schéma de passage de la description biologique (A) à un modèle électronique équivalent. À haut niveau d’abstraction, nous obtenons un schéma à base de portes logiques (C) qui correspond à une description mathématique à partir d’opérateurs booléens (B) . À bas niveau d’abstraction, nous obtenons un schéma électrique connectant des composants électriques standards et des transistors biologiques (E) , le tout correspondant à une description mathématique sous forme d’équations différentielles couplées (D) . GFP : green fluorescent protein ; I1/2 : inhibiteur 1/2 ; P1/2 : promoteur 1/2 ; R1/2 : facteur de transcription répresseur 1/2. |
Ces propriétés peuvent être traduites en équations booléennes (Figure 1B) et en un circuit électronique équivalent à base de portes logiques2 (deux inverseurs et deux portes logiques « OU », Figure 1C ). Le schéma électronique obtenu à partir de la description biologique correspond exactement à celui d’une mémoire R-S 3, un composant de base de l’électronique numérique [23]. L’abstraction numérique est utilisée aujourd’hui pour la conception de fonctions biologiques logiques servant principalement à la détection de signatures moléculaires dans les cellules. Nous pouvons citer, à titre d’exemple, les travaux de Xie en 2011 [9] portant sur l’identification de lignées de cellules cancéreuses (les cellules HeLa) en fonction d’une signature constituée de six microARN (miARN). L’architecture du circuit génétique utilisé par Xie étant assez générique, elle peut facilement être adaptée à d’autres signatures et modèles cellulaires. En parallèle, de nombreux travaux ont été menés sur l’intégration de fonctions de calcul logique dans les cellules [10, 24] avec pour objectif à long terme la fabrication de bio-ordinateurs [25, 26].
Dans le même temps, ce système peut être décrit à un niveau d’abstraction inférieur par un jeu d’équations différentielles régissant l’évolution dans le temps de la concentration de chacune des espèces chimiques qui sont impliquées. Quel que soit le problème biologique, ce jeu d’équations est toujours obtenu de la même manière : pour chaque espèce, la dérivée temporelle de sa concentration est écrite comme la somme de toutes les vitesses des réactions chimiques qui produisent ou qui consomment l’espèce (Figure 1D). Cette définition peut être transposée au domaine de l’électronique en considérant la loi des nœuds4 appliquée aux bornes d’un condensateur : la dérivée de la charge d’un condensateur (et donc de la tension à ses bornes) est égale à la somme des courants qui arrivent (et qui repartent) de celui-ci. Les vitesses de réaction en biologie peuvent dépendre de la concentration des autres espèces présentes dans le milieu. Nous retrouvons ici le rôle d’un transistor en électronique (modulation du courant traversant le transistor en fonction du potentiel de grille5). Ainsi, un système biologique peut être vu comme un circuit électronique qui sera composé de résistances, de condensateurs et de « transistors biologiques » modélisant les interactions (activation et inhibition) entre les espèces (Figure 1E) [27].
Logiciel de CAO pour la biologie des systèmes et la biologie synthétique
Les besoins d’outils de conception assistée par ordinateur (CAO) pour la biologie synthétique ont été exprimés très tôt dans la genèse de cette nouvelle science [28, 29]. Depuis le milieu de la dernière décennie, beaucoup d’outils de CAO pour la biologie des systèmes et pour la biologie synthétique ont vu le jour. Nous pouvons les classer en trois grandes catégories : les outils servant à la description des systèmes biologiques, les outils de simulation, et les outils d’aide à la conception ou à son automatisation.
Dans un premier temps, il est important de pouvoir décrire un système biologique de manière univoque, exploitable par un logiciel et compréhensible par un utilisateur. Depuis quelques années, le langage system biology markup language (SBML) s’est imposé comme un standard [30]. SBML est en effet utilisé aujourd’hui par plus de 200 outils et, en premier lieu, ceux qui servent à la description graphique de systèmes biologiques. Virtual Cell [31] en est un exemple. Cette plateforme logicielle permet de dessiner des réseaux biologiques complexes en définissant des compartiments, des espèces chimiques interagissant dans ces compartiments, et les équations de la vitesse des réactions mises en jeu, puis de générer un fichier SBML qui correspond à ce graphique. SBML est aussi un langage utilisé comme standard de partage de domaine pour de nombreuses banques de données comme BioModel qui regroupe des modèles mathématiques d’intérêt biologique [32].
La description SBML est également utilisée comme support pour de nombreux outils de simulation, le plus complet et utilisé d’entre eux étant sans doute CoPaSi [33]. Il permet la simulation transitoire, la recherche des états stationnaires, l’analyse stœchiométrique, la simulation stochastique et l’analyse de la sensibilité de la réponse aux paramètres. CoPaSi est intégré à certains logiciels de description, par exemple Virtual Cell. À côté de CoPaSi, de nombreux autres simulateurs, également fondés sur des descriptions SBML, génériques ou développés pour des applications, existent et sont pour la plupart libres d’accès (open source) [34].
En ce qui concerne la CAO pour la biologie synthétique, plusieurs outils ont également été développés [28]. GenoCAD en est un des pionniers. Il propose une interface graphique permettant la conception de vecteurs d’expression des protéines et de réseaux de gènes artificiels. En interne, il s’appuie sur un langage formel permettant de décrire et de manipuler facilement des circuits génétiques [35]. Dans GenoCAD, l’utilisateur manipule des parts connues extraites d’une bibliothèque et les assemble manuellement jusqu’à obtenir le résultat voulu. L’étape d’automatisation devrait prédire l’assemblage à réaliser directement à partir de la fonction que l’on veut atteindre. Cette étape peut être réalisée à l’aide de certains logiciels, par exemple BioJADE [36], mais le plus souvent elle se cantonne à la description à haut niveau (c’est-à-dire booléenne) des systèmes. Parmi les logiciels de CAO les plus complets et aboutis, nous pouvons également citer TASBE (toolchain to accelerate synthetic biology engineering project) qui est une suite logicielle composée d’un langage servant à décrire une fonction biologique à haut niveau (Proto), un outil permettant de transformer cette description en un réseau de régulation génétique abstrait (BioCompiler), un outil permettant de positionner sur ce réseau génétique des parts (MatchMaker), et un outil d’assemblage permettant de donner directement la séquence ADN à synthétiser correspondant à ce réseau de régulation génétique [37]. La suite logicielle Cello est un équivalent de TASBE. Elle permet également de synthétiser des réseaux de régulation génétique (GRN, genetic regulatory networks) à partir d’une description booléenne au travers de différentes étapes, certaines partageant les mêmes outils que TASBE [38].
L’ensemble des outils décrits dans cette section réalise, dans le contexte de la biologie synthétique, des opérations qui équivalent à celles que l’on est amené à effectuer lors de la conception de circuits électroniques. ICube développe une approche différente. Elle consiste à étudier la possibilité de transférer certains de ces outils existants à la biologie synthétique plutôt que d’en développer de novo pour ce contexte spécifique. Nous illustrerons ce concept par trois exemples : l’utilisation d’un synthétiseur logique pour la conception de réseaux de régulation génétique (GNR) abstraits ; la simulation de systèmes biologiques à l’aide de simulateurs de circuits électroniques de type SPICE (simulation program with integrated circuit emphasis) ; et l’adaptation d’un outil de simulation électrothermique des circuits intégrés à la simulation de problèmes biologiques dépendant de l’espace et du temps.
Utilisation d’un synthétiseur logique pour la conception automatisée de GRN abstraits
En électronique, la conception d’un circuit numérique complexe, qui peut comporter plusieurs centaines de millions de transistors, ne se fait plus manuellement depuis plus de 30 ans [39]. Le principe de base est de fournir à un outil appelé synthétiseur logique une description à haut niveau de la fonction que nous souhaitons réaliser ainsi qu’une bibliothèque de fonctions élémentaires à assembler. Le synthétiseur opère par calcul formel pour trouver la combinaison d’éléments de la bibliothèque qui réalise la fonction désirée à moindre coût. Prenons l’exemple de la mémoire décrite dans la Figure 1 . Si nous donnons au synthétiseur logique la description de haut niveau suivante : « La sortie est active si R1 vaut 1, ou si elle était déjà active et si R2 vaut 0 », et une bibliothèque contenant uniquement un inverseur et une porte « OU », celui-ci fournit, en sortie, le schéma électronique de la Figure 1C . Si nous donnons, en plus, dans la bibliothèque une mémoire R-S dont le coût est inférieur à celui de deux inverseurs et deux portes « OU », il choisira d’implémenter directement une mémoire R-S.
Dans le cadre de nos travaux, nous avons créé le logiciel GeNeDA (genetic network design automation)6 en adaptant un synthétiseur logique open source dédié à la conception de circuits électroniques et en y ajoutant certaines contraintes propres à la biologie [40]. Ce synthétiseur logique est associé à une bibliothèque contenant une quinzaine de parts (fonctions logiques élémentaires) réalisables à l’aide de réseaux de régulation génétique (principalement des promoteurs avec un nombre variable d’activateurs ou de répresseurs). Le fichier d’entrée est une description en langage Verilog [41], un langage de programmation, qui peut être saisie manuellement ou générée à partir d’une table de vérité. Le résultat de la synthèse est un fichier de type texte indiquant les parts utilisées ainsi que leur interconnexion. La visualisation du réseau génétique peut également être réalisée à l’aide du logiciel PigeonCAD [42] dont le fichier d’entrée est généré par GeNeDA. L’approche est assez similaire à celle utilisée par Nielsen et al. [38] pour les premières étapes du flot de conception de Cello.
Le logiciel a été testé sur une série de circuits logiques allant de la simple porte logique à un petit microprocesseur. Il a donné des résultats pertinents, mettant notamment en avant des optimisations possibles de certains circuits difficiles à mettre en œuvre si l’on effectuait la synthèse manuellement. Les résultats dépendent beaucoup de la liste des parts disponibles dans la bibliothèque ainsi que du coût associé à chacune d’entre elles. À titre d’exemple, les Figures 2A et 2B montrent deux résultats de synthèse pour une fonction logique « ET » à trois entrées (la GFP n’est synthétisée que si les 3 protéines d’entrée sont présentes) selon que nous autorisons, ou pas, l’utilisation d’un promoteur régulé par trois protéines. La Figure 2D représente le résultat de synthèse d’un séquenceur à trois états permettant la régulation de la concentration d’une espèce donnée et décrit par le graphe d’état de la Figure 2C.
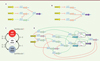 |
Figure 2. Résultats de synthèse pour une porte « ET » à 3 entrées et un séquenceur à 3 états. Dans le cas de la porte « ET » à 3 entrées, deux résultats de synthèse sont donnés : un dans le cas où la bibliothèque ne contient que des parts autorisant au plus 2 facteurs de transcription sur un promoteur (A) et un dans le cas où la bibliothèque contient des parts autorisant jusqu’à 3 facteurs de transcription par promoteurs (B) . Le séquenceur est quant à lui décrit en (C) . Il est constitué de 3 états : l’état cold que l’on atteint quand X est en défaut (matérialisé par les entrées A = 0 et B = 0) et dans lequel on synthétise davantage de X, l’état hot que l’on atteint quand X est en excès (matérialisé par A = 0 et B = 1) et dans lequel on synthétise une molécule Y dégradant X et un état idle dans lequel rien ne se passe. Le résultat de synthèse en limitant la bibliothèque à des parts à deux régulations est donné en (D) (d’après [40]). GFP : green fluorescent protein ; Op : opérateur. |
Simulation de systèmes biologiques à l’aide d’un simulateur électronique
Les besoins en terme de simulation pour la biologie des systèmes et la biologie synthétique sont proches de ceux nécessaires lors de la simulation des circuits électroniques analogiques : calcul du point de fonctionnement, analyse transitoire, analyse paramétrique, analyse du bruit, etc. Comme la description d’un système biologique peut être traduite sous la forme de circuits électroniques équivalents (Figure 1), l’utilisation des simulateurs électroniques, plus matures et plus complets que les simulateurs biologiques actuels, peut s’avérer intéressante. Nous avons montré qu’il était possible de simuler les réseaux de régulation génétique décrits en VHDL-AMS (very high speed integrated circuits hardware description language – analog-mixed signal), un langage de description de matériel universellement utilisé dans le contexte des systèmes électroniques et multiphysiques [43]. Le modèle VHDL-AMS est établi en utilisant l’analogie décrite ci-dessus. Il est généré automatiquement par un outil doté d’une interface graphique dans laquelle l’utilisateur spécifie les espèces chimiques interagissant dans le système, les gènes impliqués et le type de mécanismes biologiques intervenant entre les espèces (régulation de l’expression d’un gène, traduction d’ARN messager, réaction de liaison, réaction enzymatique, etc.) ainsi que les paramètres liés à ceux-ci.
Par la suite, une seconde version du simulateur a été développée avec pour objectifs l’extension à tous les types de mécanismes biologiques et l’intégration directe de descriptions écrites en SBML, celles-ci contenant l’ensemble des informations nécessaires à l’établissement du modèle VHDL-AMS. Un outil de traduction de modèle, développé dans le langage Java, assure le passage d’un langage à l’autre [44]. La description SBML pouvant elle-même être générée à partir d’un outil graphique (comme Virtual Cell), nous obtenons donc une suite logicielle complète, allant de la saisie du schéma biologique à sa simulation. Les résultats obtenus sont présentés sur la Figure 3 . Le cas d’étude utilisé pour illustrer les performances de notre approche est un test de compétition par ELISA7 [45]. Ce test a pour vocation d’être implémenté dans un système de détection complet (laboratoire sur puce) dédié à la détection de polluants dans l’eau potable (le test reposant sur l’induction d’une variation de pH dépendant de la concentration en polluants et détectable par un capteur électronique IsFET8) [46]. Un grand avantage de travailler en VHDL-AMS pour la partie biologique est qu’il devient alors simple de coupler le modèle biologique aux modèles électroniques et fluidiques, également établis en VHDL-AMS. Nous obtenons ainsi un modèle prédictif du laboratoire sur puce complet que nous pouvons simuler en un seul et unique environnement. À titre d’exemple, la Figure 3D montre les variations au cours du temps, tout au long du protocole de test, de la tension en sortie du capteur en fonction de la concentration initiale de la molécule que l’on cherche à détecter. Ces résultats de simulation facilitent le dimensionnement du circuit électronique associé au capteur afin d’optimiser ses performances.
 |
Figure 3. Résultats de simulation obtenus sur un test de compétition par ELISA. A. Description du système biologique effectuée sous Virtual Cell. B. Comparaison entre les résultats de simulation obtenus avec Virtual Cell (points noirs) et le modèle VHDL-AMS (traits rouges). C. Simulation réalisée avec le modèle VHDL-AMS illustrant le test par compétition : plus la quantité d’antigènes non marqués (protéine cible) est faible dans l’échantillon (différentes courbes), plus il y a d’antigènes marqués (protéine cible couplée à une enzyme) fixés à l’anticorps à l’équilibre. D. Simulation du lab-on-chip complet intégrant le modèle VHDL-AMS précédemment discuté, un modèle de l’IsFET transformant les variations de pH en signal électrique et des modèles simplifiés écrits en VHDL-AMS pour les parties microfluidiques (d’après [44]). (2) : signifie que la réaction produit 2NH3 ; ELISA : enzyme-linked immunosorbent assay ; IsFET : ion-sensitive field-effect transistor ; VHDL-AMS : very high speed integrated circuits hardware description language-analog-mixed signal. |
Adaptation d’un simulateur électrothermique à la simulation spatio-temporelle en biologie
Jusqu’à présent, les systèmes biologiques dont nous avons discuté ne font pas intervenir la notion d’espace : nous avons considéré que les réactions mises en jeu se réalisent en tout point de l’espace et que les concentrations des espèces chimiques impliquées sont homogènes en tout point de cet espace. Ce n’est malheureusement pas toujours le cas en pratique. Pour la simulation de certains systèmes, il est nécessaire de définir des réactions localisées ou des espèces dont la concentration varie dans l’espace [13, 47, 48]. Cette problématique rappelle celle de la prise en compte des effets thermiques (sources localisées de chaleur, propagation de la chaleur) dans les circuits intégrés électroniques. En effet, certains transistors, très sollicités, ont tendance à chauffer, ce qui provoque des gradients de température au sein de la puce, perturbant le fonctionnement d’autres transistors du circuit. Pour étudier ces phénomènes et les intégrer précocement dans la conception des circuits, des simulateurs électrothermiques sont développés dans lesquels sont intégrés d’une part les effets de la température sur le fonctionnement des circuits et, d’autre part, un modèle thermique de la puce qui consiste principalement à discrétiser, dans l’espace, les équations de la chaleur [49].
D’un point de vue mathématique, les équations régissant la diffusion de la chaleur dans l’espace sont similaires à celles déterminant la diffusion de molécules dans un milieu. Nous pouvons donc utiliser directement les principes des simulateurs électrothermiques dans ce contexte. L’outil développé est constitué de trois éléments principaux [50] :
-
Un outil de maillage qui divise l’espace en un certain nombre de compartiments. Pour obtenir un bon compromis entre la complexité des équations de diffusion (et donc le temps de calcul) et la précision du simulateur, nous avons choisi un maillage adaptatif carré (ou cubique en 3 dimensions), c’est-à-dire que les compartiments carrés sont d’autant plus fins qu’ils se situent proche des zones à fort gradient de concentration (Figure 4B). Cet outil est une application directe de celui utilisé pour le simulateur électrothermique.

Figure 4. Résultats de simulation obtenus avec le simulateur spatio-temporel sur un système oscillant à deux cellules. Le système est décrit en A . Il est composé d’une cellule (1) fabriquant une protéine qui active un gène de la cellule (2), qui lui-même code pour une autre protéine qui réprime le gène de la cellule (1). Les nœuds du maillage obtenus sont représentés en B . Les résultats de simulation sont donnés sur les courbes : concentration d’activateur et du répresseur au niveau de la cellule (1) (C) , concentration d’activateur et du répresseur au niveau de la cellule (2) (D) .
-
Un modèle élémentaire pour chaque maille qui traduit la manière dont les molécules diffusent au sein de la maille. Ce modèle calcule les flux de molécules arrivant et partant au niveau de chaque nœud de la maille proportionnellement aux gradients de concentration dans la maille. Les modèles sont ajustables selon certains paramètres permettant de rendre compte de comportements plus complexes, par exemple la modélisation des membranes plus ou moins perméables, des vésicules, etc.
-
Une série de modèles décrivant les réactions chimiques se produisant en chaque nœud du maillage. Ces modèles correspondent aux modèles classiques des systèmes biologiques que nous avons décrits.
Les résultats obtenus sont présentés dans la Figure 4 . Le système décrit est composé de deux types de cellules. Les cellules 1 contiennent un gène codant une molécule qui active l’expression d’un gène contenu dans la cellule 2. Ce gène code, quant à lui, une seconde molécule qui réprime un gène contenu dans la cellule 1 (Figure 4A). Nous effectuons un maillage adaptatif raffiné autour de la position des deux cellules (Figure 4B). Les résultats de simulation mettent en évidence le caractère oscillant du système avec une fréquence qui dépend de la distance entre les deux cellules (Figure 4C–D).
Discussion et conclusion
L’approche de conception globale de la biologie synthétique ainsi que les travaux spécifiques décrits dans cette revue montrent l’intérêt du transfert de certains outils et de certaines méthodologies de CAO issues de la microélectronique. Cependant, la CAO n’est pas la seule clef du succès. Ce qui a permis l’essor de la microélectronique, depuis le début des années 1970 jusqu’à nos jours, est une subtile combinaison de trois principaux ingrédients : des outils de CAO très performants, le développement de circuits et de processus de fabrication des circuits intégrés standards, fiables et à bas coût, et un modèle économique fort poussant en permanence les limites des performances. Aujourd’hui, malgré les progrès incroyables des biotechnologies ces dernières années, la biologie synthétique n’a pas encore atteint un niveau de maturité permettant de voir émerger ces trois volets.
Si le potentiel d’applications de la biologie synthétique est fort, en particulier dans des domaines dotés d’une grande puissance économique (thérapeutiques, environnement, etc.), le transfert de technologies depuis l’académique vers l’industrie reste encore rare et difficile. Les travaux de recherches de Jay Keasling sur l’artémisinine [6], dont le brevet est exploité par Sanofi depuis 2013, font figure d’exception. Le principal frein à ce transfert est le coût et le manque de fiabilité des procédés de fabrication. C’est le principal point faible sur lequel la biologie synthétique devra progresser dans les années à venir. L’absence de procédés standards de fabrication de circuits biologiques est également un frein à la démocratisation de ces technologies. Aujourd’hui, pour mettre au point de nouveaux systèmes en biologie synthétique, il est nécessaire de disposer de moyens matériels lourds et d’une équipe maîtrisant tous les volets, de leur conception in silico jusqu’à leur réalisation in vivo en intégrant toutes les phases de tests. L’électronique est parvenue, par le biais d’un certain nombre de standards, à séparer ces métiers et, aujourd’hui, une équipe d’ingénieurs peut concevoir un circuit intégré à l’aide d’outils de CAO sans avoir aucune idée de la manière dont celui-ci sera réalisé par la suite : la réalisation pratique du circuit sera confiée à un sous-traitant qui travaillera à partir de fichiers de données fournis par le logiciel de CAO.
Si nous faisons le bilan, il semble que les outils de CAO pour la biologie synthétique aient quelques longueurs d’avance par rapport aux technologies auxquelles ils sont associés. C’est en grande partie parce que ceux-ci concernent les étapes en amont de la conception, celles où les aspects biologiques à prendre en compte dans les outils restent limités et donc celles pour lesquelles des travaux antérieurs, issus d’autres domaines de l’ingénierie, ont pu être investis. La palette d’outils de CAO devra donc être complétée, dans les années à venir, par des outils allant davantage au cœur de la biologie et intégrant les contraintes qui lui sont propres. À ce niveau d’abstraction, des outils existent également dans le domaine de la microélectronique, mais rien ne dit que ceux-ci pourront être adaptés aussi facilement que ceux que nous présentons ici. Il est fort probable que des outils propres à la biologie devront être développés pour répondre à des besoins très spécifiques.
L’automatisation du flot de conception de biosystèmes synthétiques répondant à des cahiers des charges spécifiques est donc un projet à long terme, mais le potentiel applicatif qu’il laisse envisager mérite les efforts que la communauté scientifique y investit. Le développement conjugué d’outils informatiques et de procédés de fabrication est la clef du succès. Concernant les outils, la microélectronique a pu apporter des premières solutions. Dans les années à venir, même si elle ne saura pas répondre à toutes les problématiques, la philosophie et les méthodologies autour de la CAO pour la microélectronique pourront continuer à servir de guide à la biologie synthétique afin de lui permettre, à moyen terme, de connaître un succès scientifique et économique équivalent à celui que ce domaine a connu.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
En sciences pour l’ingénieur, le niveau d’abstraction d’une description désigne la précision avec laquelle l’objet est décrit. Un haut niveau d’abstraction correspond à une description fonctionnelle très simplifiée tandis que des niveaux d’abstraction bas correspondent à des descriptions de plus en plus détaillées.
Un transistor est un composant électronique à trois pattes : la grille, le drain et la source. Le transistor peut être utilisé dans diverses applications, la plus courante étant une modulation du courant qui s’écoule entre le drain et la source en fonction du potentiel de grille ; c’est notamment ce qui est utilisé dans les amplificateurs mais aussi dans les portes logiques (qui fonctionnent en tout ou rien).
Le logiciel GeNeDA est disponible en ligne à l’adresse suivante : www.geneda.fr
Glossaire
BioBricks : : base de données de séquences d’ADN artificielles, caractérisées et découplées de leur environnement que l’on peut assembler pour la réalisation de réseaux génétiques artificiels. Les séquences élémentaires sont appelées parts et sont assemblés en device puis en systems.
CAO : : conception assistée par ordinateur. Ensemble d’outils logiciels aidant l’ingénieur au cours des différentes étapes de conception d’un système.
Électronique numérique : : famille de circuits électroniques pour lesquels les fonctions de chaque bloc peuvent être décrites par une équation logique.
Équations booléennes : : relation tout-ou-rien liant plusieurs signaux. Les trois opérateurs principaux des équations booléennes sont le « NON » (le signal de sortie est actif si le signal d’entrée ne l’est pas), le « OU » (le signal de sortie est actif si au moins un des signaux d’entrée est actif) et le « ET » (le signal de sortie n’est actif que si tous les signaux d’entrée le sont).
Mémoire : : composant logique dont la fonction est de mémoriser un état (actif ou inactif) tant qu’aucune action n’est menée sur celui-ci. L’exemple type est la mémoire R-S disposant d’une entrée permettant de mettre la mémoire à 0 et une permettant de la mettre à 1. Tant qu’aucune des entrées n’est activée, le système conserve son état précédent.
Niveau d’abstraction d’une description : : degré de précision avec lequel un système est décrit. À haut niveau d’abstraction, le système est décrit dans son ensemble et par sa fonction principale. À bas niveau d’abstraction, chaque sous-système le composant est décrit en détail. On parle aussi d’abstraction booléenne lorsqu’il est possible de décrire la fonction principale d’un système par une équation logique.
GRN : : réseaux de régulation génétique, ou gene regulatory networks. Ensemble de gènes codant des protéines dont la fonction principale est de contrôler (inhiber, activer) l’expression d’autres gènes de l’ensemble.
Porte logique : : composant de base de l’électronique numérique permettant de réaliser une fonction logique élémentaire (voir équations booléennes) entre plusieurs signaux.
SBML : : langage utilisé pour la description de systèmes biologiques.
SPICE : : langage et logiciel permettant la simulation de circuits électroniques.
Synthétiseur logique : : outil du flot de CAO de l’électronique numérique permettant de générer automatiquement un circuit de portes logiques à partir d’une description sous la forme d’une équation logique.
Verilog : : langage utilisé pour la description de fonctions logiques complexes.
VHDL-AMS : : langage utilisé pour la description de systèmes multi-physiques complexes.
Références
- Beal J, Weiss R, Densmore D, et al. An end-to-end workflow for engineering of biological networks from high-level specifications. ACS Synth Biol 2012 ; 1 : 317–331. [Google Scholar]
- Endy D. Foundations for engineering biology. Nature 2005 ; 438 : 449–453. [CrossRef] [PubMed] [Google Scholar]
- Saeidi N, Wong CK, Lo TM, et al. Engineering microbes to sense and eradicate Pseudomonas aeruginosa, a human pathogen. Mol Syst Biol 2011 ; 7 : 521. [CrossRef] [PubMed] [Google Scholar]
- Lo TM, Tan MH, Hwang IY, Chang MW Designing a synthetic genetic circuit that enables cell density-dependent auto-regulatory lysis for macromolecule release. Chem Eng Sci 2013 ; 103 : 29–35. [Google Scholar]
- Weber W, Schoenmakers R, Keller B, et al. A synthetic mammalian gene circuit reveals antituberculosis compounds. Proc Natl Acad Sci USA 2008 ; 105 : 9994–9998. [CrossRef] [Google Scholar]
- Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature 2006 ; 440 : 940–943. [CrossRef] [PubMed] [Google Scholar]
- Gilad AA, McMahon MT, Walczak P, et al. Artificial reporter gene providing MRI contrast based on proton exchange. Nat Biotechnol 2007 ; 25 : 217–219. [CrossRef] [PubMed] [Google Scholar]
- Jaffe EK, Volin M, Bronson-Mullins CR, et al. An artificial gene for human porphobilinogen synthase allows comparison of an allelic variation implicated in susceptibility to lead poisoning. J Biol Chem 2000 ; 275 : 2619–2626. [CrossRef] [PubMed] [Google Scholar]
- Xie Z, Wroblewska L, Prochazka L, et al. Multi-input RNAi-based logic circuit for identification of specific cancer cells. Science 2011 ; 333 : 1307–1311. [CrossRef] [PubMed] [Google Scholar]
- Miyamoto T, Razavi S, DeRose R, Inoue T Synthesizing biomolecule-based Boolean logic gates. ACS Synth Biol 2013 ; 2 : 72–82. [Google Scholar]
- Frezza BM, Cockroft SL, Ghadiri MR Modular multi-level circuits from immobilized DNA-based logic gates. J Am Chem Soc 2007 ; 129 : 14875–14879. [Google Scholar]
- Daniel R, Rubens JR, Sarpeshkar R, Lu TK Synthetic analog computation in living cells. Nature 2013 ; 497 : 619–623. [CrossRef] [PubMed] [Google Scholar]
- Basu S, Gerchman Y, Collins CH, et al. A synthetic multicellular system for programmed pattern formation. Nature 2005 ; 434 : 1130–1134. [CrossRef] [PubMed] [Google Scholar]
- Garaschuk O, Griesbeck O, Konnerth A Troponin C-based biosensors : a new family of genetically encoded indicators for in vivo calcium imaging in the nervous system. Cell Calcium 2007 ; 42 : 351–361. [Google Scholar]
- French CE, de Mora K, Joshi N, et al. Synthetic biology and the art of biosensor design. In: Institute of medicine (US) forum on microbial threats. The science and applications of synthetic and systems biology : workshop summary. Washington (DC): National Academies Press (US), 2011 : A5. Available from : https://www.ncbi.nlm.nih.gov/books/NBK84465/. [Google Scholar]
- Kemmer C, Fluri DA, Witschi U, et al. A designer network coordinating bovine artificial insemination by ovulation-triggered release of implanted sperms. J Control Release 2011 ; 150 : 23–29. [CrossRef] [PubMed] [Google Scholar]
- Rabinovitch-Deere CA, Oliver JWK, Rodriguez GM, Atsumi S Synthetic biology and metabolic engineering approaches to produce biofuels. Chem Rev 2013 ; 113 : 4611–4632. [Google Scholar]
- Peralta-Yahya PP, Zhang F, del Cardayre SB, Keasling JD Microbial engineering for the production of advanced biofuels. Nature 2012 ; 488 : 320–328. [CrossRef] [PubMed] [Google Scholar]
- Canton B, Labno A, Endy D Refinement and standardization of synthetic biological parts and devices. Nat Biotechnol 2008 ; 26 : 787–793. [CrossRef] [PubMed] [Google Scholar]
- Knight T. Idempotent vector design for standard assembly of biobricks. In: MIT artificial intelligence laboratory. MIT Synthetic Biology Working Group, 2003. Available : http://hdl.handle.net/1721.1/21168. [Google Scholar]
- Endy D. BioBrick foundation website. http://biobricks.org/ [Google Scholar]
- Gardner TS, Cantor CR, Collins JJ Construction of a genetic toggle switch in Escherichia coli. Lett Nat 2000 ; 403 : 339–342. [Google Scholar]
- Richard N. Électronique numérique et séquentielle. Collection Sciences Sup. Paris : Dunod, 2002 : 264 p. [Google Scholar]
- Moon TS, Lou C, Tamsir A, et al. Genetic programs constructed from layered logic gates in single cells. Nature 2012 ; 7423 : 249–253. [Google Scholar]
- Mu M, Wieland M, Fussenegger M Programmable single-cell mammalian biocomputers. Nature 2012 ; 487 : 5–10. [Google Scholar]
- Wang B, Kitney RI, Joly N, Buck M Engineering modular and orthogonal genetic logic gates for robust digital-like synthetic biology. Nat Commun 2011 ; 2 : 508. [Google Scholar]
- Gendrault Y, Madec M, Lallement C, et al. Synthetic biology methodology and model refinement based on microelectronic modeling tools and languages. Biotechnol J 2011 ; 6 : 796–806. [CrossRef] [PubMed] [Google Scholar]
- Lux MW, Bramlett BW, Ball DA, Peccoud J Genetic design automation : engineering fantasy or scientific renewal? Trends Biotechnol 2012 ; 30 : 120–126. [CrossRef] [PubMed] [Google Scholar]
- MacDonald JT, Barnes C, Kitney RI, et al. Computational design approaches and tools for synthetic biology. Integr Biol (Camb) 2011 ; 3 : 97–108. [CrossRef] [PubMed] [Google Scholar]
- Hucka M. The systems biology markup language (SBML): a medium for representation and exchange of biochemical network models. Bioinformatics 2003 ; 19 : 524–531. [CrossRef] [PubMed] [Google Scholar]
- Cooling MT, Rouilly V, Misirli G, et al. Standard virtual biological parts : a repository of modular modeling components for synthetic biology. Bioinformatics 2010 ; 26 : 925–931. [CrossRef] [PubMed] [Google Scholar]
- Le Novère N, Bornstein B, Broicher A, et al. BioModels database : a free, centralized database of curated, published, quantitative kinetic models of biochemical and cellular systems. Nucleic Acids Res 2006 ; 34 : 689–691. [Google Scholar]
- Hoops S, Sahle S, Gauges R, et al. COPASI : a complex pathway simulator. Bioinformatics 2006 ; 22 : 3067–3074. [CrossRef] [PubMed] [Google Scholar]
- Bergmann FT, Sauro HM Comparing simulation results of SBML capable simulators. Bioinformatics 2008 ; 24 : 1963–1965. [CrossRef] [PubMed] [Google Scholar]
- Czar MJ, Cai Y, Peccoud J Writing DNA with GenoCAD. Nucleic Acids Res 2009 ; 37 : W40–W47. [CrossRef] [PubMed] [Google Scholar]
- Goler JA. BioJADE : a design and simulation tool for synthetic biological systems. PhD dissertation. Massachusetts Institute of Technology, 2004. [Google Scholar]
- Beal J, Weiss R, Densmore D, et al. TASBE : a tool-chain to accelerate synthetic biological engineering. Proceedings of the 3rd International Workshop on Bio-Design Automation 2011 ; 2 : 19–21. [Google Scholar]
- Nielsen AAK, Der BS, Shin J, et al. Genetic circuit design automation. Science 2016 ; 352 : aac7341. [CrossRef] [PubMed] [Google Scholar]
- Wang LT, Chang YW, Cheng KTT. Electronic design automation : synthesis, verification, and test. Morgan Kaufmann, 2009 : 972 p. [Google Scholar]
- Madec M, Pecheux F, Gendrault Y, et al. GeNeDA : an open-source workflow for the design automation of gene regulatory networks. J Comput Biol 2016 ; 23 : 841–855. [CrossRef] [PubMed] [Google Scholar]
- Thomas DE, Moorby PR. The Verilog® hardware description language New York : Springer, 2002 : 386 p. [Google Scholar]
- Bhatia S, Densmore D Pigeon : a design visualizer for synthetic biology. ACS Synth Biol 2013 ; 2 : 348–350. [Google Scholar]
- Gendrault Y, Madec M, Lallement C, Haiech J Modeling biology with HDL languages : a first step toward a genetic design automation tool inspired from microelectronics. IEEE Trans Biomed Eng 2014 ; 61 : 1231–1240. [Google Scholar]
- Rezgui A, Madec M, Lallement C, Haiech J. Integration of SBML models for the description of biological system in a lab-on-chip. 22nd International Conference Mixed Design of Integrated Circuits and Systems (MIXDES), 2015 : 165–170. [Google Scholar]
- Engvall E, Perlmann P Enzyme-linked immunosorbent assay (ELISA) quantitative assay of immunoglobulin G. Immunochemistry 1971 ; 8 : 871–874. [CrossRef] [PubMed] [Google Scholar]
- Guiton S, Rezgui A, Madec M, et al. Modeling and simulation of a Lab-On-Chip for micropollutants detection. Proceedings of the 21st International Conference Mixed Design of Integrated Circuits and Systems (MIXDES), 2014 : 256–261. [Google Scholar]
- Tamsir A, Tabor JJ, Voigt CA Robust multicellular computing using genetically encoded NOR gates and chemical wires. Nature 2011 ; 469 : 212–215. [CrossRef] [PubMed] [Google Scholar]
- Balagaddé FK, Song H, Ozaki J, et al. A synthetic Escherichia coli predator-prey ecosystem. Mol Syst Biol 2008 ; 4 : 187. [PubMed] [Google Scholar]
- Krencker JC, Kammerer JB, Hervé Y, Hébrard L. Direct electro-thermal simulation of integrated circuits using standard CAD tools. Thermal Investigations of ICs and Systems (Therminic), 16th International Workshop, 2010. [Google Scholar]
- Rosati E, Madec M, Kammerer JB, et al. Verilog-A compact space-dependent model for biology. Mixed design of integrated circuits and systems (MIXDES), 22nd International Conference, 2015. [Google Scholar]
- Képès F. Biologie synthétique et intégrative. Med Sci (Paris) 2009 ; 25 (hs2) : 39–42. [PubMed] [Google Scholar]
- Haiech J. Une vision de l’évolution de la biologie par un biologiste. D’une biologie descriptive vers une biologie prédictive. Med Sci (Paris) 2013 ; 29 (hs2) : 43–46. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Mazier D, Prix Thellier M Nobel de médecine, Youyou Tu, de Mao Zedong au Prix Nobel. Mec Sci (Paris) 2015 ; 2016 ; 32 : 106–109. [Google Scholar]
Liste des figures
 |
Figure 1. Schéma de passage de la description biologique (A) à un modèle électronique équivalent. À haut niveau d’abstraction, nous obtenons un schéma à base de portes logiques (C) qui correspond à une description mathématique à partir d’opérateurs booléens (B) . À bas niveau d’abstraction, nous obtenons un schéma électrique connectant des composants électriques standards et des transistors biologiques (E) , le tout correspondant à une description mathématique sous forme d’équations différentielles couplées (D) . GFP : green fluorescent protein ; I1/2 : inhibiteur 1/2 ; P1/2 : promoteur 1/2 ; R1/2 : facteur de transcription répresseur 1/2. |
| Dans le texte | |
 |
Figure 2. Résultats de synthèse pour une porte « ET » à 3 entrées et un séquenceur à 3 états. Dans le cas de la porte « ET » à 3 entrées, deux résultats de synthèse sont donnés : un dans le cas où la bibliothèque ne contient que des parts autorisant au plus 2 facteurs de transcription sur un promoteur (A) et un dans le cas où la bibliothèque contient des parts autorisant jusqu’à 3 facteurs de transcription par promoteurs (B) . Le séquenceur est quant à lui décrit en (C) . Il est constitué de 3 états : l’état cold que l’on atteint quand X est en défaut (matérialisé par les entrées A = 0 et B = 0) et dans lequel on synthétise davantage de X, l’état hot que l’on atteint quand X est en excès (matérialisé par A = 0 et B = 1) et dans lequel on synthétise une molécule Y dégradant X et un état idle dans lequel rien ne se passe. Le résultat de synthèse en limitant la bibliothèque à des parts à deux régulations est donné en (D) (d’après [40]). GFP : green fluorescent protein ; Op : opérateur. |
| Dans le texte | |
 |
Figure 3. Résultats de simulation obtenus sur un test de compétition par ELISA. A. Description du système biologique effectuée sous Virtual Cell. B. Comparaison entre les résultats de simulation obtenus avec Virtual Cell (points noirs) et le modèle VHDL-AMS (traits rouges). C. Simulation réalisée avec le modèle VHDL-AMS illustrant le test par compétition : plus la quantité d’antigènes non marqués (protéine cible) est faible dans l’échantillon (différentes courbes), plus il y a d’antigènes marqués (protéine cible couplée à une enzyme) fixés à l’anticorps à l’équilibre. D. Simulation du lab-on-chip complet intégrant le modèle VHDL-AMS précédemment discuté, un modèle de l’IsFET transformant les variations de pH en signal électrique et des modèles simplifiés écrits en VHDL-AMS pour les parties microfluidiques (d’après [44]). (2) : signifie que la réaction produit 2NH3 ; ELISA : enzyme-linked immunosorbent assay ; IsFET : ion-sensitive field-effect transistor ; VHDL-AMS : very high speed integrated circuits hardware description language-analog-mixed signal. |
| Dans le texte | |
 |
Figure 4. Résultats de simulation obtenus avec le simulateur spatio-temporel sur un système oscillant à deux cellules. Le système est décrit en A . Il est composé d’une cellule (1) fabriquant une protéine qui active un gène de la cellule (2), qui lui-même code pour une autre protéine qui réprime le gène de la cellule (1). Les nœuds du maillage obtenus sont représentés en B . Les résultats de simulation sont donnés sur les courbes : concentration d’activateur et du répresseur au niveau de la cellule (1) (C) , concentration d’activateur et du répresseur au niveau de la cellule (2) (D) . |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




