| Issue |
Med Sci (Paris)
Volume 31, Number 6-7, Juin–Juillet 2015
|
|
|---|---|---|
| Page(s) | 622 - 628 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20153106015 | |
| Published online | 07 July 2015 | |
Qu’en est-il de la biogenèse des ribosomes chez l’homme ?
About the ribosomal biogenesis in human
Laboratoire de biologie cellulaire, institut de recherche en biosciences, université de Mons, place du Parc 20, 7000
Mons, Belgique
*
lionel.tafforeau@umons.ac.be
Le ribosome est requis dans un mécanisme fondamental pour toute cellule, la traduction de l’information génétique en protéines. La biogenèse du ribosome est un processus très complexe qui nécessite un grand nombre d’étapes de maturation (synthèse, clivages et modifications des ARN ribosomiques précurseurs dans le nucléole, assemblage avec les protéines ribosomiques, export des sous-unités vers le cytoplasme). Des études récentes soulignent les limites de l’utilisation de la levure pour étudier la biogenèse du ribosome et appréhender la formation des ribosomes chez l’homme et, en particulier, les défauts de synthèse qui caractérisent les ribosomopathies.
Abstract
Ribosomes are cellular ribonucleoprotein particles required for a fundamental mechanism, translation of the genetic information into proteins. Ribosome biogenesis is a highly complex pathway involving many maturation steps: ribosomal RNA (rRNA) synthesis, rRNA processing, pre-rRNA modifications, its assembly with ribosomal proteins in the nuceolus, export of the subunit precursors to the nucleoplasm and the cytoplasm. Ribosome biogenesis has mainly being investigated in yeast during these last 25 years. However, recent works have shown that, despite many similarities between yeast and human ribosome structure and biogenesis, human pre-rRNA processing is far more complex than in yeast. In order to better understand diseases related to a malfunction in ribosome synthesis, the ribosomopathies, research should be conducted directly in human cells and animal models.
© 2015 médecine/sciences – Inserm

Le ribosome est un complexe ribonucléoprotéique (RNP) de très grande taille qui a les propriétés d’un ribozyme, c’est-à-dire que l’ARN ribosomique (ARNr) possède l’activité catalytique du complexe RNP. Le ribosome traduit l’information génétique codée par l’ARN messager (ARNm) en protéines. Il est constitué de deux sous-unités. La petite sous-unité (40S) porte l’activité de décodage de l’ARNm, elle est donc requise pour la reconnaissance codon/anti-codon (respectivement portés sur l’ARNm et l’ARNt [ARN de transfert]) et assure une traduction correcte de l’information génétique. La grande sous-unité (60S) est responsable de la formation de la liaison peptidique. C’est au sein du centre peptidyl-transférase que la chaîne des acides aminés est formée pour générer une protéine [33]. Le peptide naissant sort du ribosome par le tunnel de sortie, où il est pris en charge par des chaperons pour permettre le repliement de la protéine en cours de formation. Chaque ribosome est composé de 4 ARNr et de 80 protéines ribosomiques : la petite sous-unité est constituée de l’ARNr 18S et de 33 protéines, et la grande sous-unité de trois ARNr (5S, 5.8S et 28S) et de 47 protéines.
La biogenèse du ribosome est étudiée depuis plus de 25 ans chez la levure bourgeonnante Saccharomyces cerevisiae. Cet organisme modèle présente d’énormes avantages tant au niveau génétique (génération de mutants, criblages génétiques, premier génome eucaryote séquencé, etc.) que biochimique (purification de complexes protéiques et notamment de pré-ribosomes, etc.). À ce jour, environ 200 facteurs protéiques requis dans la biogenèse du ribosome ont été identifiés mais, paradoxalement, la fonction moléculaire de la plupart d’entre eux reste encore inconnue [1]. La structure tridimensionnelle des ribosomes chez les eucaryotes est très conservée. Elle a récemment été caractérisée chez la levure, la drosophile et l’homme [2, 3]. La biogenèse des ribosomes est également globalement conservée [4], de sorte que l’on a tendance à extrapoler le modèle de la biogenèse du ribosome décrit chez la levure aux eucaryotes supérieurs, dont l’homme. Même si elle est conservée, cette voie présente cependant de nombreuses différences entre la levure et l’homme, tant au niveau structurel qu’au niveau des acteurs requis [4–6]. Les ARNr matures humains sont de taille comparable à ceux de la levure, sauf pour l’ARNr 28S (humain) qui est 1,5 fois plus grand que l’ARNr 25S (levure). En revanche, les espaceurs externes (5’-ETS et 3’-ETS, 5’- et 3’-external transcribed sequences) et internes (ITS1 et 2, internal transcribed sequences), c’est-à-dire les extrémités flanquantes et les séquences séparant les 3 ARNr matures 18S, 5.8S et 25S/28S, sont jusqu’à 5 fois plus longs chez l’homme (Figure 2A). Cela sous-entend une régulation plus complexe des voies de maturation des ARNr (voir plus loin).
Le nucléole, site principal de la biogenèse des ribosomes, est bicompartimentalisé chez les levures et tricompartimentalisé chez l’homme, ce qui démontre une complexification structurelle de la synthèse des ribosomes [7]. De plus, le protéome nucléolaire est beaucoup plus fourni chez les mammifères : le nucléole contiendrait environ 200 protéines nucléolaires chez la levure, mais jusqu’à 4 500 chez l’homme [8, 9].
La biogenèse du ribosome est intimement liée à la régulation du cycle cellulaire. Elle est donc également liée à la transformation cellulaire et au cancer chez les eucaryotes supérieurs. Ainsi, lorsque la biogenèse du ribosome est affectée, un stress nucléolaire va, entre autres, activer le suppresseur de tumeur p53 qui régule le cycle cellulaire et l’entrée de la cellule en apoptose, mais aussi la synthèse des ribosomes [10]. Dans les cellules cancéreuses, il existe une régulation quantitative de la synthèse des ribosomes, mais également une régulation qualitative. En effet, on peut observer des variations dans la composition des constituants des ribosomes mais aussi dans leurs modifications, comme cela a été récemment prouvé [11–13].
La maturation des ribosomes chez l’homme
La biogenèse des ribosomes est un processus très complexe et hiérarchisé qui implique de nombreuses étapes, dont un grand nombre débutent dans le nucléole (Figure 1). Trois des quatre composants ARNr sont synthétisés dans le nucléole sous forme d’un grand ARN précurseur ; les protéines ribosomiques sont traduites dans le cytoplasme à partir d’ARNm transcrits par l’ARN polymérase II et sont importées dans le nucléole où elles vont s’assembler avec les ARNr naissants. Les ARNr précurseurs vont subir des réactions de clivages ainsi que des modifications chimiques conduisant à la formation des ARNr matures. Les protéines ribosomiques sont aussi modifiées post-traductionnellement (phosphorylation, ubiquitination, sumoylation, etc.). Les facteurs d’assemblage ainsi que les protéines ribosomiques s’assemblent aux ARNr précurseurs et les remodèlent petit à petit pour former les complexes ribonucléoprotéiques qui donneront les sous-unités 40S et 60S. L’assemblage des ARNr aux protéines ribosomiques et le remodelage des pré-sous-unités font intervenir de nombreuses ARN hélicases, ATPases et GTPases qui permettent de fournir l’énergie nécessaire à ces modifications moléculaires. Au cours de leur assemblage, les précurseurs des sous-unités ribosomiques transitent dans le nucléoplasme, puis sont exportés au travers du pore nucléaire dans le cytoplasme, où ils subissent leurs dernières étapes de maturation. Notons qu’il existe un système de contrôle qualité de la biogenèse des ribosomes, appelé la surveillance ARN. Elle a récemment fait l’objet d’une revue dans médecine/sciences [14], nous ne l’évoquerons pas dans cet article.
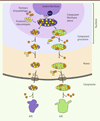 |
Figure 1. Représentation schématique de la biogenèse du ribosome. L’ARN ribosomique (ARNr) est transcrit dans le nucléole par l’ARN polymérase I à partir de l’ADN ribosomique (qui est présent en un grand nombre de copies sur les bras courts des chromosomes acrocentriques chez l’homme). Cette synthèse se réalise à l’interface du centre fibrillaire et du composant fibrillaire dense. L’ARNr précurseur primaire va s’associer avec les protéines ribosomiques et les facteurs d’assemblage, qui réaliseront les clivages, modifications et assemblages, aboutissant à la formation des ARNr matures, constituants des sous-unités ribosomiques. Chacune des deux sous-unités va subir sa propre maturation (pré-40S et pré-60S), transiter par le nucléoplasme et être exportée dans le cytoplasme par le pore nucléaire. Dans le cytoplasme, chaque sous-unité subit encore d’ultimes modifications. Les protéines ribosomiques restent associées aux ARNr ; en revanche, les facteurs d’assemblage sont recyclés pour la formation d’un autre ribosome. |
Synthèse des ARNr
l’ARNr est transcrit dans le nucléole par l’ARN polymérase I en un long précurseur, le pré-ARNr 47S (Figures 1 et 2A). Ce pré-ARNr contient les séquences de trois des quatre ARNr matures (les ARNr 18S, 5.8S et 28S) et va subir une succession de clivages endonucléolytiques et de digestions exonucléolytiques pour donner naissance aux ARNr matures (voir plus loin). L’ARNr 5S, troisième ARNr de la grande sous-unité ribosomique, est transcrit indépendamment par l’ARN polymérase III. Il subit une maturation à son extrémité 3’ et est incorporé dans le précurseur de la sous-unité 60S sous forme d’un complexe RNP avec les protéines ribosomiques (ribosomal protein) RPL5 et RPL11 [15].
Clivages des ARNr
Les séquences des ARNr matures 18S, 5.8S et 28S sont flanquées des espaceurs externes (5’-ETS et 3’-ETS) et internes (ITS1 et ITS2) (Figure 2A). Le transcrit primaire 47S est clivé aux sites 01 et 02, générant le précurseur (pré-ARNr) 45S (Figure 2B). Celui-ci est ensuite clivé selon deux voies alternatives, dépendant de l’ordre dans lequel les clivages sont réalisés. Dans la voie majoritaire, l’ARNr 45S est coupé au site 2, séparant les précurseurs destinés à la formation de la petite et de la grande sous-unité du ribosome, les pré-ARNr 30S et 32S, respectivement. Le clivage du pré-ARNr 30S aux sites A0 et 1 conduit à la formation du 26S et du 21S ; ces deux clivages ont lieu de manière concomitante, et le 26S est observé lorsqu’il y a découplage entre les deux clivages. Le précurseur 21S subit une maturation séquentielle médiée par l’exosome. On peut détecter un intermédiaire (21S-C) lors de la mutation de certains facteurs d’assemblage, notamment des composants de l’exosome [5, 16, 17]. Le 21S-C est coupé au site E, donnant le 18S-E, qui est alors clivé au site 3 pour générer l’ARNr mature 18S, constituant de la petite sous-unité. Ce dernier clivage a lieu dans le cytoplasme.
 |
Figure 2. Voies de clivages de l’ARN ribosomique précurseur 47S menant aux ARN ribosomiques matures 18S, 5.8S et 28S. A. Séquence de l’ARNr précurseur primaire 47S. Les séquences des ARNr matures sont bordées en 5’ et 3’ des espaceurs externes 5’-ETS et 3’-ETS, et flanquées des espaceurs internes ITS1 et ITS2. Les différents sites de clivages endonucléolytiques répertoriés sont positionnés sur le précurseur primaire. B. Voies de clivages menant aux ARNr matures. Deux voies coexistent dans les cellules humaines, dont la différence réside uniquement dans l’ordre des clivages. Dans la voie majeure (70 % des clivages dans le type cellulaire HeLa), le précurseur 45S est d’abord coupé au site 2, séparant les ARN précurseurs menant à l’ARNr de la petite sous-unité de ceux de la grande sous-unité (30S et 32S respectivement). Dans la voie mineure (30 % des clivages dans les cellules HeLa), l’ARN précurseur 45S est clivé dans le 5’-ETS aux sites A0 et 1, générant l’ARNr 41S. Celui-ci est ensuite clivé au site 2, générant le pré-ARNr 21S et le pré-ARNr 32S. Leurs clivages se réalisent alors comme dans la voie majoritaire, pour mener aux ARNr matures 18S, 5.8S et 28S (voir texte). C. Clivage alternatif du précurseur ARNr 41S au site E plutôt qu’au site 2 dans l’ITS1. Ce clivage alternatif produit l’ARNr 18S-E, précurseur direct de l’ARNr de la petite sous-unité et de l’ARNr 36S. Ce dernier subit une digestion exonucléolytique en 5’, produisant le précurseur 36S-C. Il est ensuite clivé au site 2, menant à l’ARNr 32S, dont les étapes de maturation sont décrites en B . |
De la même manière que pour le 21S, le pré-ARNr 18S-E subit un rognage 3’-5’ préalablement au clivage au site 3 [18]. D’autre part, le pré-ARNr 32S est clivé au site 3’ dans l’ITS2, générant l’ARNr mature 28S et le pré-ARNr 12S. Ce dernier va subir une série de digestions exonucléolytiques médiées par l’exosome, dont plusieurs formes intermédiaires sont détectables : le pre-ARNr 7S et le pre-ARNr 5.8S + 40. Ces rognages aboutissent finalement à l’ARNr mature 5.8S.
Dans la voie minoritaire, les clivages dans le 5’-ETS (sites A0 et 1) s’effectuent en premier, générant le pré-ARNr 41S à partir du pré-ARNr 45S. Un précurseur intermédiaire, le 43S, peut être observé lorsqu’il y a découplage entre ces deux clivages. Le pré-ARNr 41S est ensuite coupé au site 2, générant les pré-ARNr 21S et 32S (Figure 2B). Ces derniers suivent alors les étapes de clivage décrites précédemment.
Dans certaines conditions, le clivage au niveau de l’ITS1 se produit au site E plutôt qu’au site 2 [17, 18], générant alors le pré-ARNr 18S-E et un précurseur appelé 36S (Figure 2C). Ce clivage alternatif n’a lieu que lorsque l’espaceur 5’-ETS a préalablement été enlevé, au niveau du pré-ARNr 41S. Le 36S subit une digestion exonucléolytique à son extrémité 5’, formant le 36S-C. Il sera ensuite clivé au site 2 pour produire le 32S, qui suit alors les voies de clivages décrites précédemment. Notons que, comme chez la levure, il existe dans les cellules humaines deux formes d’ARNr matures 5.8S : la forme courte et la forme longue (5.8SS et 5.8SL).
Modifications des ARNr
Les pré-ARNr des deux sous-unités subissent de nombreuses modifications au cours de leur maturation. À ce jour, 95 isomérisations d’uridines (pseudo-uridylation), 105 méthylations de riboses et 10 méthylations de bases nucléotidiques ont été identifiées sur l’ARNr humain [19]. La majeure partie de ces modifications sont localisées à proximité immédiate des sites fonctionnels du ribosome. Les pseudo-uridylations et les méthylations des riboses sont synthétisées par des petits complexes RNP contenant des snoARN (small nucleolar RNA). On en distingue deux classes selon leurs structures et leurs motifs conservés : les snoARN à boîte H/ACA1 et les snoARN à boîte C/D2 (Figure 3). Ils ne possèdent pas d’activité catalytique intrinsèque, mais servent de guide pour la fibrillarine et la dyskérine, les protéines respectivement responsables des réactions de méthylation et de pseudo-uridylation. En effet, la position à modifier dans l’ARNr cible est spécifiée par la complémentarité des petites séquences anti-sens localisées dans les snoARN [20]. En revanche, les méthylations des bases nucléotidiques sont, quant à elles, réalisées par des enzymes ne nécessitant pas la présence de snoARN. Chez l’homme, 5 bases de l’ARNr 18S et 5 bases de l’ARNr 28S sont méthylées. Les enzymes responsables de ces modifications sont partiellement identifiées, essentiellement sur la base de ce qui est connu chez la levure S. cerevisiae.
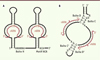 |
Figure 3. Structure des snoARN à boîte H/ACA (A) et à boîte C/D (B). Les séquences conservées sont présentées en gris et l’ARNr cible en rouge. La position du résidu qui va être modifié est indiquée : pseudouridylation, R¬ ( A ) et méthylation de ribose, cercle rouge ( B ). Adapté de [20]. |
Export des pré-ribosomes
Les précurseurs des sous-unités ribosomiques doivent être transportés dans le cytoplasme pour les dernières étapes de maturation, menant aux sous-unités du ribosome fonctionnel (Figure 1). Leur export est très régulé. Seuls les précurseurs arrivés à une étape de maturation suffisante peuvent être transportés dans le cytoplasme. Un système de contrôle de cette maturation est assuré en partie par l’assemblage de protéines ribosomiques. Ainsi, la petite sous-unité n’est exportée du noyau que lorsque les protéines RPS15/RPS18 (selon la nouvelle nomenclature, uS19/uS13, [21]) et RPS5/RPS28 (eS25/eS28) sont assemblées sur la particule [22, 23].
Certains facteurs d’assemblage servent d’adaptateurs pour recruter les exportines et ont ainsi un rôle dans le contrôle de qualité. Dans le cas des précurseurs de la grande sous-unité, c’est la protéine NMD3 qui recrute l’exportine CRM1 (chromosome region maintenance 1; also referred to as exportin1 or Xpo1) [24], et, pour les pré-40S, ce sont les facteurs d’assemblage RIOK2 (RIO kinase 2) et TSR1 qui seraient requis pour lier CRM1 [16, 25]. L’interaction entre CRM1 et les sous-unités ribosomiques va permettre leur passage au travers des pores nucléaires, par le biais de RAN-GTP [24]. D’autres karyophérines sont requises dans le transport nucléocytoplasmique du ribosome, notamment le complexe TAP/p15 (Mex67/Mtr2 chez la levure), qui est l’exportine principale des ARNm [26].
Dans le cytoplasme, les précurseurs des sous-unités ribosomiques subissent un nouveau contrôle de qualité. Les facteurs d’assemblage des dernières étapes de maturation bloquent les interactions entre les sous-unités d’une part et entre les sous-unités et les ARNm et ARNt d’autre part. Ce ne sera qu’une fois leur fonction effectuée que ces facteurs seront relâchés des sous-unités ribosomiques et que la traduction pourra avoir lieu (pour une revue récente, voir [27]).
Les acteurs requis dans les clivages de l’ARNr
Petits ARN nucléolaires (snoARN) impliqués dans les clivages
SnoARN U3 : ce petit ARN de type boîte C/D forme le cœur du complexe appelé « SSU processome », nécessaire aux clivages menant à l’ARNr de la petite sous-unité. Chez la levure, le SSU processome est requis pour les clivages A0, A1 et A2. Chez les eucaryotes supérieurs, il se lie au précurseur primaire et est requis pour les clivages aux sites 01, A0 et 1, situés dans le 5’-ETS. Le rôle du snoARN U3 a notamment été bien décrit chez le xénope [20].
SnoARN U8, U13 et U22 : ces ARN nucléolaires sont aussi des snoARN à boîte C/D. Ils sont spécifiques aux métazoaires. Le snoARN U8 est requis dans les clivages menant à la production de l’ARNr mature 5.8S et l’assemblage des ARNr 5.8S et 28S. Il est également impliqué dans le clivage en position 3’ du précurseur primaire. Le snoARN U13 pourrait être impliqué dans la formation de l’extrémité 3’ de l’ARNr 18S. Le snoARN U22 est nécessaire aux clivages menant à la maturation de l’ARNr 18S chez le xénope [20].
SnoARN U17 : ce petit ARN nucléolaire contient des motifs H/ACA. Il est requis pour le clivage de l’ARNr 18S chez la levure (snR30), le xénope et la souris.
Facteurs protéiques d’assemblage
Jusqu’il y a deux ans, quelques dizaines de facteurs impliqués dans la maturation des ARNr étaient identifiés chez l’homme, la plupart essentiellement par homologie avec un facteur de clivage décrit chez la levure. Dans le but d’identifier de nouvelles protéines requises dans la biogenèse des ribosomes et, plus particulièrement, dans le clivage des ARNr, nous avons réalisé un criblage dans une lignée cellulaire humaine (les cellules HeLa) en inactivant la fonction de 625 gènes codant des protéines nucléolaires par la méthode de l’interférence à ARN (siARN) [5]. Ces protéines ont été choisies car elles sont les protéines nucléolaires les plus abondantes. Elles ont été identifiées par purification et analyse protéomique des nucléoles. Les ARN ont été analysés par northern-blot et l’ensemble des principaux précurseurs des ARNr examinés (jusqu’à l’ARNr 12S). Nous avons identifié 286 protéines nécessaires à la biogenèse du ribosome. Parmi celles-ci, 153 ont un homologue impliqué dans la biogenèse des ribosomes chez la levure. Ce résultat était attendu puisque la formation des ribosomes est un mécanisme très conservé dans l’évolution. Cependant, environ un quart des protéines humaines présentent des fonctions supplémentaires, voire différentes de celles de leurs équivalents chez la levure. L’exemple le plus démonstratif est celui de l’exosome qui, outre son rôle dans la maturation de l’ITS2, est également requis dans la maturation de l’ITS1 chez l’homme. Parmi ces 286 protéines humaines, 59 ont un homologue chez la levure, mais leur fonction dans la formation des ribosomes n’a pas été démontrée. Enfin, 74 protéines n’ont pas d’homologue de séquence chez la levure. Parmi ces 286 facteurs humains requis dans la maturation des ARNr, 109 sont des biomarqueurs : ils sont liés à des maladies génétiques et/ou des cancers.
Nous avons montré que les défauts de clivage des ARNr observés dans les cellules HeLa sont majoritairement conservés dans d’autres types cellulaires comme les fibroblastes primaires WI-38 ou la lignée cellulaire HCT116 (provenant d’un carcinome du côlon). Nous avons également montré que les défauts de clivage observés sont directs, plutôt qu’indirects. Parmi les 625 gènes testés, plus de la moitié des inactivations n’induisent pas de phénotype et parmi celles qui induisent une modification des niveaux de précurseurs d’ARNr, on observe une large gamme d’effets. Nous avons d’autre part montré par une expérience de « time course » que les défauts de clivage sont détectables avant d’autres défauts influençant la survie de la cellule (tels que l’arrêt du cycle cellulaire, la stabilisation de la protéine p53 et l’entrée en apoptose). Une conclusion majeure de ce travail est que la voie menant à la formation des ribosomes est bien plus complexe chez l’homme que chez la levure. Le modèle établi chez cet organisme n’est donc plus suffisant pour expliquer ce processus chez l’homme.
L’ensemble des résultats de notre criblage sont consultables sur la base de données à l’adresse www.ribosomesynthesis.com.
Conclusion
La biogenèse du ribosome, processus fondamental pour toute cellule, comprend de nombreuses étapes très complexes. Elle est intimement liée aux voies de régulation cellulaire telles que les voies du métabolisme et du cycle cellulaire. Depuis quelques années, la biogenèse du ribosome est étudiée dans des lignées cellulaires humaines, notamment grâce à l’essor de l’interférence par ARN, qui permet d’inhiber l’expression d’une protéine d’intérêt dans des cellules en culture. Bien que le mécanisme de formation des ribosomes soit très conservé chez les eucaryotes, nous notons des différences notoires chez l’homme par rapport à la levure. Une nouvelle classe de maladies génétiques humaines, définie sous le terme de ribosomopathies, a récemment été décrite [28]. Ces maladies sont associées à des mutations dans des facteurs d’assemblage du ribosome ou dans des protéines ribosomiques. Elles sont caractérisées par des défauts de l’hématopoïèse et du développement, et un risque accru de développer un cancer. Citons à titre d’exemple la dyskératose congénitale3 [29, 34], l’anémie de Diamond-Blackfan4 [30, 31], le syndrome de Shwachman-Diamond5 [32]. Le modèle de la maturation du ribosome chez la levure n’est plus suffisant pour comprendre les implications de la biogenèse du ribosome dans ce type de maladies, même s’il s’avère encore très puissant dans le cadre d’analyses fines de l’effet des mutations de certains gènes ou d’interactions protéine-protéine. Comme notre criblage l’a montré, certains facteurs d’assemblage du ribosome présents chez l’homme n’ont pas d’homologues chez la levure. Il est donc nécessaire d’appréhender spécifiquement les mécanismes de synthèse du ribosome dans la cellule humaine.
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt avec les données publiées dans cet article.
« Le phénotype de la dyskératose congénitale est très variable d’un patient à l’autre et, pour un patient donné, évolutif au cours du temps. La forme typique est caractérisée par des atteintes cutanées, phanériennes et muqueuses. Les patients peuvent aussi développer un retard statural, des anomalies dentaires, une ostéoporose précoce [entre autres]. Au moins 80 % des patients développeront une pancytopénie secondaire à une aplasie médullaire. […]. Les mutations de quatre gènes sont associées à la dyskératose (DKC1, hTERC, hTERTet NOP10) codent des composants de la télomérase » (tiré de [34]).
L’anémie de Blackfan-Diamond ou érythroblastopénie congénitale, se présente comme une anémie arégénérative le plus souvent macrocytaire. L’anémie est découverte tôt dans la vie, en règle générale avant 2 ans. à ce jour, des mutations des gènes codant pour des protéines ribosomiques (RP) de la petite ou de la grande sous-unité du ribosome peuvent être identifiées chez 40 à 45 % des patients ; (source : orphanet) [31].
Le syndrome de Shwachman-Diamond associe une atteinte hématologique et un syndrome malformatif dont l’élément le plus constant est une lipomatose du pancréas responsable d’une insuffisance pancréatique externe. Cette maladie très rare a une transmission autosomique récessive ; des mutations du gène SBDS impliqué dans le métabolisme des ARN et en particulier dans les étapes finales de maturation de la sous-unité 60S, sont retrouvées chez la plupart des patients (> 95 %) (source : orphanet).
Références
- Woolford JL, Baserga SJ. Ribosome biogenesis in the yeast Saccharomyces cerevisiae. Genetics 2013 ; 195 : 643–681. [CrossRef] [PubMed] [Google Scholar]
- Ben-Shem A, Garreau de Loubresse N, Melnikov S, et al. The structure of the eukaryotic ribosome at 3.0 Å resolution. Science 2011 ; 334 : 1524–1529. [CrossRef] [PubMed] [Google Scholar]
- Anger AM, Armache JP, Berninghausen O, et al. Structures of the human and Drosophila 80S ribosome. Nature 2013 ; 497 : 80–85. [CrossRef] [PubMed] [Google Scholar]
- Mullineux ST, Lafontaine DLJ. Mapping the cleavage sites on mammalian pre-rRNAs: where do we stand? Biochimie 2012 ; 94 : 1521–1532. [CrossRef] [PubMed] [Google Scholar]
- Tafforeau L, Zorbas C, Langhendries JL, et al. The complexity of human ribosome biogenesis revealed by systematic nucleolar screening of pre-rRNA processing factors. Mol Cell 2013 ; 51 : 539–551. [CrossRef] [PubMed] [Google Scholar]
- Henras AK, Plisson-Chastang C, O’Donohue MF, et al. An overview of pre-ribosomal RNA processing in eukaryotes. Wiley Interdiscip Rev RNA 2014 ; doi : 10.1002/wrna.1269. [Google Scholar]
- Thiry M, Lafontaine DLJ. Birth of a nucleolus: the evolution of nucleolar compartments. Trends Cell Biol 2005 ; 15 : 194–199. [CrossRef] [PubMed] [Google Scholar]
- Huh W-K, Falvo JV, Gerke LC, et al. Global analysis of protein localization in budding yeast. Nature 2003 ; 425 : 686–691. [CrossRef] [PubMed] [Google Scholar]
- Ahmad Y, Boisvert FM, Gregor P, et al. NOPdb: Nucleolar proteome database - 2008 update. Nucleic Acids Res 2009 ; 37 : D181–D184. [CrossRef] [PubMed] [Google Scholar]
- Ruggero D. Translational control in cancer etiology. Cold Spring Harb Perspect Biol 2013 ; 5 : a012336. [CrossRef] [PubMed] [Google Scholar]
- Marcel V, Catez F, Mertani HC, et al. Le ribosome. Med Sci (Paris) 2014 ; 30 : 21–24. [CrossRef] [EDP Sciences] [Google Scholar]
- Marcel V, Ghayad SE, Belin S, et al. p53 Acts as a safeguard of translational control by regulating fibrillarin and rRNA methylation in cancer. Cancer Cell 2013 ; 24 : 318–330. [CrossRef] [PubMed] [Google Scholar]
- Bellodi C, McMahon M, Contreras A, et al. H/ACA small RNA dysfunctions in disease reveal key roles for noncoding RNA modifications in hematopoietic stem cell differentiation. Cell Rep 2013 ; 3 : 1493–1502. [CrossRef] [PubMed] [Google Scholar]
- Leporé N, Lafontaine DLJ. « Attrape-moi si tu peux » - Surveillance de l’intégrité structurale et fonctionnelle des ARN cellulaires eucaryotes. Med Sci (Paris) 2010 ; 26 : 259–266. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Ciganda M, Williams N. Eukaryotic 5S rRNA biogenesis. Wiley Interdiscip Rev RNA 2011 ; 2 : 523–533. [CrossRef] [PubMed] [Google Scholar]
- Carron C, O’Donohue MF, Choesmel V, et al. Analysis of two human pre-ribosomal factors, bystin and hTsr1, highlights differences in evolution of ribosome biogenesis between yeast and mammals. Nucleic Acids Res 2011 ; 39 : 280–291. [CrossRef] [PubMed] [Google Scholar]
- Sloan KE, Mattijssen S, Lebaron S, et al. Both endonucleolytic and exonucleolytic cleavage mediate ITS1 removal during human ribosomal RNA processing. J Cell Biol 2013 ; 200 : 577–588. [CrossRef] [PubMed] [Google Scholar]
- Preti M, O’Donohue MF, Montel-Lehry N, et al. Gradual processing of the ITS1 from the nucleolus to the cytoplasm during synthesis of the human 18S rRNA. Nucleic Acids Res 2013 ; 41 : 4709–4723. [CrossRef] [PubMed] [Google Scholar]
- Decatur WA, Fournier MJ. rRNA modifications and ribosome function. Trends Biochem Sci 2002 ; 27 : 344–351. [CrossRef] [PubMed] [Google Scholar]
- Watkins NJ, Bohnsack MT. The box C/D and H/ACA snoRNPs: key players in the modification, processing and the dynamic folding of ribosomal RNA. Wiley Interdiscip Rev RNA 2011 ; 3 : 397–414. [CrossRef] [PubMed] [Google Scholar]
- Ban N, Beckmann R, Cate JH, et al. A new system for naming ribosomal proteins. Curr Opin Struct Biol 2014 ; 24 : 165–169. [CrossRef] [PubMed] [Google Scholar]
- Ferreira-Cerca S, Pöll G, Gleizes PE, et al. Roles of eukaryotic ribosomal proteins in maturation and transport of pre-18S rRNA and ribosome function. Mol Cell 2005 ; 20 : 263–275. [CrossRef] [PubMed] [Google Scholar]
- Léger-Silvestre I, Milkereit P, Ferreira-Cerca S, et al. The ribosomal protein Rps15p is required for nuclear exit of the 40S subunit precursors in yeast. EMBO J 2004 ; 23 : 2336–2347. [CrossRef] [PubMed] [Google Scholar]
- Trotta CR, Lund E, Kahan L, et al. Coordinated nuclear export of 60S ribosomal subunits and NMD3 in vertebrates. EMBO J 2003 ; 22 : 2841–2851. [CrossRef] [PubMed] [Google Scholar]
- Zemp I, Wild T, O’Donohue MF, et al. Distinct cytoplasmic maturation steps of 40S ribosomal subunit precursors require hRio2. J Cell Biol 2009 ; 185 : 1167–1180. [CrossRef] [PubMed] [Google Scholar]
- Yao W, Roser D, Köhler A, et al. Nuclear export of ribosomal 60S subunits by the general mRNA export receptor Mex67-Mtr2. Mol Cell 2007 ; 26 : 51–62. [CrossRef] [PubMed] [Google Scholar]
- Karbstein K. Quality control mechanisms during ribosome maturation. Trends Cell Biol 2013 ; 23 : 242–250. [CrossRef] [PubMed] [Google Scholar]
- Narla A, Ebert BL. Ribosomopathies: human disorders of ribosome dysfunction. Blood 2010 ; 115 : 3196–3205. [CrossRef] [PubMed] [Google Scholar]
- Ruggero D, Grisendi S, Piazza F, et al. Dyskeratosis congenita and cancer in mice deficient in ribosomal RNA modification. Science 2003 ; 299 : 259–262. [CrossRef] [PubMed] [Google Scholar]
- Choesmel V, Bacqueville D, Rouquette J, et al. Impaired ribosome biogenesis in Diamond-Blackfan anemia. Blood 2007 ; 109 : 1275–1283. [CrossRef] [PubMed] [Google Scholar]
- Aguissa-Touré AH, Da Costa L, Leblanc T, et al. Anémie de Diamond-Blackfan. Med Sci (Paris) 2009 ; 25 : 69–76. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Johnson AW, Ellis SR. Of blood, bones, and ribosomes: is Swachman-Diamond syndrome a ribosomopathy? Genes Dev 2011 ; 25 : 898–900. [CrossRef] [PubMed] [Google Scholar]
- Kevin Macé K, Giudice E, Gillet R. La synthèse des protéines par le ribosome. Un chemin semé d’embûches. Med Sci (Paris) 2015 ; 31 : 282–290. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Hoareau-Aveilla C, Henry Y. Leblanc TLa dyskératose congénitale. Une maladie méconnue due à un maintien défectueux des télomères. Med Sci (Paris) 2008 ; 24 : 390–398. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Représentation schématique de la biogenèse du ribosome. L’ARN ribosomique (ARNr) est transcrit dans le nucléole par l’ARN polymérase I à partir de l’ADN ribosomique (qui est présent en un grand nombre de copies sur les bras courts des chromosomes acrocentriques chez l’homme). Cette synthèse se réalise à l’interface du centre fibrillaire et du composant fibrillaire dense. L’ARNr précurseur primaire va s’associer avec les protéines ribosomiques et les facteurs d’assemblage, qui réaliseront les clivages, modifications et assemblages, aboutissant à la formation des ARNr matures, constituants des sous-unités ribosomiques. Chacune des deux sous-unités va subir sa propre maturation (pré-40S et pré-60S), transiter par le nucléoplasme et être exportée dans le cytoplasme par le pore nucléaire. Dans le cytoplasme, chaque sous-unité subit encore d’ultimes modifications. Les protéines ribosomiques restent associées aux ARNr ; en revanche, les facteurs d’assemblage sont recyclés pour la formation d’un autre ribosome. |
| Dans le texte | |
 |
Figure 2. Voies de clivages de l’ARN ribosomique précurseur 47S menant aux ARN ribosomiques matures 18S, 5.8S et 28S. A. Séquence de l’ARNr précurseur primaire 47S. Les séquences des ARNr matures sont bordées en 5’ et 3’ des espaceurs externes 5’-ETS et 3’-ETS, et flanquées des espaceurs internes ITS1 et ITS2. Les différents sites de clivages endonucléolytiques répertoriés sont positionnés sur le précurseur primaire. B. Voies de clivages menant aux ARNr matures. Deux voies coexistent dans les cellules humaines, dont la différence réside uniquement dans l’ordre des clivages. Dans la voie majeure (70 % des clivages dans le type cellulaire HeLa), le précurseur 45S est d’abord coupé au site 2, séparant les ARN précurseurs menant à l’ARNr de la petite sous-unité de ceux de la grande sous-unité (30S et 32S respectivement). Dans la voie mineure (30 % des clivages dans les cellules HeLa), l’ARN précurseur 45S est clivé dans le 5’-ETS aux sites A0 et 1, générant l’ARNr 41S. Celui-ci est ensuite clivé au site 2, générant le pré-ARNr 21S et le pré-ARNr 32S. Leurs clivages se réalisent alors comme dans la voie majoritaire, pour mener aux ARNr matures 18S, 5.8S et 28S (voir texte). C. Clivage alternatif du précurseur ARNr 41S au site E plutôt qu’au site 2 dans l’ITS1. Ce clivage alternatif produit l’ARNr 18S-E, précurseur direct de l’ARNr de la petite sous-unité et de l’ARNr 36S. Ce dernier subit une digestion exonucléolytique en 5’, produisant le précurseur 36S-C. Il est ensuite clivé au site 2, menant à l’ARNr 32S, dont les étapes de maturation sont décrites en B . |
| Dans le texte | |
 |
Figure 3. Structure des snoARN à boîte H/ACA (A) et à boîte C/D (B). Les séquences conservées sont présentées en gris et l’ARNr cible en rouge. La position du résidu qui va être modifié est indiquée : pseudouridylation, R¬ ( A ) et méthylation de ribose, cercle rouge ( B ). Adapté de [20]. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




