Figure 2.
Télécharger l'image originale
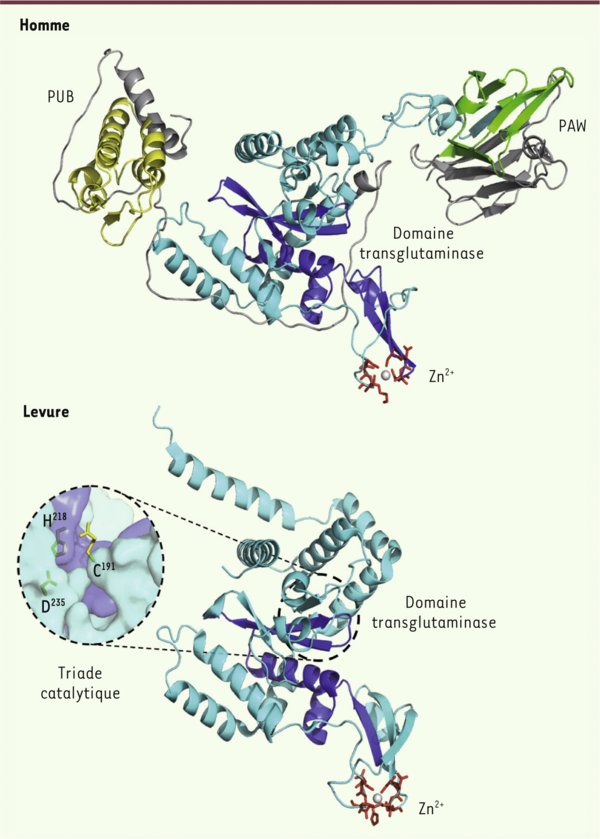
Comparaison des structures tridimensionnelles des PNGases de levure et humaine. La PNGase de Saccharomyces cerevisiae (ScPng1p) est codée par le gène PNG1 : le domaine central transglutaminase, qui comporte le site actif (en bleu foncé), et les deux motifs thioredoxin-like CXXC de liaison au zinc (dont les acides aminés sont indiqués en rouge), constituent une structure hautement conservée qui est commune à toutes les espèces. Un agrandissement du site actif de ScPng1p montre que la triade catalytique (C191, H218 et D235) se situe au fond d’une structure qui a la forme d’un sillon. Le modèle tridimensionnel de la PNGase humaine (HsNgly1p) a été construit à l’aide du logiciel Modeller v9.11 (http://salilab.org/modeller/) en se basant sur l’homologie de séquence des différents domaines de HsNgly1p, de l’enzyme murine (PUB, pdb 2CCQ et 2D5U ; PAW, pdb 2C9G) et du complexe murin PNGaseI/HR23 (pdb 2F4M). La PNGase de mammifère est codée par le gène NGLY1 : en plus du domaine transglutaminase, elle comporte, respectivement, aux extrémités amino- et carboxy-terminales, le domaine PUB (en jaune, domaine d’interaction protéique) et le domaine PAW (en vert, domaine de liaison aux résidus de mannose).
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




