| Issue |
Med Sci (Paris)
Volume 29, Number 3, Mars 2013
|
|
|---|---|---|
| Page(s) | 301 - 307 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2013293017 | |
| Published online | 27 March 2013 | |
Fonction des suppresseurs de tumeur PALB2 et BRCA2 dans la réparation des cassures double-brin de l’ADN
Functions of PALB2 and BRCA2 tumor suppressors in DNA double-strand break repair
Genome stability laboratory, Laval university cancer research center, Hôtel-Dieu de Quebec research center (CHUQ), 9 McMahon, Québec, G1R 2J6, Canada
*
jean-yves.masson@crhdq.ulaval.ca
Le cancer est aujourd’hui la première cause de mortalité en France devant les maladies cardiovasculaires. Les dommages à l’ADN, comme les cassures double-brin, sont une source d’instabilité génique menant au développement des cancers. Il a en effet été clairement démontré que les mutations dans l’information génétique représentent l’événement initiateur. Les cellules possèdent plusieurs mécanismes pour réparer les cassures double-brin de l’ADN, mais la réparation par recombinaison homologue est le seul qui permet de les réparer de manière fidèle sans créer de mutations dans l’ADN. Nous décrivons ici les rôles de PALB2 et BRCA2, deux protéines clés pour le fonctionnement de la recombinaison homologue et le maintien de la stabilité du génome.
Abstract
Cancer is now the leading cause of mortality in France. It has been clearly demonstrated that mutations in the genetic information is the initiating event of cancer. DNA damage such as DNA double-strand breaks leads to genomic instability and cancer development. Cells can repair DNA double-strand breaks through several mechanisms. Nevertheless, only homologous recombination repair is faithful and repairs DNA without creating mutations. Here, we review the roles of PALB2 and BRCA2 in homologous recombination and genome stability.
© 2013 médecine/sciences – Inserm / SRMS

Les mécanismes de réparation des cassures double-brin de l’ADN
Les cellules de notre organisme subissent en permanence l’attaque d’agents endogènes ou exogènes qui entraînent des dizaines de milliers de lésions de l’ADN. Celles de type endogène sont provoquées par la cellule elle-même, et leur nombre a été estimé à plus de 100 000 par jour [1]. Ces lésions sont créées, par exemple, lors de la réplication de l’ADN ou encore par l’action de métabolites réagissant avec la molécule d’ADN. Les lésions exogènes, quant à elles, sont produites par des sources physiques ou chimiques extérieures à la cellule. Les agents physiques, par exemple les radiations ionisantes, ou les agents chimiques génotoxiques, comme ceux qui sont utilisés lors des chimiothérapies anticancéreuses, créent une grande variété de lésions de l’ADN : modifications de bases, cassures simple-brin ou double-brin [1, 2]. Hormis les cassures physiologiques programmées, lors de la méiose par exemple, les cassures double-brin sont les lésions de l’ADN les plus dangereuses pour les cellules, parce qu’elles menacent directement l’intégrité du génome et doivent être rapidement détectées, puis réparées. Les cellules ont développé pour ce faire plusieurs mécanismes de réparation.
Une cassure double-brin de l’ADN peut être réparée via quatre principaux mécanismes : le NHEJ (non-homologous end joining), le NHEJ alternatif (alt-NHEJ), la recombinaison homologue (RH), ou le SSA (single-strand annealing) (Figure 1 et Encadré) [1]. La cellule choisit l’un ou l’autre de ces mécanismes en fonction de la phase du cycle cellulaire et du type de cassure. Les NHEJ ou alt-NHEJ sont actifs durant tout le cycle cellulaire, alors que la RH ou le SSA sont actifs seulement à la fin de la phase S et pendant la phase G2 [3]. Contrairement aux trois autres mécanismes, la RH est un mécanisme généralement fidèle de réparation de l’ADN. Dans cette revue, nous nous focaliserons plus particulièrement sur la réparation des cassures double-brin par recombinaison homologue.
 |
Figure 1. Mécanismes de réparation des cassures double-brin de l’ADN. |
NHEJ (non-homologous end joining)
Réparation par jonction d’extrémités non homologues permettant une religation directe des deux extrémités de l’ADN brisé (Figure 1B). Dans ce mécanisme, la cassure double-brin est détectée par le complexe Ku/DNA-PKcs (protein kinase, catalytic subunit), puis réparée par le complexe de ligation XLF-XRCC4 (X-ray repair complementing defective repair in chinese hamster cells-4)-ADN ligase IV. Dans certains cas, l’exo-endonucléase Artémis est recrutée pour permettre la dégradation des extrémités de l’ADN afin de les rendre compatibles avec la ligation. Toutefois, cette activité est responsable de l’absence de fidélité du NHEJ puisqu’elle provoque la perte de plusieurs bases d’ADN.
NHEJ alternatif (alt-NHEJ)
Mécanisme indépendant du NHEJ initié par une nucléase, vraisemblablement par le complexe MRN (Mre11/Rad50/Nbs1)-CtIP, afin de générer des extrémités d’ADN simple-brin. Cette activité permet de découvrir des séquences de part et d’autre de la cassure comportant des microhomologies (2-4 nucléotides), dont l’hybridation permettrait la ligation de l’ADN (Figure 1E).
SSA (single-strand annealing)
Réparation par appariement entre molécules d’ADN simple-brin consistant en l’hybridation directe, grâce à la présence de séquences homologues (Figure 1F), des ADN simple-brin générés lors de la résection des extrémités de la cassure. La protéine RPA (replication protein A) et la recombinase RAD52 sont au centre de ce mécanisme. Le SSA est principalement actif dans les régions du génome où les séquences sont dupliquées, ce qui représente environ 15 % du génome humain.
La recombinaison homologue
La RH utilise la chromatide sœur présente après la réplication de l’ADN ou, lors de la méiose, un chromosome homologue comme une matrice comportant l’information génétique intacte permettant de réparer l’ADN brisé. Elle est subdivisée en trois étapes : la phase présynaptique, la phase synaptique et la phase postsynaptique (Figure 1A). La recombinase RAD51, une protéine clé de ce processus, joue un rôle central au cours de ces trois phases. La phase présynaptique consiste en la résection des extrémités de la cassure pour former un ADN 3’-protubérant. RAD51 se lie alors sur l’ADN simple-brin, autour duquel elle forme un filament nucléoprotéique. Durant la phase synaptique, RAD51 provoque l’invasion de l’ADN 3’-protubérant dans l’ADN double-brin complémentaire de la chromatide sœur pour former une structure dite en D-loop (displacement loop ou boucle de déplacement). La phase postsynaptique consiste en l’extension de l’ADN envahissant par l’action d’une ADN polymérase afin de recopier l’information manquante [4, 5] (Figure 1).
Après l’étape d’extension, trois mécanismes différents peuvent être utilisés pour compléter la réparation : le DSBR, le SDSA ou le BIR. Dans la voie du DSBR (double-strand break repair), la seconde partie du brin brisé est capturée par les protéines RPA (replication protein A) et RAD52, puis associée au brin d’ADN déplacé, entraînant la formation de deux jonctions de Holliday1. La double jonction de Holliday (dHJ) est ensuite soit résolue, créant des produits d’ADN de type crossover ou non-crossover, soit dissoute pour donner naissance uniquement à des produits de type non-crossover (Figure 1C). Dans la voie du SDSA (synthesis-dependent strand annealing), la D-loop est dissoute avant la formation de la dHJ pour s’hybrider avec l’ADN simple-brin de la deuxième extrémité de la cassure. Ce mécanisme forme uniquement des produits d’ADN de type non-crossover (Figure 1D). Dans la troisième voie appelée BIR (break-induced replication), la structure D-loop se convertit en fourche de réplication pour recopier le bras entier du chromosome (Figure 1G). La voie du SDSA est prédominante dans la réparation par recombinaison homologue de l’ADN, tandis que la voie du BIR est la moins fréquente. Cette dernière est principalement utilisée lorsqu’il y a perte de la deuxième extrémité de la cassure, ou lors du processus de rallongement des télomères dans les cellules déficientes pour la télomérase [3–5].
Régulation de la recombinaison homologue par les médiateurs BRCA2 et PALB2
Lors de la phase présynaptique, l’ADN simple-brin formé à la suite de la résection de la cassure double-brin est dans un premier temps lié par la protéine RPA. La liaison de RPA protège l’ADN des attaques de nucléases et empêche la formation de structures secondaires ou la liaison de protéines inappropriées. RAD51 identifie ensuite un des segments d’ADN brisé, forme un filament nucléoprotéique, puis recherche dans le génome une séquence identique à celle qui a été endommagée [31] (→). Ce filament d’ADN-RAD51 est l’élément central pour une réparation fidèle de la cassure double-brin. Pour ce faire, RAD51 s’oligomérise autour de l’ADN simple-brin pour former ce long filament hélicoïdal. Le filament a une conformation constante : un tour d’hélice du filament de RAD51 est constitué de six monomères, chacun liant trois bases d’ADN. L’association des monomères pour former ce nucléofilament requiert la liaison à l’ATP, dont l’hydrolyse toutefois n’est pas nécessaire [6]. La formation du filament de RAD51 nécessite le recrutement de cofacteurs afin notamment de déplacer RPA qui empêche la liaison de RAD51 sur l’ADN. Lors de la phase synaptique, ces mêmes cofacteurs stimulent son activité recombinase permettant ainsi l’étape d’invasion [4, 5]. Ces cofacteurs sont des protéines appelées « médiateurs de la recombinaison homologue ». Chez l’humain, les protéines PALB2 (partner and localizer of BRCA2) et BRCA2 (breast cancer 2 protein) possèdent ces deux propriétés.
(→) Voir m/s 2012, vol. 28, n° 8-9, page 714
BRCA2 régule RAD51 pour réparer les CDB
Le gène BRCA2, localisé sur le chromosome 13q12-q13, a été identifié en 1995 comme étant un gène de susceptibilité au cancer du sein initialement, puis au cancer des ovaires, de la prostate ou encore du pancréas [7]. Entre 5 et 7 % des cancers du sein sont provoqués par une mutation dans le gène BRCA2. Une personne possédant une mutation monoallélique de ce gène possède un risque de 50 % de développer un cancer avant l’âge de 50 ans, et de 80 % avant l’âge de 70 ans. Des mutations hypomorphes bialléliques dans BRCA2 entraînent le développement de l’anémie de Fanconi, une maladie génétique rare, causée par des mutations dans les gènes régulant la réparation des pontages interbrins de l’ADN [7].
La protéine humaine BRCA2 est une protéine de grande taille de 3 418 acides aminés (384 kDa), qui est essentielle au fonctionnement de la recombinaison homologue [3, 7].
Cette dernière est diminuée de 80 à 90 % en son absence. BRCA2 lie l’ADN simple-brin et, avec une plus faible affinité, l’ADN double-brin. La région de BRCA2 qui se lie à l’ADN est composée de cinq domaines [8] :un domaine en hélice (HD), trois domaines de liaison aux oligonucléotides (OB1, OB2 et OB3) qui lient l’ADN simple-brin, et un domaine en tour (tower) qui possède un sous-domaine en hélice-tour-hélice (HTH) responsable de l’activité de liaison à l’ADN double-brin (Figure 2). Les domaines HD et OB1 sont également associés avec une autre protéine, appelée DSS1, importante pour la stabilité de BRCA2.
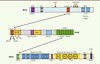 |
Figure 2. Organisation des domaines fonctionnels des protéines BRCA1, PALB2 et BRCA2. Les protéines interagissant avec BRCA1, PALB2 ou BRCA2 sont indiquées en dessous des domaines correspondants. Les flèches indiquent les interactions entre BRCA1, PALB2 et BRCA2. Les sites de phosphorylation sont indiqués par les cercles roses. |
Le domaine de liaison à l’ADN de BRCA2 est important pour le recrutement de RAD51 aux cassures double-brin, qui est un des rôles majeurs de BRCA2. Par son interaction directe avec BRCA2 (une molécule de BRCA2 est capable de lier six molécules de RAD51 [9]), RAD51 est transportée dans le noyau de la cellule puis aux cassures double-brin de l’ADN [10]. BRCA2 interagit avec RAD51 par deux domaines distincts : son domaine central composé de huit motifs BRC (environ 30 acides aminés) et son domaine carboxy-terminal. RAD51 lie préférentiellement les motifs BRC 1, 2, 3 et 4, alors que les motifs BRC 5, 6, 7 et 8 lient plutôt le complexe RAD51-ADN [11]. Les fonctions exactes de ces répétitions de BRC sont encore imprécises. Dans les cellules, il a été montré que la surexpression de BRC4 empêche la localisation de RAD51 aux cassures double-brin [12]. In vitro, BRC3 et BRC4 détruisent le filament de RAD51 [10]. Néanmoins, cet effet ne s’observe que si la molarité de BRC est très supérieure à celle de RAD51. À plus faible molarité, les motifs BRC3 ou BRC4 lient le filament de RAD51 et forment avec lui un complexe stable [13]. Récemment, deux autres études proposent que BRC4 ou BRC1-8 stimulent la formation du filament de RAD51 sur l’ADN simple-brin, ainsi que son activité d’échange de brin. L’interaction de BRC4 avec RAD51 diminue également l’activité d’hydrolyse de l’ATP de cette dernière (nécessaire au désassemblage du filament). À l’inverse, BRC4 empêche la formation du filament de RAD51 sur l’ADN double-brin [11]. Ce résultat est confirmé in vivo par une autre étude montrant que l’expression, dans des cellules déficientes en BRCA2, des motifs BRC3 ou BRC4 fusionnés au domaine de liaison à l’ADN de RPA (pour permettre leur localisation à la cassure indépendamment de BRCA2) restaure les foyers de RAD51, suggérant que BRC3 et BRC4 sont importants pour la stimulation du filament de RAD51 [14]. L’équilibre entre la stimulation de la formation et la déstabilisation du filament de RAD51 est donc probablement finement régulé par les motifs BRC de BRCA2.
Le deuxième domaine d’interaction avec RAD51 est situé sur l’extrémité carboxy-terminale de BRCA2 et, plus exactement, au niveau d’un petit motif nommé TR2 qui est phosphorylé sur la sérine 3291 (Figure 2) [15]. La forme phosphorylée de TR2 est incapable de lier RAD51 mais, en présence de cassures double-brin dans la cellule, la sérine 3291 est déphosphorylée, ce qui provoque son interaction avec RAD51 [15] et la protection de son filament contre l’activité destructrice du motif BRC4 [16, 17]. Une autre étude a montré que BRCA2 protège les brins d’ADN néosynthétisés lorsque la progression des fourches de réplication est perturbée. Le domaine TR2 joue un rôle important dans cette activité en protégeant l’ADN nouvellement formé de la dégradation par MRE11 (meiotic recombination 11 homolog 1) [18]. Toutes ces observations indiquent que BRCA2, par ses domaines BRC et TR2, contrôle la formation du filament de RAD51 pour la réparation des cassures double-brin ou le redémarrage des fourches de réplication, puis son désassemblage pour permettre la poursuite du cycle cellulaire [19].
Rôle des interactions protéiques de PALB2 et de sa dimérisation dans la réparation des cassures double-brin
La protéine PALB2 a été identifiée en 2006 en immunoprécipitant la protéine BRCA2 [20]. Le gène PALB2 est localisé sur le chromosome 16p12.2 et consiste en 13 exons qui codent pour une protéine de 1186 acides aminés (131 kDa). Une mutation hétérozygote dans le gène PALB2 est à l’origine de prédispositions au cancer du sein et du pancréas, alors que des mutations homozygotes, comme pour le gène BRCA2, causent l’anémie de Fanconi [21].
PALB2 interagit avec BRCA2 via un domaine constitué de sept motifs WD40 situé en carboxy-terminal de la protéine. BRCA2, quant à elle, interagit avec PALB2 grâce à son domaine situé en amino-terminal. L’absence de PALB2 dans les cellules induit une perte de la localisation de BRCA2, et donc de RAD51, aux cassures double-brin, d’où son nom : partenaire localisateur de BRCA2 (partner and localizer of BRCA2) [20]. PALB2 lie également la protéine BRCA1 via son domaine coiled-coil situé à son extrémité amino-terminal, et cette interaction est essentielle à sa propre localisation aux cassures double-brin [22]. Ce domaine permet également la dimérisation de PALB2 [23]. Un troisième domaine, nommé ChAM (chromatin association motif), est quant à lui important pour l’association de PALB2 à la chromatine [24]. La fonction initialement attribuée à PALB2 était d’ancrer BRCA2 aux structures nucléaires, mais sa fonction biochimique exacte, en revanche, restait à élucider.
Des études biochimiques, conduites indépendamment dans notre laboratoire et celui du Pr. P. Sung, ont révélé une interaction directe entre PALB2 et RAD51, ainsi qu’une activité de liaison de PALB2 à l’ADN. PALB2 possède deux domaines de liaison à l’ADN situés en amino-terminal et au centre de la protéine (Figure 2), qui lient préférentiellement les structures d’ADN en forme de D-loop [25, 26]. Ceci suggère que PALB2 pourrait directement influencer l’activité de RAD51 dans la recombinaison homologue. À l’aide d’essais enzymatiques, il a été récemment démontré que PALB2, ainsi que BRCA2, (1) stimulent la formation du filament de RAD51 sur un ADN simple-brin protégé par la protéine RPA, et (2) stimulent l’échange des brins catalysé par le filament nucléoprotéique RAD51 [9, 25–28]. Ces activités font de PALB2 et BRCA2 des médiateurs de la recombinaison homologue (Figure 3).
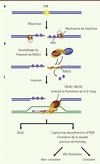 |
Figure 3. Fonction des médiateurs de la recombinaison homologue PALB2 et BRCA2. A. Résection de la cassure double-brin (CDB) par le complexe de résection pour former un ADN simple-brin protégé par RPA. B. Le complexe BRCA1-PALB2-BRCA2 recrute RAD51 à la cassure double-brin, puis PALB2 et BRCA2 stimulent la formation de son filament autour de l’ADN simple-brin. C. PALB2 et BRCA2 stimulent l’étape d’invasion impliquant la RAD51. dHJ : double jonction de Holliday. SDSA : synthesis-dependent strand annealing. |
Récemment, une autre étude conduite par notre laboratoire a montré que l’activité de ces deux médiateurs est contrôlée par la dimérisation de PALB2. Notre modèle propose qu’en absence de dommages à l’ADN, la protéine PALB2 complexée avec BRCA2 est sous une forme dimérique qui possède une activité basale (PALB2 off) (Figure 4). Lorsqu’il y a formation d’une cassure double-brin de l’ADN, PALB2 devient monomérique - une forme plus active (PALB2 on) - (Figure 4), et forme un complexe BRCA1-PALB2-BRCA2. BRCA1 est responsable de la localisation du complexe à la cassure double-brin qui permet à PALB2 et BRCA2 de stimuler la formation du filament de RAD51 sur l’ADN simple-brin fraîchement formé, puis l’étape d’invasion de la recombinaison homologue [23].
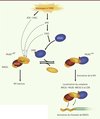 |
Figure 4. Modèle de la régulation de la dimérisation de PALB2 dans le contrôle de la réparation des cassures double-brin (CDB) de l’ADN. RH : recombinaison homologue. |
Conclusions et perspectives
Les progrès réalisés en génétique humaine ont permis d’identifier des gènes, tels que PALB2 et BRCA2, qui contribuent au développement du cancer ou de l’anémie de Fanconi. Il est maintenant établi que ces protéines jouent des rôles prépondérants dans la recombinaison homologue. Malgré ces grandes avancées, plusieurs interrogations subsistent. Quel est le rôle spécifique de ces protéines dans le développement de cancers du sein ou du pancréas, plus que d’autres tissus ? Quels sont les rôles des interactions entre le complexe PALB2-BRCA2 et ses partenaires ? Par exemple, PALB2 interagit avec les protéines MRG15, KEAP1 (Kelch-like ECH-associated protein-1) et RAD51AP1, mais la fonction exacte de ces interactions encore à déterminer.
Nous avons récemment proposé un nouveau mode de régulation pour PALB2 qui dépend de son auto-interaction. Nous savons que PALB2 est phosphorylée par la voie ATR (ATM and Cas3-related)/ATM (ataxia telangiectasia, mutated) en présence de cassures double-brin dans les cellules [29]. Cette phosphorylation pourrait être importante pour la régulation de la dimérisation de PALB2 (Figure 4), mais cela reste encore à démontrer. De même, le laboratoire du Dr S.N. Powell a proposé que la phosphorylation de la sérine 988 de BRCA1, par la kinase CHK2 en réponse à un dommage à l’ADN, active la formation du complexe BRCA1-PALB2-BRCA2 [7]. Par cette phosphorylation, BRCA1 pourrait donc contrôler l’équilibre entre les formes monomérique et dimérique de PALB2 dans la cellule (Figure 4) et ainsi activer ou non la recombinaison homologue. Par ailleurs, il a été montré que BRCA1 favorise cette recombinaison homologue en stimulant l’étape de résection, au détriment de la réparation par NHEJ [30]. La régulation de la dimérisation de PALB2 par BRCA1 serait un autre moyen pour la cellule de choisir la RH en phase S du cycle cellulaire au détriment du NHEJ pour la réparation des cassures double-brin. Il reste donc beaucoup à explorer, mais l’étude des médiateurs de la recombinaison homologue permettra de mieux comprendre les mécanismes de préservation de l’intégrité du génome et, ainsi, de prévenir les maladies humaines s’y rattachant.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Les auteurs remercient Joris Pauty, Anthony Couturier, Eloise Dray, Patrick Sung, Bing Xia, Isabelle Brodeur, et le laboratoire Masson pour les discussions sur le sujet. Ce projet est soutenu par les IRSC. R.B. est boursier du FQRNT et J.-Y. M. est chercheur senior du FRSQ.
Références
- Ciccia A, Elledge SJ. The DNA damage response: making it safe to play with knives. Mol Cell 2010 ; 40 : 179–204. [Google Scholar]
- Lord CJ, Ashworth A. The DNA damage response and cancer therapy. Nature 2012 ; 481 : 287–294. [CrossRef] [PubMed] [Google Scholar]
- Moynahan ME, Jasin M. Mitotic homologous recombination maintains genomic stability and suppresses tumorigenesis. Nat Rev Mol Cell Biol 2010 ; 11 : 196–207. [Google Scholar]
- San Filippo J, Sung P, Klein H. Mechanism of eukaryotic homologous recombination. Ann Rev Biochem 2008 ; 77 : 229–257. [CrossRef] [Google Scholar]
- Krejci L, Altmannova V, Spirek M, Zhao X. Homologous recombination and its regulation. Nucleic Acids Res 2012 ; 40 : 5795–5818. [CrossRef] [PubMed] [Google Scholar]
- West SC. Molecular views of recombination proteins and their control. Nat Rev Mol Cell Biol 2003 ; 4 : 435–445. [CrossRef] [PubMed] [Google Scholar]
- Roy R, Chun J, Powell SN. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nat Rev Cancer 2012 ; 12 : 68–78. [CrossRef] [Google Scholar]
- Yang H, Jeffrey PD, Miller J, et al. BRCA2 function in DNA binding and recombination from a BRCA2-DSS1-ssDNA structure. Science 2002 ; 297 : 1837–1848. [CrossRef] [PubMed] [Google Scholar]
- Liu J, Doty T, Gibson B, Heyer WD. Human BRCA2 protein promotes RAD51 filament formation on RPA-covered single-stranded DNA. Nat Struct Mol Biol 2010 ; 17 : 1260–1262. [CrossRef] [PubMed] [Google Scholar]
- Davies AA, Masson JY, McIlwraith MJ, et al. Role of BRCA2 in control of the RAD51 recombination and DNA repair protein. Mol Cell 2001 ; 7 : 273–282. [CrossRef] [PubMed] [Google Scholar]
- Carreira A, Kowalczykowski SC. Two classes of BRC repeats in BRCA2 promote RAD51 nucleoprotein filament function by distinct mechanisms. Proc Natl Acad Sci USA 2011 ; 108 : 10448–10453. [CrossRef] [Google Scholar]
- Chen CF, Chen PL, Zhong Q, et al. Expression of BRC repeats in breast cancer cells disrupts the BRCA2-Rad51 complex and leads to radiation hypersensitivity and loss of G(2)/M checkpoint control. J Biol Chem 1999 ; 274 : 32931–32935. [CrossRef] [PubMed] [Google Scholar]
- Galkin VE, Esashi F, Yu X, et al. BRCA2 BRC motifs bind RAD51-DNA filaments. Proc Natl Acad Sci USA 2005 ; 102 : 8537–8542. [CrossRef] [Google Scholar]
- Saeki H, Siaud N, Christ N, et al. Suppression of the DNA repair defects of BRCA2-deficient cells with heterologous protein fusions. Proc Natl Acad Sci USA 2006 ; 103 : 8768–8773. [CrossRef] [Google Scholar]
- Esashi F, Christ N, Gannon J, et al. CDK-dependent phosphorylation of BRCA2 as a regulatory mechanism for recombinational repair. Nature 2005 ; 434 : 598–604. [CrossRef] [PubMed] [Google Scholar]
- Esashi F, Galkin VE, Yu X, et al. Stabilization of RAD51 nucleoprotein filaments by the C-terminal region of BRCA2. Nat Struct Mol Biol 2007 ; 14 : 468–474. [CrossRef] [PubMed] [Google Scholar]
- Davies OR, Pellegrini L. Interaction with the BRCA2 C terminus protects RAD51-DNA filaments from disassembly by BRC repeats. Nat Struct Mol Biol 2007 ; 14 : 475–483. [PubMed] [Google Scholar]
- Schlacher K, Christ N, Siaud N, et al. Double-strand break repair-independent role for BRCA2 in blocking stalled replication fork degradation by MRE11. Cell 2011 ; 145 : 529–542. [CrossRef] [PubMed] [Google Scholar]
- Ayoub N, Rajendra E, Su X, et al. The carboxyl terminus of Brca2 links the disassembly of Rad51 complexes to mitotic entry. Curr Biol 2009 ; 19 : 1075–1085. [CrossRef] [PubMed] [Google Scholar]
- Xia B, Sheng Q, Nakanishi K, et al. Control of BRCA2 cellular and clinical functions by a nuclear partner, PALB2. Mol Cell 2006 ; 22 : 719–729. [CrossRef] [PubMed] [Google Scholar]
- Tischkowitz M, Xia B. PALB2/FANCN: recombining cancer and Fanconi anemia. Cancer Res 2010 ; 70 : 7353–7359. [CrossRef] [PubMed] [Google Scholar]
- Zhang F, Ma J, Wu J, et al. PALB2 links BRCA1 and BRCA2 in the DNA-damage response. Curr Biol 2009 ; 19 : 524–529. [CrossRef] [PubMed] [Google Scholar]
- Buisson R, Masson JY. PALB2 self-interaction controls homologous recombination. Nucleic Acids Res 2012 ; 40 : 10312–10323. [CrossRef] [PubMed] [Google Scholar]
- Bleuyard JY, Buisson R, Masson JY, Esashi F. ChAM, a novel motif that mediates PALB2 intrinsic chromatin binding and facilitates DNA repair. EMBO Rep 2012 ; 13 : 135–141. [CrossRef] [PubMed] [Google Scholar]
- Buisson R, Dion-Cote AM, Coulombe Y, et al. Cooperation of breast cancer proteins PALB2 and piccolo BRCA2 in stimulating homologous recombination. Nat Struct Mol Biol 2010 ; 17 : 1247–1254. [CrossRef] [PubMed] [Google Scholar]
- Dray E, Etchin J, Wiese C, et al. Enhancement of RAD51 recombinase activity by the tumor suppressor PALB2. Nat Struct Mol Biol 2010 ; 17 : 1255–1259. [CrossRef] [PubMed] [Google Scholar]
- Jensen RB, Carreira A, Kowalczykowski SC. Purified human BRCA2 stimulates RAD51-mediated recombination. Nature 2010 ; 467 : 678–683. [CrossRef] [PubMed] [Google Scholar]
- Thorslund T, McIlwraith MJ, Compton SA, et al. The breast cancer tumor suppressor BRCA2 promotes the specific targeting of RAD51 to single-stranded DNA. Nat Struct Mol Biol 2010 ; 17 : 1263–1265. [CrossRef] [PubMed] [Google Scholar]
- Matsuoka S, Ballif BA, Smogorzewska A, et al. ATM and ATR substrate analysis reveals extensive protein networks responsive to DNA damage. Science 2007 ; 316 : 1160–1166. [CrossRef] [PubMed] [Google Scholar]
- Bunting SF, Callen E, Wong N, et al. 53BP1 inhibits homologous recombination in Brca1-deficient cells by blocking resection of DNA breaks. Cell 2010 ; 141 : 243–254. [CrossRef] [PubMed] [Google Scholar]
- Miné-Hattab J, Rothstein R. Réparation de l’ADN : comment trouver le bon partenaire ? Med sci (Paris) 2012 ; 28 : 714–716. [Google Scholar]
Liste des figures
 |
Figure 1. Mécanismes de réparation des cassures double-brin de l’ADN. |
| Dans le texte | |
 |
Figure 2. Organisation des domaines fonctionnels des protéines BRCA1, PALB2 et BRCA2. Les protéines interagissant avec BRCA1, PALB2 ou BRCA2 sont indiquées en dessous des domaines correspondants. Les flèches indiquent les interactions entre BRCA1, PALB2 et BRCA2. Les sites de phosphorylation sont indiqués par les cercles roses. |
| Dans le texte | |
 |
Figure 3. Fonction des médiateurs de la recombinaison homologue PALB2 et BRCA2. A. Résection de la cassure double-brin (CDB) par le complexe de résection pour former un ADN simple-brin protégé par RPA. B. Le complexe BRCA1-PALB2-BRCA2 recrute RAD51 à la cassure double-brin, puis PALB2 et BRCA2 stimulent la formation de son filament autour de l’ADN simple-brin. C. PALB2 et BRCA2 stimulent l’étape d’invasion impliquant la RAD51. dHJ : double jonction de Holliday. SDSA : synthesis-dependent strand annealing. |
| Dans le texte | |
 |
Figure 4. Modèle de la régulation de la dimérisation de PALB2 dans le contrôle de la réparation des cassures double-brin (CDB) de l’ADN. RH : recombinaison homologue. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




