| Issue |
Med Sci (Paris)
Volume 29, Number 2, Février 2013
|
|
|---|---|---|
| Page(s) | 124 - 126 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2013292003 | |
| Published online | 28 February 2013 | |
Des polymorphismes silencieux plutôt bruyants
Noisy silent polymorphisms
1
Institut de recherche sur le cancer et le vieillissement de Nice (IRCAN), Inserm U1081, CNRS UMR 7284, Nice, France
2
Université de Nice Sophia-Antipolis, faculté de médecine, 28, avenue Valombrose, Nice, France
3
Centre hospitalier universitaire de Nice, 30, avenue de la voie romaine, Nice, France
*
sylviakorz@gmail.com
**
hofman@unice.fr
***
brest@unice.fr
Les polymorphismes nucléotidiques (single nucleotide polymorphisms, SNP) sont la forme la plus fréquente de variation génétique chez l’homme. Présents avec des fréquences variables dans la population générale, ils constituent une des sources majeures de variation interindividuelle génétique et phénotypique. Ils sont associés à la diversité entre des populations, aux différences de susceptibilité aux maladies et de sensibilité aux traitements. À ce jour, plus de 13 000 SNP ont été associés à des pathologies. Il s’agit de mutations germinales présentes aussi bien dans les régions codantes que dans les régions non codantes : séquences intergéniques, régions 3’ ou 5’ non traduites, régions introniques et autres régions non codantes comme les sites de fixation des facteurs de transcription.
Polymorphismes synonymes
Les SNP concernant les régions codantes se répartissent en trois catégories : les SNP synonymes, les SNP non synonymes et les SNP non-sens. Du fait de la redondance du code génétique, la substitution d’un codon par un autre codon peut engendrer le même acide aminé : c’est le cas pour les SNP synonymes. À l’inverse, les SNP non synonymes ou non-sens engendrent, respectivement, l’incorporation d’un acide aminé différent ou l’arrêt de la traduction ; ils modifient donc la structure primaire du polypeptide. Or, si l’on conçoit facilement que les SNP non synonymes et non-sens engendrent des perturbations moléculaires, cellulaires et physiologiques, les dernières données de la littérature ont, de façon surprenante, apporté la preuve qu’il en était de même pour les SNP synonymes (→) [11]. Il semblerait même que la probabilité d’association à des pathologies des SNP synonymes et non synonymes soit similaire et de même importance [1]. Ainsi, des études ont montré que les polymorphismes synonymes pouvaient modifier l’épissage de l’ARNm [2], sa stabilité [3] ou sa structure [4], mais aussi la traduction et la structure tertiaire des protéines (protein folding) [5]. Cependant, les mécanismes moléculaires précis expliquant la façon dont ces polymorphismes participent à l’émergence des processus pathologiques restent encore largement inconnus.
(→) Voir m/s 2012, vol. 28, n° 4, page 435
Polymorphismes synonymes en pathologie humaine
De nombreuses pathologies, cancers, maladies cardiovasculaires entre autres, sont multifactorielles et relèvent de facteurs génétiques pouvant être modulés par des facteurs environnementaux [6]. Le modèle couramment admis pour décrire le déterminisme génétique est un modèle polygénique, où l’apparition d’une pathologie est liée à la somme des effets d’un grand nombre de gènes. Pour conforter ce modèle et identifier les facteurs de risque génétiques influençant le développement et la progression de pathologies, des études d’association ont été mises en œuvre depuis 2006, comme l’International HapMap project ou les Genome-wide association studies (GWAS). Ces études permettent, en analysant le génome d’un grand nombre d’individus sains et de patients, de détecter l’effet, même modeste, des variants génétiques dans une pathologie donnée. De plus, ces études établissent des connexions entre les gènes et des voies cellulaires impliquées dans une pathologie. Ainsi, un nombre important de polymorphismes synonymes ont été associés à diverses pathologies [7].
Par exemple, quelques polymorphismes synonymes sont associés à un risque accru de développer un cancer. En effet, si le risque relatif d’apparition de cancer est généralement faible, la fréquence relativement importante de ces polymorphismes dans la population leur confère un poids considérable. La découverte du gène TERT (telomerase reverse transcriptase), qui joue un rôle majeur dans l’immortalité et/ou le vieillissement cellulaire, a valu à Elizabeth Blackburn, Jack Szostak et Carol Greider l’attribution du prix Nobel de médecine en 2009 [12] (→). Ce gène code pour la sous-unité catalytique de la télomérase et possède de très nombreuses variations génétiques. Par exemple, le polymorphisme synonyme rs2736098 est associé à une augmentation du risque de développer un cancer (risque relatif compris entre 1,13 et 1,35) [8]. Il est fréquent dans la population puisque trouvé chez 10 % de la population européenne à l’état homozygote, et 53 % à l’état hétérozygote.
(→) Voir m/s 2009, vol. 25, n° 11, page 973
Dernièrement, notre équipe a montré pour la première fois que la présence d’un polymorphisme synonyme pouvait altérer le site de fixation d’un microARN en phase codante et entraîner des perturbations biologiques [9]. Ainsi, dans la maladie de Crohn, une maladie inflammatoire chronique de l’intestin, le variant synonyme rs10065172, situé dans la région codante du gène IRGM (immune-related GTPase family M), a longtemps été considéré comme étant sans causalité avec la pathologie car il n’engendre ni altération dans la séquence de la protéine IRGM, ni modification des sites d’épissage. Il est pourtant fortement associé à la maladie de Crohn dans la population européenne. Nous avons montré qu’une famille de microARN, les miR-196, inhibe la traduction du variant protecteur d’IRGM (c.313C) (Figure 1A), mais pas celle du variant à risque (c.313T) (Figure 1B). De façon intéressante, nous avons aussi montré que l’expression de cette famille de microARN était restreinte aux cellules épithéliales intestinales, et détectée uniquement chez les patients en phase active (inflammatoire) de leur maladie. Nos résultats suggèrent que le variant rs10065172 altère le site de fixation du miR-196, ce qui engendre une perte de régulation de l’autophagie liée à la protéine IRGM [13] (→) et une persistance anormale des souches d’Escherichia coli adhérentes et invasives (AIEC) associées à la maladie de Crohn dans les cellules intestinales des individus porteurs de l’haplotype à risque. Par ailleurs, nous avons découvert que l’effet de ce polymorphisme (relativement fréquent dans la population) était complexe, car il n’induit une différence d’effet que lorsque le microARN est présent, c’est-à-dire en conditions inflammatoires et dans les entérocytes. En conclusion, nous avons montré que la perte de la fixation d’un microARN pouvait être l’un des mécanismes expliquant l’association d’un polymorphisme synonyme à une pathologie humaine.
(→) Voir m/s 2009, vol. 25, n° 4, page 349
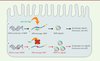 |
Figure 1. Régulation de la protéine IRGM par le mIR-196. Le miR-196 est surexprimé dans la cellule épithéliale intestinale. Il a une bonne affinité pour la forme sauvage de l’ARN messager d’IRGM (A) et régule ainsi l’autophagie, dont l’induction requiert la protéine IRGM. En revanche, il ne peut réguler l’ARN messager porteur du polymorphisme rs10065172 (B) du fait de sa moins bonne affinité pour cette forme. Il en résulte une perturbation de l’autophagie. AIEC : adherent-invasive-E. coli. |
Perspectives et conclusion
À l’heure où la médecine devient de plus en plus personnalisée et axée sur la prévention des risques, il semble essentiel de prendre en considération l’existence des mutations synonymes, et en particulier parce que les polymorphismes synonymes peuvent influencer le pronostic des cancers. Par exemple, dans les leucémies myéloïdes aiguës de l’adulte, le polymorphisme synonyme rs11554137 d’IDH1 (isocitrate déhydrogénase 1 [NADP+], soluble) est associé à un risque plus élevé de récidive [10]. D’autre part, l’efficacité et la tolérance de petites molécules thérapeutiques peuvent être affectées par des variations synonymes interindividuelles concernant des protéines impliquées dans leur métabolisme, comme les transporteurs et les enzymes. Par exemple, il a été montré que des mutations synonymes du transporteur ABCB1 (adénosine triphosphate-binding cassette, sous-famille B, membre 1) sont impliquées dans la résistance aux chimiothérapies [5].
En conclusion, bien que les mécanismes moléculaires associés aux polymorphismes synonymes soient encore incomplètement connus et l’importance globale à accorder à leur effet pathologique encore débattue, il est incontestable que les SNP synonymes jouent un rôle dans de nombreuses pathologies humaines.
Par ailleurs, ce type de variations génétiques introduit un nouveau concept de mutations conditionnelles : leurs effets, jusque-là silencieux, ne s’exprimeraient « bruyamment » qu’en présence d’autres facteurs et/ou conditions bien particulières, se situant ainsi au carrefour du déterminisme génétique et des facteurs environnementaux.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Les auteurs remercient la Fondation pour la recherche médicale (contrat n° DEA20110621547), l’Agence nationale de la recherche (contrat n° ANR-12-JSV1-0009-01) et la Fondation ARC (contrat n° 12ARC001MRAR) pour la recherche contre le cancer qui ont soutenu ce travail.
Références
- Chen R, Davydov EV, Sirota M, Butte AJ. Non-synonymous synonymous coding SNPs show similar likelihood, effect size of human disease association. PLoS One 2010 ; 5 : e13574. [CrossRef] [PubMed] [Google Scholar]
- Cartegni L, Chew SL, Krainer AR. Listening to silence and understanding nonsense: exonic mutations that affect splicing. Nat Rev Genet 2002 ; 3 : 285–298. [CrossRef] [PubMed] [Google Scholar]
- Nackley AG, Shabalina SA, Tchivileva IE, et al. Human catechol-O-methyltransferase haplotypes modulate protein expression by altering mRNA secondary structure. Science 2006 ; 314 : 1930–1933. [CrossRef] [PubMed] [Google Scholar]
- Shen LX, Basilion JP, Stanton VP. Single-nucleotide polymorphisms can cause different structural folds of mRNA. Proc Natl Acad Sci USA 1999 ; 96 : 7871–7876. [CrossRef] [Google Scholar]
- Kimchi-Sarfaty C, Oh JM, Kim IW, et al. A «Silent» polymorphism in the MDR1 gene changes substrate specificity. Science 2007 ; 315 : 525–528. [CrossRef] [PubMed] [Google Scholar]
- Hunt R, Sauna ZE, Ambudkar SV, et al. Silent (synonymous) SNP: should we care about them? Methods Mol Biol 2009 ; 578 : 23–39. [CrossRef] [PubMed] [Google Scholar]
- Sauna ZE, Kimchi-Sarfaty C. Understanding the contribution of synonymous mutations to human disease. Nat Rev Genet 2011 ; 12 : 683–691. [CrossRef] [PubMed] [Google Scholar]
- Mocellin S, Verdi D, Pooley KA, et al. Telomerase reverse transcriptase locus polymorphisms and cancer risk: a field synopsis and meta-analysis. J Natl Cancer Inst 2012 ; 104 : 840–854. [CrossRef] [PubMed] [Google Scholar]
- Brest P, Lapaquette P, Souidi M, et al. A synonymous variant in IRGM alters a binding site for miR-196 and causes deregulation of IRGM-dependent xenophagy in Crohn’s disease. Nat Genet 2011 ; 43 : 242–245. [CrossRef] [PubMed] [Google Scholar]
- Ho PA, Kopecky KJ, Alonzo TA, et al. Prognostic implications of the IDH1 synonymous SNP rs11554137 in pediatric and adult AML: a report from the Children’s Oncology Group and SWOG. Blood 2011 ; 118 : 4561–4566. [CrossRef] [PubMed] [Google Scholar]
- Jordan B. Chroniques génomiques. The sound of silence. Med Sci (Paris) 2012 ; 28 : 435–437. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Londoño-Vallejo A. Un Nobel centenaire célèbre télomères et télomérase. Med Sci (Paris) 2009 ; 25 : 973–976. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Glasser AL, Pierre Lapaquette P, Darfeuille-Michaud A. Altération de l’autophagie chez les patients atteints de maladie de Crohn : une porte ouverte pour des bactéries invasives ? Med Sci (Paris) 2009 ; 25 : 349–351. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
© 2013 médecine/sciences – Inserm / SRMS
Liste des figures
 |
Figure 1. Régulation de la protéine IRGM par le mIR-196. Le miR-196 est surexprimé dans la cellule épithéliale intestinale. Il a une bonne affinité pour la forme sauvage de l’ARN messager d’IRGM (A) et régule ainsi l’autophagie, dont l’induction requiert la protéine IRGM. En revanche, il ne peut réguler l’ARN messager porteur du polymorphisme rs10065172 (B) du fait de sa moins bonne affinité pour cette forme. Il en résulte une perturbation de l’autophagie. AIEC : adherent-invasive-E. coli. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




