| Issue |
Med Sci (Paris)
Volume 28, Number 11, Novembre 2012
|
|
|---|---|---|
| Page(s) | 911 - 914 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/20122811003 | |
| Published online | 12 November 2012 | |
Rôle de la duplication partielle du gène SRGAP2 dans l’évolution et le développement du cerveau humain
How human-specific SRGAP2 gene duplications control human brain development
Scripps Research Institute, Department of Cell Biology, Dorris Neuroscience Center, 10550 North Torrey Pines Road, Mail Drop DNC 202, La Jolla, CA 92037-1000, États-Unis
Un événement majeur de l’évolution du cerveau des mammifères est l’expansion du néocortex et l’accroissement de sa complexité. Ces différences représentent le substrat anatomique des différences cognitives et culturelles entre les humains et les primates non-humains. Cependant, notre compréhension des mécanismes génétiques, qui ont permis l’émergence de ces différences durant l’évolution, et leurs conséquences sur la composition neuronale, les réseaux neuronaux ou le fonctionnement synaptique reste fragmentaire.
Il a été montré récemment qu’un pic de duplications de segments de chromosomes s’est produit dans la lignée humaine lors de la séparation de cette dernière d’avec les primates non-humains (chimpanzés, bonobos), il y environ six millions d’années. Les duplications de séquences génomiques représentent un mécanisme d’innovation moléculaire majeur en permettant la création de nouveaux gènes au cours de l’évolution [1, 2]. Une trentaine de gènes ont été dupliqués uniquement chez l’homme [3, 4], dont SRGAP2 (Slit-Robo-GTPase activating protein 2). Ce gène est exprimé dans le cerveau au cours du développement et a été initialement identifié par notre laboratoire comme un régulateur de la migration et de la différenciation des neurones pyramidaux dans le cortex [5]. Les premières analyses suggéraient l’existence de deux copies supplémentaires de ce gène dans le génome humain [4]. Nous avons donc postulé que ces duplications pouvaient avoir contribué à certains aspects de la spéciation du cerveau humain.
SRGAP2 : un gène partiellement dupliqué dans le génome humain
Les séquences récemment dupliquées présentent un très fort taux d’identité (> 99,5 %) avec les séquences d’origine et sont donc en général absentes ou mal assemblées dans le génome de référence. Pour déterminer la localisation et la structure des duplications de SRGAP2, le laboratoire d’E. Eichler à l’université de Washington (Seattle, États-Unis) a construit et séquencé une banque de chromosomes artificiels bactériens issus d’ADN génomique d’une môle hydatiforme complète1 [6]. Cette dernière résulte de la fécondation d’un ovocyte humain énucléé par un spermatozoïde unique ; elle contient donc un équivalent haploïde du génome humain qui a l’avantage de ne pas présenter de variation allélique. Les auteurs ont ainsi mis en évidence trois copies supplémentaires de SRGAP2 dans le génome humain. La comparaison des séquences a permis de retracer l’évolution des duplications (Figure 1A). Le gène ancestral SRGAP2A a subi une première duplication partielle générant SRGAP2B, il y a environ 3,4 millions d’années. Cette duplication d’environ 260 000 paires de bases (260 kpb) comprend les neuf premiers exons du gène ancestral SRGAP2A (22 exons au total) ainsi que les séquences régulatrices. La duplication de SRGAP2B, il y a environ 2,4 millions d’années, a ensuite généré SRGAP2C. Cette seconde duplication (> 500 kpb) contient les séquences de la première duplication dans leur totalité ainsi que d’autres régions adjacentes. SRGAP2B a été dupliqué à nouveau il y a environ 1 million d’années. Cependant, le produit de cette dernière duplication (SRGAP2D) contient des délétions supplémentaires, et il est peu probable qu’il soit exprimé. Le séquençage complet du génome de plus de 600 individus sains (1 000 Genomes Project) a révélé que seuls SRGAP2A et SRGAP2C présentent un nombre fixe de copies dans le génome humain, alors que le nombre de copies de SRGAP2B varie [6]. Ceci suggère que SRGAP2B est en train de devenir un pseudogène alors que SRGAP2C est potentiellement un nouveau gène spécifiquement humain.
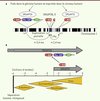 |
Figure 1. Évolution du gène SRGAP2 dans la lignée humaine. A. Représentation de la localisation et de l’évolution des paralogues du gène SRGAP2 (triangles bleus) sur le chromosome 1 humain. Le décours temporel des duplications est indiqué sous le chromosome. Les protéines prédites à partir des séquences dupliquées sont indiquées au-dessus du chromosome. Tous les paralogues spécifiquement humains codent potentiellement pour des formes tronquées de SRGAP2. Seuls SRGAP2A et SRGAP2C (entourés) sont fixés dans le génome humain et exprimés dans le cerveau humain (ma : millions d’années) (modifié d’après [6]). B. Schéma représentant l’expression de SRGAP2A et SRGAP2C (gris) au cours de l’évolution de la lignée humaine depuis la séparation d’avec les primates non-humains. Différents genres d’hominidés sont représentés en jaune le long de la ligne du temps. SRGAP2A est présent chez tous les mammifères, alors que l’apparition de SRGAP2C coïncide avec la séparation entre le genre Homo et les australopithèques [6]. SRGAP2C inhibe la fonction de SRGAP2A en inter-agissant avec son domaine F-BAR [7]. |
Inhibition de SRGAP2 ancestral par ses copies humaines : conséquences sur le développement du cortex
Protéines codées par les copies du gène SRGAP2 ancestral
SRGAP2A code pour une protéine fortement conservée chez les mammifères (SRGAP2). SRGAP2 comprend trois domaines fonctionnels : un domaine F-BAR (Bin, amphiphysin, Rvs) en amino-terminal, capable d’induire des filopodes lorsqu’il est exprimé dans des lignées cellulaires [5] ; suit un domaine central de type Rho-GAP (GTPase-activating protein) qui catalyse l’activité GTPase de Rac1 ; et enfin une queue carboxy-terminale contenant un domaine SH3 (src homology 3 domain). SRGAP2B et SRGAP2C, qui ont une séquence très similaire, codent pour une protéine correspondant au domaine F-BAR de SRGAP2, tronqué de ses 49 derniers acides aminés (Figure 1A). SRGAP2A et SRGAP2C sont transcrits dans le cerveau humain embryonnaire et adulte, ainsi que dans des neurones de type « télencéphaliques » dérivés in vitro de cellules souches embryonnaires humaines [7]. Dans le cerveau humain, leur patron d’expression est similaire dans les progéniteurs neuronaux et les neurones postmitotiques. Une protéine correspondant à SRGAP2B/C est également détectée dans des lignées issues de neuroblastomes humains.
Les protéines SRGAP2B et SRGAP2C ont perdu la capacité de déformation de membrane qu’exprime SRGAP2A, mais conservent la capacité de dimérisation conférée par leur domaine F-BAR [7]. L’interaction entre SRGAP2A et la forme humaine tronquée SRGAP2C inhibe la fonction de SRGAP2A (Figure 1B). Ainsi, in vivo, au cours du développement embryonnaire, SRGAP2A promeut le branchement des neurones pyramidaux, ce qui ralentit leur migration dans le cortex. En revanche, l’introduction de SRGAP2C dans des neurones de souris par électroporation in utero phénocopie la perte de fonction de SRGAP2 et accélère la migration des neurones dans le cortex [7]. Cet effet ne perturbe pas le placement final des neurones dans le cortex, mais pourrait représenter un avantage dans le cerveau humain où la distance à parcourir est beaucoup plus longue.
Antagonisme entre SRGAP2A et SRGAP2C dans le contrôle de la formation des épines dendritiques
Puisque la perte de fonction de SRGAP2 ne modifie pas l’organisation laminaire du cortex, nous nous sommes intéressés au rôle de la protéine à des stades plus tardifs du développement. Chez la souris, SRGAP2 est abondamment exprimée dans le cortex pendant les premières semaines après la naissance, une période pendant laquelle se forment la majorité des connexions synaptiques. Nous avons montré que SRGAP2 s’accumule dans les épines dendritiques, des protrusions localisées le long des dendrites qui reçoivent la majorité des afférences excitatrices dans le cortex où la protéine est associée à la densité postsynaptique (Figure 2). Nous avons testé la fonction de SRGAP2 dans les épines en utilisant différentes approches de perte et de gain de fonctions. Nos résultats montrent que SRGAP2 régule trois caractéristiques des épines dendritiques : (1) la taille de la tête des épines ; celle-ci est corrélée à la taille de la densité postsynaptique, au nombre de récepteurs du glutamate de type AMPA (α-amino-3-hydroxy-5-methyl-4-isoxazolepropionic acid) et elle peut être utilisée comme un indicateur de la maturation synaptique. (2) La longueur du cou des épines, qui influence l’isolation biochimique et électrique entre le contact synaptique localisé dans la tête et le tronc dendritique. (3) La densité des épines dendritiques. Ainsi, in vivo, la forme ancestrale de SRGAP2 promeut la maturation des épines et limite leur densité, alors que l’expression de SRGAP2C dans des neurones corticaux de souris retarde leur maturation (néoténie) et conduit à une forte augmentation de la densité des épines. Celles-ci maintiennent, au stade adulte, des cous allongés par rapport aux épines contrôles (Figure 2) [7].
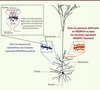 |
Figure 2. SRGAP2A et son paralogue humain SRGAP2C contrôlent la morphologie et la densité des épines dendritiques. Les épines dendritiques (encart noir) couvrent les dendrites des neurones pyramidaux du néocortex. Elles représentent le compartiment postsynaptique des synapses excitatrices et sont généralement apposées à un bouton présynaptique qui libère des vésicules contenant du glutamate, le neurotransmetteur excitateur principal dans le système nerveux central. Les récepteurs ionotropiques du glutamate sont accumulés dans les épines en face des terminaisons axonales où ils sont stabilisés de manière dynamique par les protéines de la densité postsynaptique, ce qui assure l’efficacité de la neurotransmission. Le cytosquelette d’actine est un déterminant majeur de la plasticité morphologique des épines. Chez tous les mammifères non-humains (encart bleu), SRGAP2A limite la densité d’épines dendritiques et promeut leur maturation au cours du développement (non montré). SRGAP2C, qui est exprimée uniquement dans les neurones humains, inhibe la fonction de SRGAP2A. L’expression de SRGAP2C dans les neurones de souris retarde la maturation des épines et conduit à l’augmentation de leur densité ainsi qu’à leur allongement (encart rouge). Ces effets miment la déficience génétique en SRGAP2A dans les neurones de souris et sont caractéristiques du développement des neurones corticaux pyramidaux humains (voir [7]). |
Il est remarquable que l’expression de SRGAP2C mime, au stade embryonnaire comme aux stades juvénile et adulte, les effets consécutifs à la réduction de l’expression de la protéine SRGAP2 endogène, de même que ceux qui sont observés dans une souris mutante hypomorphe. Ceci indique que SRGAP2C est capable d’inhiber la fonction de la forme ancestrale SRGAP2A dans les neurones corticaux tout au long du développement, avec pour conséquences des épines plus grandes et présentes à une plus forte densité. Plusieurs groupes ont montré que les neurones corticaux pyramidaux humains se caractérisent par une prolongation de la période de développement des épines (néoténie) [8, 9] et une plus grande complexité morphologique (incluant une densité d’épines plus élevée et des épines aux cous plus longs) par rapport aux neurones de singes ou de souris [10, 11]. Nos résultats suggèrent que les duplications spécifiquement humaines de SRGAP2, survenues au moment de la séparation entre le genre Homo et les australopithèques, ont contribué à l’émergence de ces caractéristiques.
Conclusion
Chez l’homme, l’augmentation de la densité d’épines indique que les neurones sont engagés dans un nombre plus important de contacts synaptiques, alors que l’allongement du cou des épines suggère des différences dans les propriétés d’intégration synaptique. La complexification des neurones corticaux pyramidaux, associée au prolongement de la période particulièrement plastique de développement synaptique, pourrait augmenter les possibilités de stockage et la flexibilité du traitement de l’information dans les réseaux corticaux. Elle pourrait ainsi contribuer à la richesse des interactions entre l’homme et son environnement, ainsi qu’à ses capacités cognitives, d’apprentissage et de mémorisation. Dans ce contexte, l’identification des mécanismes moléculaires par lesquels SRGAP2 régule le développement des épines et qui sont modifiés spécifiquement chez l’homme du fait de l’expression de SRGAP2C, pourrait ouvrir de nouvelles voies pour la compréhension des troubles neurodéveloppementaux ou psychiatriques, comme l’autisme ou la schizophrénie.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Une môle hydatiforme se développe lors d’une grossesse résultant d’une anomalie de la fécondation et caractérisée par une prolifération trophoblastique sans développement embryonnaire normal possible (0,1 à 0,5 % des grossesses). La môle complète dérive de la fécondation d’un ovule anucléé par un ou deux spermatozoïdes haploïdes. C’est une grossesse sans embryon où seules les membranes placentaires se développent. Les môles hydatiformes partielles sont triploïdes, issues de la fécondation d’un ovocyte contenant un pronucléus femelle par deux spermatozoïdes, ou par un spermatozoïde anormal.
Références
- Lynch M, Katju V. The altered evolutionary trajectories of gene duplicates. Trends Genet 2004 ; 20 : 544–549. [CrossRef] [PubMed] [Google Scholar]
- Ohno S. Evolution of gene duplication. Berlin: Springer-Verlag, 1970. [Google Scholar]
- Marques-Bonet T, Kidd JM, Ventura M, et al. A burst of segmental duplications in the genome of the African great ape ancestor. Nature 2009 ; 457 : 877–881. [CrossRef] [PubMed] [Google Scholar]
- Sudmant PH, Kitzman JO, Antonacci F, et al. Diversity of human copy number variation and multicopy genes. Science 2010 ; 330 : 641–646. [CrossRef] [PubMed] [Google Scholar]
- Guerrier S, Coutinho-Budd J, Sassa T, et al. The F-BAR domain of srGAP2 induces membrane protrusions required for neuronal migration and morphogenesis. Cell 2009 ; 138 : 990–1004. [CrossRef] [PubMed] [Google Scholar]
- Dennis MY, Nuttle X, Sudmant PH, et al. Evolution of human-specific neural SRGAP2 genes by incomplete segmental duplication. Cell 2012 ; 149 : 912–922. [CrossRef] [PubMed] [Google Scholar]
- Charrier C, Joshi K, Coutinho-Budd J, et al. Inhibition of SRGAP2 function by its human-specific paralogs induces neoteny during spine maturation. Cell 2012 ; 149 : 923–935. [CrossRef] [PubMed] [Google Scholar]
- Huttenlocher PR, Dabholkar AS. Regional differences in synaptogenesis in human cerebral cortex. J Comp Neurol 1997 ; 387 : 167–178. [CrossRef] [PubMed] [Google Scholar]
- Petanjek Z, Judas M, Simic G, et al. Extraordinary neoteny of synaptic spines in the human prefrontal cortex. Proc Natl Acad Sci USA 2011 ; 108 : 13281–13286. [CrossRef] [Google Scholar]
- Benavides-Piccione R, Ballesteros-Yanez I, DeFelipe J, Yuste R. Cortical area and species differences in dendritic spine morphology. J Neurocytol 2002 ; 31 : 337–346. [CrossRef] [PubMed] [Google Scholar]
- Elston GN, Benavides-Piccione R, DeFelipe J. The pyramidal cell in cognition: a comparative study in human and monkey. J Neurosci 2001 ; 21 : RC163. [PubMed] [Google Scholar]
© 2012 médecine/sciences – Inserm / SRMS
Liste des figures
 |
Figure 1. Évolution du gène SRGAP2 dans la lignée humaine. A. Représentation de la localisation et de l’évolution des paralogues du gène SRGAP2 (triangles bleus) sur le chromosome 1 humain. Le décours temporel des duplications est indiqué sous le chromosome. Les protéines prédites à partir des séquences dupliquées sont indiquées au-dessus du chromosome. Tous les paralogues spécifiquement humains codent potentiellement pour des formes tronquées de SRGAP2. Seuls SRGAP2A et SRGAP2C (entourés) sont fixés dans le génome humain et exprimés dans le cerveau humain (ma : millions d’années) (modifié d’après [6]). B. Schéma représentant l’expression de SRGAP2A et SRGAP2C (gris) au cours de l’évolution de la lignée humaine depuis la séparation d’avec les primates non-humains. Différents genres d’hominidés sont représentés en jaune le long de la ligne du temps. SRGAP2A est présent chez tous les mammifères, alors que l’apparition de SRGAP2C coïncide avec la séparation entre le genre Homo et les australopithèques [6]. SRGAP2C inhibe la fonction de SRGAP2A en inter-agissant avec son domaine F-BAR [7]. |
| Dans le texte | |
 |
Figure 2. SRGAP2A et son paralogue humain SRGAP2C contrôlent la morphologie et la densité des épines dendritiques. Les épines dendritiques (encart noir) couvrent les dendrites des neurones pyramidaux du néocortex. Elles représentent le compartiment postsynaptique des synapses excitatrices et sont généralement apposées à un bouton présynaptique qui libère des vésicules contenant du glutamate, le neurotransmetteur excitateur principal dans le système nerveux central. Les récepteurs ionotropiques du glutamate sont accumulés dans les épines en face des terminaisons axonales où ils sont stabilisés de manière dynamique par les protéines de la densité postsynaptique, ce qui assure l’efficacité de la neurotransmission. Le cytosquelette d’actine est un déterminant majeur de la plasticité morphologique des épines. Chez tous les mammifères non-humains (encart bleu), SRGAP2A limite la densité d’épines dendritiques et promeut leur maturation au cours du développement (non montré). SRGAP2C, qui est exprimée uniquement dans les neurones humains, inhibe la fonction de SRGAP2A. L’expression de SRGAP2C dans les neurones de souris retarde la maturation des épines et conduit à l’augmentation de leur densité ainsi qu’à leur allongement (encart rouge). Ces effets miment la déficience génétique en SRGAP2A dans les neurones de souris et sont caractéristiques du développement des neurones corticaux pyramidaux humains (voir [7]). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




