| Issue |
Med Sci (Paris)
Volume 27, Number 4, Avril 2011
|
|
|---|---|---|
| Page(s) | 354 - 356 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2011274007 | |
| Published online | 28 April 2011 | |
Attaque bactérienne du système ubiquitine-protéasome
A bacterial attack on the ubiquitin-proteolytic system
Institut Jacques Monod, CNRS, Université Paris Diderot, Bâtiment Buffon, 15, rue Hélène Brion, 75205 Paris Cedex 13, France
*
pintard.lionel@ijm.univ-paris-diderot.fr
Escherichia coli est un hôte naturel de la flore intestinale des mammifères, très commun chez l’homme. Cependant, certaines souches sont pathogènes, comme les EPEC (E. coli entéropathogènes) qui provoquent des infections intestinales à l’origine de fortes diarrhées induisant une importante mortalité chez les nouveau-nés et les jeunes enfants dans les pays en développement. Des travaux récents, tout à fait fascinants, indiquent que ces bactéries pathogènes détournent la cellule hôte à leur profit en piratant un acteur majeur du fonctionnement cellulaire, le système ubiquitine-protéasome (UbPr).
© 2011 médecine/sciences – Inserm / SRMS
Les cullines : protéines plates-formes du système ubiquitine-protéasome
Le système UbPr joue un rôle central dans le contrôle de l’homéostasie cellulaire en permettant la dégradation des protéines. Outre son rôle majeur dans l’élimination de protéines incorrectement repliées et/ou agrégées, le système UbPr permet de changer, rapidement et de manière irréversible, le contenu cellulaire en protéines régulatrices. Ainsi, ce système contrôle de nombreux processus biologiques et cellulaires comme, par exemple, les transitions entre les différentes phases du cycle cellulaire. Le système UbPr met en jeu trois enzymes, E1, E2 et E3, dont l’action coordonnée conduit au transfert covalent de l’ubiquitine, un petit polypeptide de 76 acides aminés, sur un substrat protéique. La réitération de ce processus conduit à l’assemblage d’une chaîne d’ubiquitines sur le substrat (polyubiquitinylation), ciblant ainsi sa dégradation par le protéasome 26S, un complexe macromoléculaire doté d’activités protéasiques [1, 2]. L’étape centrale du système UbPr est la reconnaissance spécifique du substrat à ubiquitinyler. Celle-ci est assurée par l’ubiquitine ligase (E3) dont la plus importante famille est constituée des complexes recrutés par des cullines ou CRL (cullin RING ligases) [3] (Figure 1). Ces complexes sont les promoteurs du système ubiquitine-protéasome puisqu’ils contrôlent la dégradation d’une myriade de protéines régulatrices et notamment de nombreux régulateurs du cycle cellulaire comme p27kip1, un inhibiteur de Cdk (cyclin dependent kinase), la protéine Cdt1 (chromatin licensing and DNA replication factor 1) qui est dégradée en phase S ce qui assure une seule réplication de l’ADN par cycle cellulaire, ou encore la GTPase RhoA qui régule le cytosquelette d’actine (Figure 1). Bien que la composition des complexes soit variable, ils sont régulés par des mécanismes identiques qui impliquent la conjugaison de la protéine Nedd8 (neural precursor cell expressed, developmentally down-regulated 8), une protéine similaire à l’ubiquitine, sur une lysine carboxy-terminale de la sous-unité culline (Figure 2). Cette modification, également appelée neddylation, induit un changement de conformation du site catalytique per- mettant de situer l’enzyme E2 activée à proximité du substrat et de favoriser ainsi sa polyubiquitinylation [4]. Cette modification est réversible : la liaison culline-Nedd8 est hydrolysée par un complexe multiprotéique appelé le COP9 signalosome.
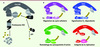 |
Figure 1 Composition, fonction et régulation des complexes CRL. A. Voie de dégradation des protéines par les complexes CRL. Ces complexes sont échafaudés par une protéine plate-forme, la culline qui recrute, ia son extrémité carboxy-terminale, la protéine à domaine RING-finger Rbx1 sur laquelle vient se fixer la protéine E2 activée, et via son extrémité amino-terminale, le module de reconnaissance du substrat. B. Les génomes eucaryotes codent plusieurs cullines qui assemblent divers complexes CRL utilisant le même site catalytique mais des modules de reconnaissance de substrats distincts. Ces modules sont composés généralement d’une protéine adaptatrice (ex. SKP1) qui lie la culline et d’une protéine responsable de la reconnaissance spécifique du substrat à dégrader (ex. F-box). Le complexe CRL1SKP2 (violet) est impliqué dans la dégradation de p27Kip1 et la progression du cycle cellulaire. Les complexes CRL2VHL, CRL3BACURD et CRL4 Cdt2 ciblent respectivement la dégradation d’HIF-1 (hypoxia inducing factor), RhoA et Cdt1 contrôlant ainsi divers processus. |
 |
Figure 2 Cif modifie Nedd8 et neutralise l’activation des complexes CRL. A. La conjugaison de Nedd8 (vert) sur une lysine carboxy-terminale de la culline induit un changement de conformation du domaine RING qui permet de situer l’enzyme E2 activé à proximité du substrat favorisant ainsi sa polyubiquitinylation et dégradation par le protéasome. Le COP9 signalosome (bleu) hydrolyse la liaison culline- Nedd8. B. Cif modifie Nedd8 en déamidant une glutamine en position 40. Cette version mutée de Nedd8 (rouge) est correctement conjuguée aux cullines mais bloque vraisemblablement le changement de conformation du domaine RING qui est nécessaire à l’activation des CRL et à leur déneddylation par le COP9 signalosome. |
Facteur bactérien Cif et neutralisation des complexes recrutés par les cullines
Des travaux récents indiquent que les bactéries entéropathogènes piratent ce cycle de neddylation et déneddylation. Les EPEC agissent en injectant dans la cellule hôte un cocktail d’effecteurs via une seringue moléculaire. Cif (cycle inhibiting factor), qui est l’un de ces effecteurs, bloque les cellules en phases G1 et G2 du cycle cellulaire et remodèle le cytosquelette d’actine ce qui facilite l’adhésion des bactéries aux cellules du tissu infecté. Bien que Cif présente la signature d’une enzyme appartenant à la superfamille des cystéines protéases [5], sa cible et son mécanisme d’action restaient à découvrir. Afin de rechercher les cibles cellulaires de Cif, Jubelin et al. ont réalisé un crible double hybride et identifié Nedd8 comme l’unique protéine qui interagit avec Cif [6]. Les auteurs ont identifié une hélice α de Cif spécifiquement requise pour l’interaction avec Nedd8. L’interaction entre Cif et Nedd8 est significative in vivo puisque l’expression de Cif dans les cellules s’accompagne d’une accumulation massive de la forme neddylée des cullines 1, 2, 3, 4A et 4B et d’une stabilisation de leurs substrats respectifs dont p27, RhoA, Cdt1. Ceci entraîne un arrêt des cellules en phases G1 et G2 du cycle cellulaire et le remodelage du cytosquelette d’actine. L’expression d’une version inactive, mutée sur la cystéine catalytique, n’a pas d’effet cytostatique ce qui démontre que l’activité enzymatique de Cif est requise pour sa fonction. De plus Cif neutralise l’activité E3-ligase d’un complexe CRL in vitro alors que la version mutée de Cif n’a aucun effet, ce qui indique que l’effet de Cif sur les complexes CRL est direct.
Comment Cif neutralise-t-il les complexes CRL et quel est son mécanisme d’action ? Dans une autre étude, Cui et al. ont noté que Cif induit un changement de la mobilité électrophorétique de Nedd8, mais également de l’ubiquitine, suggérant que Cif modifie directement ces protéines [7]. L’analyse par spectrométrie de masse en tandem a confirmé cette hypothèse et montré que Cif catalyse la déamidation d’une glutamine (Q) en position 40 de Nedd8 en induisant la formation d’un acide glutamique (E) sur cette position. De façon remarquable, l’expression d’une version mutée de Nedd8, contenant un acide glutamique en position 40, récapitule entièrement les phénotypes résultant de l’expression de Cif dans les cellules. De plus, des mutations ponctuelles affectant l’interaction entre Cif et Nedd8 suppriment l’effet cytostatique de Cif, ce qui démontre que Nedd8 est la cible principale de Cif.
Comment la déamidation de la glutamine 40 en glutamate affecte-t-elle la fonction de Nedd8 et des complexes CRL ? La conjugaison de cette version mutée de Nedd8 sur les cullines n’est clairement pas affectée. En revanche, la déneddylation par le COP9 signalosome est sévèrement compromise. Ces données suggèrent que Nedd8 Q40E bloque le changement de conformation du site catalytique des complexes CRL qui est nécessaire à leur activation, et que ce changement de conformation est requis pour la dénéddylation par le COP9 signalosome. En accord avec cette hypothèse, l’analyse de la structure tridimensionnelle du complexe CRL5 neddylé [4] révèle que la glutamine 40 se trouve à proximité de deux résidus de Cul5 chargés positivement et négativement. Il est donc raisonnable de penser que l’introduction d’un résidu chargé négativement (E) sur cette position altère les interactions électrostatiques entre la culline et Nedd8 et ainsi le changement de conformation du site catalytique de l’enzyme.
Comme les virus, des pirates bien connus du système ubiquitine-protéasome et des complexes CRL en particulier [8], les bactéries entéropathogènes détournent à leur profit les promoteurs du système ubiquitine-protéasome, en ciblant leur cycle d’activation par neddylation et déneddylation. Ainsi les bactéries et les virus révèlent un « tendon d’Achille » du cycle cellulaire. Il est d’ailleurs intéressant de noter qu’un inhibiteur spécifique de la voie de neddylation émerge comme un composé très prometteur en cancérologie [9].
Conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
Références
- HershkoA, HellerH, EliasS, Ciechanover A Components of ubiquitin-protein ligase system. Resolution, affinity purification, and role in protein breakdown. J Biol Chem 1983 ; 258 : 8206-8214. [PubMed] [Google Scholar]
- StrochlicL. Nedd4/PTEN : un couple très branché !Med Sci (Paris) 2010 ; 26 : 703-705. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- PetroskiMD, DeshaiesRJ. Function and regulation of cullin-RING ubiquitin ligases. Nat Rev Mol Cell Biol 2005 ; 6 : 9-20. [CrossRef] [PubMed] [Google Scholar]
- DudaDM, BorgLA, ScottDC, et al. Structural insights into NEDD8 activation of cullin-RING ligases : conformational control of conjugation. Cell 2008 ; 134 : 995-1006. [CrossRef] [PubMed] [Google Scholar]
- HsuY, JubelinG, TaiebF,et al. Structure of the cyclomodulin Cif from pathogenic Escherichia coli. J Mol Biol 2008 ; 384 : 465-477. [CrossRef] [PubMed] [Google Scholar]
- JubelinG, TaiebF, DudaDM, et al. Pathogenic bacteria target NEDD8-conjugated cullins to hijack host-cell signaling pathways. PLoS Pathog 2010 ; 6. [Google Scholar]
- CuiJ, YaoQ, LiS, et al. Glutamine deamidation and dysfunction of ubiquitin/NEDD8 induced by a bacterial effector family. Science 2010 ; 329 : 1215-1218. [CrossRef] [PubMed] [Google Scholar]
- GastaldelloS, HildebrandS, FaridaniO, et al. A deneddylase encoded by Epstein-Barr virus promotes viral DNA replication by regulating the activity of cullin-RING ligases. Nat Cell Biol 2010 ; 12 : 351-361. [CrossRef] [PubMed] [Google Scholar]
- SoucyTA, SmithPG, RolfeM. Targeting NEDD8-activated cullin-RING ligases for the treatment of cancer. Clin Cancer Res 2009 ; 15 : 3912-3916. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1 Composition, fonction et régulation des complexes CRL. A. Voie de dégradation des protéines par les complexes CRL. Ces complexes sont échafaudés par une protéine plate-forme, la culline qui recrute, ia son extrémité carboxy-terminale, la protéine à domaine RING-finger Rbx1 sur laquelle vient se fixer la protéine E2 activée, et via son extrémité amino-terminale, le module de reconnaissance du substrat. B. Les génomes eucaryotes codent plusieurs cullines qui assemblent divers complexes CRL utilisant le même site catalytique mais des modules de reconnaissance de substrats distincts. Ces modules sont composés généralement d’une protéine adaptatrice (ex. SKP1) qui lie la culline et d’une protéine responsable de la reconnaissance spécifique du substrat à dégrader (ex. F-box). Le complexe CRL1SKP2 (violet) est impliqué dans la dégradation de p27Kip1 et la progression du cycle cellulaire. Les complexes CRL2VHL, CRL3BACURD et CRL4 Cdt2 ciblent respectivement la dégradation d’HIF-1 (hypoxia inducing factor), RhoA et Cdt1 contrôlant ainsi divers processus. |
| Dans le texte | |
 |
Figure 2 Cif modifie Nedd8 et neutralise l’activation des complexes CRL. A. La conjugaison de Nedd8 (vert) sur une lysine carboxy-terminale de la culline induit un changement de conformation du domaine RING qui permet de situer l’enzyme E2 activé à proximité du substrat favorisant ainsi sa polyubiquitinylation et dégradation par le protéasome. Le COP9 signalosome (bleu) hydrolyse la liaison culline- Nedd8. B. Cif modifie Nedd8 en déamidant une glutamine en position 40. Cette version mutée de Nedd8 (rouge) est correctement conjuguée aux cullines mais bloque vraisemblablement le changement de conformation du domaine RING qui est nécessaire à l’activation des CRL et à leur déneddylation par le COP9 signalosome. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




