| Issue |
Med Sci (Paris)
Volume 24, Number 8-9, Août-Septembre 2008
|
|
|---|---|---|
| Page(s) | 747 - 752 | |
| Section | M/S revues | |
| DOI | https://doi.org/10.1051/medsci/20082489747 | |
| Published online | 15 August 2008 | |
Asymétrie des génomes parentaux
Implications en pathologie
Differential epigenetic marking on imprinted genes and consequences in human diseases
Institut de Génétique Moléculaire de Montpellier, CNRS et Université de Montpellier, 34293 Montpellier Cedex 5, France
Les modifications épigénétiques touchent l’organisation de la chromatine et l’expression des gènes, sans affecter la séquence de l’ADN, et de manière transmissible. L’un des exemples majeurs de cette régulation épigénétique est l’asymétrie des génomes parentaux chez les mammifères. En effet, au moment de la fécondation, les génomes maternel et paternel sont globalement configurés d’une manière très différente, le génome de l’ovocyte est empaqueté avec des histones, celui du spermatozoïde est empaqueté avec des protamines. Les deux génomes sont également très différents sur le plan fonctionnel : dans les cellules germinales mâles et femelles, des marques épigénétiques distinctes sont acquises au niveau des gènes soumis à l’empreinte. Une mise en place ou un maintien inadéquat de cette asymétrie épigénétique entre les deux génomes parentaux se traduit par différentes pathologies dont la tumorigenèse.
Abstract
At the time of fertilisation, the parental genomes have a strikingly different organisation. Sperm DNA is packaged globally with protamines, whereas the oocyte’s genome is wrapped around nucleosomes. The maternal and paternal genomes are functionally different as well, and are both required for normal uterine and postnatal development. The functional requirement of both parental genomes is a consequence of differential epigenetic marking by DNA methylation during oogenesis and spermatogenesis, on a group of genes called imprinted genes. After fertilisation, these parental marks persist throughout development and convey the allelic expression of imprinted genes. Pathological perturbation of methylation imprints, before fertilisation in the germ cells, or during development, gives rise to growth-related syndromes, and is frequently observed in cancer as well. Alteration of imprints is thought to occur early in carcinogenesis and shows similarities with the acquisition of aberrant DNA methylation at tumour suppressor genes. This suggests that similar underlying mechanisms could be involved.
© 2008 médecine/sciences - Inserm / SRMS
Asymétrie épigénétique des génomes parentaux
La parthénogenèse, c’est-à-dire le développement de l’œuf sans fécondation, s’effectue chez de nombreuses espèces mais elle est impossible chez les mammifères : cela est dû à une différence entre les génomes parentaux qui sont tous les deux nécessaires à un développement normal.
L’existence de cette asymétrie entre les génomes parentaux a été mise en évidence dans les années 1980 par des expériences de transfert de noyaux après la fécondation de l’œuf. Ces expériences chez la souris [1, 2] ont permis de créer des gynogénotes (issus de deux pronucléus femelles) et des androgénotes (issus de deux pronucléus mâles). Malgré la présence de deux copies du génome, les conceptus issus de ces manipulations présentent un phénotype délétère : les cellules androgénétiques participent fortement à la formation du trophectoderme mais peu à celle des tissus embryonnaires tandis que les cellules gynogénétiques contribuent à former les tissus embryonnaires mais pas ceux du trophectoderme [1, 2]. Dans les deux cas, ces défauts de morphogenèse ne permettent pas aux embryons de se développer au-delà de 10 jours de gestation. Le phénotype de ces androgénotes murins n’est pas sans rappeler un phénotype similaire chez l’homme dans le cas des môles hydatidiformes (voir plus loin).
Domaines soumis à empreinte et méthylation de l’ADN
La poursuite de ces recherches a montré que ces phénotypes étaient principalement dus à l’expression anormale de gènes spécifiques [2, 3]. Pour conduire à un être viable, ces gènes doivent n’être exprimés qu’à partir d’un seul des deux génomes parentaux. Ils sont appelés « gènes soumis à l’empreinte génomique » [4]. Aujourd’hui, environ 90 gènes soumis à l’empreinte ont été identifiés chez l’homme et chez la souris. La plupart d’entre eux sont organisés en domaines chromosomiques [5]. Ces domaines soumis à l’empreinte sont régulés par des régions de contrôle de l’empreinte (ICR, pour imprinting control region) qui portent des modifications épigénétiques spécifiques. En effet, ces régions, qui sont pour la plupart riches en dinucléotides CpG [6, 7], ont la particularité d’être méthylées de manière allélique. Ainsi, l’ICR du locus IGF2-H19 est méthylée uniquement sur l’allèle paternel, ce qui entraîne une expression paternelle du gène de croissance IGF2 et une expression maternelle du gène voisin H19 (Figure 2). Cette méthylation différentielle de l’ADN est acquise dans les cellules germinales via l’enzyme DNMT3A (DNA methyltransferase 3A) en collaboration avec un cofacteur spécifique, DNMT3L (DNA methyltransferase 3-like) (voir ci-dessous) [8, 9]. Il est maintenant admis que la méthylation différentielle de l’ADN joue un rôle clé dans le mécanisme de l’empreinte. Toutefois, ce n’est probablement pas la seule marque épigénétique impliquée. Notre laboratoire ainsi que d’autres, ont mis en évidence des modifications d’histone alléliques au niveau des ICR [10] : l’allèle méthylé est associé à des modifications d’histone particulières dites « répressives » telles que la triméthylation de la lysine 20 de l’histone H4 (H4K20me3) ou la triméthylation de la lysine 9 de l’histone H3 (H3K9me3), tandis que l’allèle non méthylé est associé à d’autres modifications dites « permissives », comme la diméthylation de la lysine 4 de l’histone H3 (H3K4me2) [11, 12]. Le rôle de ces modifications dans le mécanisme de l’empreinte fait l’objet de recherches intensives. Une des hypothèses privilégiées est leur implication dans la mise en place et/ou le maintien de la méthylation de l’ADN au niveau des ICR [13].
 |
Figure 1. Oligospermie : patrons de méthylation perturbés au niveau des ICR. L’oligozoospermie est caractérisée par une réduction drastique du nombre de spermatozoïdes dans le sperme. Plusieurs études récentes [25, 26] ont montré que ce phénotype est fréquemment associé à une perturbation de la méthylation de l’ADN au niveau des ICR. Dans des spermatozoïdes normaux, l’ICR du locus IGF2/H19 est méthylé (ronds rouge foncé) et des ICR à méthylation maternelle sont non méthylés (comme celui de ZAC1, un gène impliqué dans le diabète néonatal). À l’inverse, chez certains patients oligospermiques, l’ICR du locus IGF2/H19 est peu méthylé tandis que celui de ZAC1 présente des gains de méthylation. |
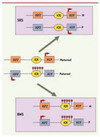 |
Figure 2. Perturbation de l’empreinte dans des syndromes liés à la croissance fœtale. Dans des cellules somatiques normales (au centre), l’ICR du locus IGF2-H19 est seulement méthylé (ronds noirs) sur l’allèle paternel. Cette méthylation allélique permet les expressions alléliques d’IGF2 à partir de l’allèle paternel et de H19 à partir de l’allèle maternel. Chez des patients atteints du syndrome de Beckwith-Wiedemann (BWS), la méthylation est présente sur les deux allèles ce qui entraîne une expression d’IGF2 à partir des deux allèles et la non expression de H19. À l’inverse, chez la moitié des patients atteints du syndrome de Silver-Russell (SRS), la méthylation n’est présente sur aucun des deux allèles ce qui entraîne la non expression d’IGF2 et l’expression de H19 à partir des deux allèles. |
Mise en place de l’asymétrie des génomes parentaux
L’asymétrie des génomes parentaux débute lors de la gamétogenèse. En effet, dans les cellules germinales embryonnaires murines, dès 12,5 jours post-coïtum (jpc), alors qu’une déméthylation globale du génome, y compris des ICR, a eu lieu, ces cellules commencent à se différencier selon le sexe de l’embryon. Une des premières différences que l’on peut observer est l’entrée en méiose dès 13,5 jpc des cellules germinales femelles qui restent ensuite bloquées en prophase alors que les cellules germinales mâles sont bloquées en phase G1 de la mitose et ne commenceront leur méiose que bien plus tard [14]. Par ailleurs, l’acquisition de la méthylation de l’ADN au niveau des ICR des gènes soumis à l’empreinte diffère également dans les deux lignées germinales. Bien que les protéines responsables de cette méthylation de novo (DNMT3A et DNMT3L) soient les mêmes chez les embryons mâles et les femelles, elles ne ciblent pas les mêmes séquences : dans la lignée germinale mâle, la méthylation concerne à la fois certains ICR et des rétro-éléments [15], tandis que dans la lignée germinale femelle, elle est acquise seulement au niveau d’autres ICR. Bien plus, la chronologie de l’acquisition de cette méthylation est très différente selon le sexe de l’embryon. En effet, dans les cellules germinales mâles, les premières séquences ICR méthylées sont détectées dès 15,5 jpc et seront complètement méthylées en période périnatale, ce qui implique une acquisition longue et progressive. À l’inverse, dans les cellules germinales femelles, l’acquisition de la méthylation de l’ADN se produit après la naissance, dans les ovocytes en phase de croissance [14]. Ainsi, tout au long de la gamétogenèse, les futurs génomes parentaux vont suivre deux chemins très différents, qui vont avoir des conséquences fonctionnelles au moment de la fécondation et durant le développement de l’embryon.
Lors de la fécondation, l’ovocyte ne terminera sa méiose qu’en expulsant le second globule polaire. Alors que le génome de l’ovocyte est empaqueté avec des histones, celui du spermatozoïde est empaqueté avec des protamines qui ont été incorporées à la fin de la spermatogenèse pour permettre une forte compaction du génome (→). Après la fécondation, le premier événement distinctif est le remplacement des protamines par des histones au niveau du génome paternel. Une vague de déméthylation permettant la reprogrammation des génomes parentaux se produit dans un deuxième temps : le génome paternel subit dès le stade zygote une déméthylation globale, rapide et active. Il faut noter que cette déméthylation épargne les régions ICR et certaines autres séquences comme les séquences satellites ou celles des rétrotransposons [16]. Les facteurs impliqués dans cet effacement de la méthylation de l’ADN sont inconnus à ce jour. Le génome maternel, quant à lui, est protégé de cette déméthylation active : il subira une déméthylation passive, au cours des premières divisions cellulaires de l’embryon. Ce mécanisme de protection est encore inconnu. Cependant, la différence de patron de modifications des histones entre les deux génomes pourrait jouer un rôle important à ce stade. En effet, les triméthylations de la lysine 9 et de la lysine 27 de l’histone H3 sont principalement détectées au niveau du génome maternel et pourraient protéger celui-ci du processus de déméthylation de l’ADN [16]. L’incorporation de variants d’histones tels que H3.3 pourrait aussi jouer un rôle important pour différencier les deux génomes [17]. Par ailleurs, d’autres travaux récents chez l’embryon précoce murin ont montré qu’une protéine appartenant à un complexe de remodelage de la chromatine, MBD3 [18], est impliquée dans le maintien de la méthylation au niveau de l’ICR paternel de H19. Cette étude apporte un premier indice pour comprendre ces mécanismes de protection de la méthylation des ICR dans les stades préimplantatoires. D’autres protéines produites au cours de l’ovogenèse, pourraient aussi être impliquées dans la protection du génome maternel. Actuellement, une seule de ces protéines a été identifiée : Stella, aussi appelée PGC7. Après sa localisation dans le pronucléus femelle, Stella semble protéger le génome maternel de la déméthylation et pourrait notamment prévenir la déméthylation de certains ICR [19]. Mais le mécanisme précis reste à élucider.
(→) Voir l’article de S. Rousseaux et al., page 735 de ce numéro
L’asymétrie entre les génomes paternel et maternel joue un rôle crucial lors du développement. La non-équivalence des deux génomes parentaux s’exprime notamment au niveau des gènes soumis à l’empreinte. Ainsi, des défauts dans l’acquisition ou le maintien des patrons épigénétiques des ICR peuvent entraîner différentes pathologies [20].
Pathologies liées à une perturbation de l’asymétrie des génomes parentaux
Perturbation de l’empreinte dans la lignée germinale
Chez l’homme, différentes études ont montré que ces marques spécifiques qui permettront l’asymétrie entre les génomes parentaux peuvent être perturbées dans les cellules germinales. L’un des cas les plus drastiques de pathologies liées à un défaut de la mise en place de l’empreinte nous est apporté par l’analyse des môles hydatidiformes [21]. Les môles hydatidiformes complètes font partie des maladies trophoblastiques gestationnelles. Elles donnent lieu à une prolifération excessive des villosités placentaires et à une absence d’embryon [22] et peuvent potentiellement se développer en choriocarcinomes. La plupart de ces môles résultent d’une fécondation dispermique et sont d’origine androgénique. Cependant, 20 % des cas sont d’origine biparentale [23]. Un de ces cas familiaux a été étudié en détail par Judson et al. [24] : les tissus issus de la môle présentaient une absence globale de méthylation au niveau des ICR qui ont une méthylation d’origine maternelle, mais pas au niveau de l’ICR du domaine IGF2-H19, dont la méthylation est d’origine paternelle. Cela suggère que cette pathologie est due à un défaut de mise en place de l’empreinte dans les cellules germinales femelles. Cependant, les études des cas familiaux de môle hydatidiforme complète montrent que DNMT3L et DNMT3A ne sont pas impliquées dans cette pathologie [25]. D’autres protéines pourraient être impliquées mais le mécanisme reste aujourd’hui un mystère.
Des analyses menées au cours de la spermatogenèse ont permis de mettre en évidence que l’infertilité masculine peut être liée à des défauts de l’empreinte génomique. Une étude a notamment été réalisée sur une centaine de patients oligospermiques [26]. Ces patients ont un nombre de spermatozoïdes pathologique, inférieur à 20 millions/ml, ce qui entraîne souvent une infertilité. Les analyses ont porté sur la méthylation de l’ADN de l’ICR du domaine IGF2-H19, qui est normalement fortement méthylé dans le sperme. Une baisse de la méthylation avait été observée dans le sperme de 25 % des patients oligospermiques (Figure 1). Par une étude similaire sur des spermatozoïdes pathologiques, une autre équipe a pu observer que sur une centaine d’hommes qui présentaient des problèmes d’infertilité, 14 % d’entre eux avaient une méthylation réduite au niveau des ICR paternels. D’une manière inattendue, un cinquième des patients présentaient également une méthylation aberrante au niveau des ICR maternels, alors que le reste du génome semblait correctement méthylé [27]. Ces études confirment qu’une dérégulation aberrante de la méthylation au niveau des gènes soumis à l’empreinte serait associée à l’oligospermie et à l’infertilité masculine. Les mécanismes impliqués semblent complexes et actuellement, il est impossible de déterminer si ces « mutations » épigénétiques sont dues à un défaut d’effacement ou d’acquisition des empreintes dans les cellules germinales mâles.
Syndromes liés à la croissance fœtale
Plusieurs syndromes liés à des anomalies de la croissance fœtale peuvent être causés par des perturbations de la méthylation de l’ADN au niveau des ICR, celles-ci se produisant lors des premiers stades du développement. En effet, après la fécondation et pendant le stade préimplantatoire a lieu une importante reprogrammation qui, si elle est défectueuse, pourrait être à l’origine de ces pathologies.
L’un de ces syndromes, le syndrome de Beckwith-Wiedemann (BWS), est caractérisé par une surcroissance foetale et des défauts de développement comme une macroglossie (grosse langue) ou une hypertrophie de certains organes. Par ailleurs, les enfants atteints de ce syndrome ont une prédisposition à développer des tumeurs et plus particulièrement une tumeur de Wilms qui se forme dans le rein. Ce syndrome est lié à une perte de fonction de l’allèle maternel du chromosome 11p15. Cette région chromosomique contient les domaines soumis à l’empreinte IGF2-H19 (Figure 2) et KCNQ1 qui ont respectivement des ICR à méthylation paternelle et maternelle. Chez de nombreux patients atteints du syndrome de BWS, la perte de fonction de l’allèle maternel est due à un défaut de méthylation au niveau de l’ICR qui régule le domaine KCNQ1. Cette perte de méthylation entraîne une répression biallélique du domaine et est corrélée à une diminution de l’expression du gène CDKN1C qui est un gène suppresseur de tumeur [28]. Par ailleurs, environ 10 % des patients présentent un gain de méthylation au niveau de l’ICR du domaine IGF2-H19 (Figure 2). Cela entraîne une expression biallélique du gène IGF2 qui est un facteur de croissance majeur.
Le syndrome de Silver-Russell (SRS) présente un phénotype contraire à celui du syndrome de BWS : retard de croissance intra-utérin et postnatal. Ce phénotype inversé s’explique par un défaut épigénétique opposé à celui du BWS. En effet, la moitié des patients atteints du SRS présentent une perte de méthylation au niveau de l’ICR qui régule le domaine IGF2-H19 (Figure 2). Cette perte de méthylation mène à la perte d’expression d’IGF2 et, en conséquence, à un retard de croissance [29].
Il est intéressant de noter que chez les jumeaux homozygotes [30], qui sont issus d’un œuf unique, il arrive que l’un des jumeaux soit atteint du syndrome SRS ou BWS, alors que l’autre est sain. Dans ces cas, la perturbation épigénétique a eu lieu durant le développement préimplantatoire après la scission de l’embryon. Le mécanisme précis de cette transformation reste inconnu ; il reste notamment à découvrir si des facteurs environnementaux comme le régime alimentaire durant la grossesse influencent ce processus.
Perturbation de l’empreinte et tumorigenèse
Les études décrites ci-dessus (choriocarcinome, tumeur de Wilms) ont été les premières à mettre en évidence qu’une perturbation de l’empreinte génomique pouvait mener à la tumorigenèse. Ces pertes de l’empreinte sont dues à l’absence de la méthylation différentielle entre les deux allèles. Elles se manifestent par une expression, ou une répression, biallélique d’un gène normalement soumis à l’empreinte et qui s’exprime habituellement à partir d’un seul allèle. Les mécanismes qui régissent ce défaut de méthylation différentielle sont actuellement inconnus. Pour y remédier, il serait important de décortiquer les mécanismes impliqués dans le maintien somatique de la méthylation différentielle. Des études sur le gène IGF2, premier gène pour lequel cette perte d’empreinte au cours de la tumorigenèse a été identifiée (Figure 3), ont permis des avancées importantes. IGF2 est notamment dérégulé dans de nombreuses tumeurs solides telles que le cancer du côlon, du foie, du poumon et du sein [31]. Cette surexpression (expression biallélique) d’IGF2 est souvent associée à un gain de méthylation au niveau de l’ICR de ce domaine soumis à l’empreinte (Figure 3). Cela n’est pas sans rappeler le gain de méthylation que subissent les gènes suppresseurs de tumeur au cours de la tumorigenèse [32, 33]. Selon l’hypothèse du « two-hit » de Knudson [34], l’inactivation des gènes suppresseurs de tumeurs se produirait en deux étapes : tout d’abord l’inactivation du promoteur du gène sur un allèle puis un deuxième évènement génétique ou épigénétique perturberait le second allèle. Si l’on fait le parallèle avec les gènes soumis à l’empreinte, il semble que ceux-ci aient une forte probabilité de jouer un rôle dans la tumorigenèse puisqu’il suffit que la perturbation, notamment de la méthylation, se produise sur un seul allèle pour que l’expression du gène soit totalement changée. Par ailleurs, de nombreux gènes soumis à l’empreinte sont impliqués dans la prolifération cellulaire et apporteront un avantage prolifératif à la cellule cancéreuse, s’ils sont surexprimés. D’autre part, le gène soumis à l’empreinte CDKN1C impliqué dans le cycle cellulaire est un gène suppresseur de tumeur, et il est souvent inactivé dans des tumeurs via l’acquisition de la méthylation de l’ADN sur l’allèle normalement non méthylé [35]. Plusieurs études chez la souris et chez l’Homme [36, 37] montrent aussi que la perte de l’empreinte d’IGF2 constitue un véritable risque de développement de tumeur, notamment quand elle est associée à des événements d’ordre génétique par exemple. Ces données impliquent que les gènes soumis à l’empreinte, de par leur fonction et leur mécanisme de régulation, ont de fortes chances d’être impliqués dans des cancers. Mais là encore, les mécanismes restent énigmatiques. Certaines études suggèrent que les modifications des histones pourraient participer à la mise en place de la méthylation de l’ADN au niveau des gènes suppresseurs de tumeur [38]. Cette piste d’exploration pourrait aussi s’avérer prometteuse au niveau des gènes soumis à l’empreinte.
 |
Figure 3. Méthylation de novo au cours de la tumorigenèse. Dans les cellules tumorales, un parallèle peut être fait entre l’acquisition de la méthylation d’ADN (ronds noirs) au niveau des ICR des gènes soumis à l’empreinte tels que celui du locus IGF2-H19 et celle qui s’effectue au niveau des promoteurs des gènes suppresseurs de tumeur tels que p16 (INK4a). Au niveau de ces locus, on trouve notamment des marques chromatiniennes répressives telles que la triméthylation de la lysine 9 de l’histone H3. |
Conclusions
L’asymétrie des génomes parentaux, principalement caractérisée par les gènes soumis à l’empreinte, joue un rôle primordial au cours du développement des mammifères. En effet, la dérégulation des mécanismes qui régissent ce phénomène entraîne diverses pathologies chez l’Homme dont la tumorigenèse. Il est donc capital dans les années à venir de comprendre les mécanismes d’établissement et de maintien de cette asymétrie. L’un des objectifs principaux est d’identifier les machineries enzymatiques impliquées, et de comprendre comment celles-ci sont recrutées vers leurs cibles, d’une manière allélique. Il est notamment possible que la spécificité germinale soit déterminée par des protéines reconnaissant des séquences spécifiques de l’ADN. Une hypothèse alternative serait que l’expression de petits ARN soit impliquée dans cette spécificité, une possibilité suggérée par des études chez la levure et les plantes [39]. Des mécanismes similaires, ainsi que des modifications des histones, pourraient être impliqués, dans les cellules somatiques, dans le maintien de l’asymétrie épigénétique entre les génomes parentaux. Il faut attendre que les acteurs principaux soient connus pour commencer à explorer leur expression dans le contexte des différents cancers. Cela permettra de mieux comprendre les causes et les conséquences des dérégulations de l’expression mono-allélique des gènes et leur impact dans les maladies humaines.
Références
- McGrath J, Solter D. Completion of mouse embryogenesis requires both the maternal and paternal genomes. Cell 1984; 37 : 179–83. [Google Scholar]
- Surani MA, Barton SC, Norris ML. Development of reconstituted mouse eggs suggests imprinting of the genome during gametogenesis. Nature 1984; 308 : 548–50. [Google Scholar]
- Thomson, JASolter D. The developmental fate of androgenetic, parthenogenetic, and gynogenetic cells in chimeric gastrulating mouse embryos. Genes Dev 1988; 10 : 1344–51. [Google Scholar]
- Gabory A, Dandolo L. Épigénétique et développement : l’empreinte parentale. Med. Sci. (Paris) 2005; 21 : 390–5 [Google Scholar]
- Cattanach BM, Kirk M. Differential activity of maternally and paternally derived chromosome regions in mice. Nature 1985; 315 : 496–8. [Google Scholar]
- Hutter B, Helms V, Paulsen M. Tandem repeats in the CpG islands of imprinted genes. Genomics 2006; 88 : 323–32. [Google Scholar]
- Kobayashi H, Suda C, Abe T, et al. Bisulfite sequencing and dinucleotide content analysis of 15 imprinted mouse differentially methylated regions (DMRs) : paternally methylated DMRs contain less CpGs than maternaly methylated DMRs. Cytogenet Genome Res 2006; 113 : 130–7. [Google Scholar]
- Bourc’his D, Xu GL, Lin CS, et al. Dnmt3L and the establishment of maternal genomic imprints. Science 2002; 294 : 2536–9. [Google Scholar]
- Kaneda M, Okano M, Hata K, et al. Essential role for de novo DNA methyltransferase Dnmt3a in paternal and maternal imprinting. Nature 2004; 429 : 900–3. [Google Scholar]
- Ray-Gallet D, Gérard A, Polo S, Almouzni G. Variations sur le theme du code histone. Med Sci (Paris) 2005; 21 : 384–9. [Google Scholar]
- Fournier C, Goto Y, Ballestar E, et al. Allele-specific histone lysine methylation marks regulatory regions at imprinted mouse genes. EMBO J 2002; 21 : 6560–70. [Google Scholar]
- Delaval K, Govin J, Cerqueira F, et al. Differential histone modifications mark mouse imprinting control regions during spermatogenesis. EMBO J 2007; 26 : 720–9. [Google Scholar]
- Delaval K, Feil R. Epigenetic regulation of mammalian genomic imprinting. Curr Opin Genet Dev 2004; 14 : 188–95. [Google Scholar]
- Sasaki H, Matsui Y. Epigenetic events in mammalian germ-cell development : reprogramming and beyond. Nat Rev Genet 2008; 9 : 129–40. [Google Scholar]
- Kato Y, Kaneda M, Hata K, et al. Role of the Dnmt3 family in de novo methylation of imprinted and repetitive sequences during male germ cell development in the mouse. Hum Mol Genet 2007; 16 : 2272–80. [Google Scholar]
- Morgan, HD, Santos F, Green K, et al. Epigenetic reprogramming in mammals. Hum Mol Genet 2005; 14 (suppl 1) : R47–58. [Google Scholar]
- Van der Heijden, GW, Dieker JW, Derijck AA, et al. Asymmetry in histone H3 variants and lysine methylation between paternal and maternal chromatin of the early embryo. Mech Dev 2005; 122 : 1008–22. [Google Scholar]
- Reese KJ, Lin S, Verona RI, et al. Maintenance of paternal methylation and repression of the imprinted H19 gene requires MBD3. PLoS Genet 2007; 3 : 137. [Google Scholar]
- Nakamura T, Arai Y, Umehara H, et al. PGC7/Stella protects against DNA demethylation in early embryogenesis. Nat Cell Biol 2007; 9 : 64–71. [Google Scholar]
- Arnaud P, Feil R. Epigenetic deregulation of genomic imprinting in human disorders and following assisted reproduction. Birth Defects Res C Embryo Today 2005; 75 : 81–97. [Google Scholar]
- Gilgenkrantz S. À la recherche des empreintes perdues : les épigénotypes anormaux. Med Sci (Paris) 2003; 19 : 15–8. [Google Scholar]
- Van den Veyver IB, Al-Hussaini TK. Biparental hydatidiform moles : a maternal effect mutation affecting imprinting in the offspring. Hum Reprod Update 2006; 12 : 233–42. [Google Scholar]
- Kovacs BW, Shahbahrami B, Tast DE, Curtin JP. Molecular genetic analysis of complete hydatidiform moles. Cancer Genet Cytogenet 1991; 54 : 143–52. [Google Scholar]
- Judson H, Hayward BE, Sheridan E, Bonthron DT. A global disorder of imprinting in the human female germ line. Nature 2002; 416 : 539–42. [Google Scholar]
- Hayward BE, De Vos M, Judson H, et al. Lack of involvement of known DNA methyltransferases in familial hydatidiform mole implies the involvement of other factors in establishment of imprinting in the human female germline. BMC Genet 2003; 4 : 2. [Google Scholar]
- Marques CJ, Carvalho F, Sousa M, Barros A. Genomic imprinting in disruptive spermatogenesis. Lancet 2004; 363 : 1700–2. [Google Scholar]
- Kobayashi H, Sato A, Otsu E, et al. Aberrant DNA methylation of imprinted loci in sperm from oligospermic patients. Hum Mol Genet 2007; 16 : 2542–51. [Google Scholar]
- Diaz-Meyer N, Day CD, Khatod K, et al. Silencing of CDKN1C (p57KIP2) is associated with hypomethylation at KvDMR1 in Beckwith-Wiedemann syndrome. J Med Genet 2003; 40 : 797–801. [Google Scholar]
- Gicquel C, Rossignol S, Cabrol S, et al. Epimutation of the telomeric imprinting center region on chromosome 11p15 in Silver-Russell syndrome. Nat Genet 2005; 37 : 1003–7. [Google Scholar]
- Weksberg R, Shuman C, Caluseriu O, et al. Discordant KCNQ1OT1 imprinting in sets of monozygotic twins discordant for Beckwith-Wiedemann syndrome. Hum Mol Genet 2002; 11 : 1317–25. [Google Scholar]
- Feinberg AP, Cui H, Ohlsson R. DNA methylation and genomic imprinting : insights from cancer into epigenetic mechanisms. Semin Cancer Biol 2002; 12 : 389–98. [Google Scholar]
- Jones PA, Baylin SB. The epigenomics of cancer. Cell 2007; 128 : 683–92. [Google Scholar]
- Pannetier M, Feil R. Epigenetic stability of embryonic stem cells and developmental potential. Trends Biotechnol 2007; 25 : 556–62. [Google Scholar]
- Garinis GA, Patrinos GP, Spanakis NE, Menounos PG. DNA hypermethylation : when tumour suppressor genes go silent. Hum Genet 2002; 111 : 115–27. [Google Scholar]
- Higashimoto K, Soejima H, Saito T, et al. Imprinting disruption of the CDKN1C/KCNQ1OT1 domain : the molecular mechanisms causing Beckwith-Wiedemann syndrome and cancer. Cytogenet Genome Res 2006; 113 : 306–12. [Google Scholar]
- Sakatani T, Kaneda A, Iacobuzio-Donahue CA, et al. Loss of imprinting of Igf2 alters intestinal maturation and tumorigenesis in mice. Science 2005; 307 : 1976–8. [Google Scholar]
- Cui H, Cruz-Correa M, Giardiello FM, et al. Loss of IGF2 imprinting : a potential marker of colorectal cancer risk. Science 2003; 299 : 1753–5. [Google Scholar]
- Bachman KE, Park BH, Rhee I, et al. Histone modifications and silencing prior to DNA methylation of a tumor suppressor gene. Cancer Cell 2003; 3 : 89–95. [Google Scholar]
- Zaratiegui M, Irvine DV, Martienssens RA. Noncoding RNAs and gene silencing. Cell 2007; 128 : 763–76. [Google Scholar]
Liste des figures
 |
Figure 1. Oligospermie : patrons de méthylation perturbés au niveau des ICR. L’oligozoospermie est caractérisée par une réduction drastique du nombre de spermatozoïdes dans le sperme. Plusieurs études récentes [25, 26] ont montré que ce phénotype est fréquemment associé à une perturbation de la méthylation de l’ADN au niveau des ICR. Dans des spermatozoïdes normaux, l’ICR du locus IGF2/H19 est méthylé (ronds rouge foncé) et des ICR à méthylation maternelle sont non méthylés (comme celui de ZAC1, un gène impliqué dans le diabète néonatal). À l’inverse, chez certains patients oligospermiques, l’ICR du locus IGF2/H19 est peu méthylé tandis que celui de ZAC1 présente des gains de méthylation. |
| Dans le texte | |
 |
Figure 2. Perturbation de l’empreinte dans des syndromes liés à la croissance fœtale. Dans des cellules somatiques normales (au centre), l’ICR du locus IGF2-H19 est seulement méthylé (ronds noirs) sur l’allèle paternel. Cette méthylation allélique permet les expressions alléliques d’IGF2 à partir de l’allèle paternel et de H19 à partir de l’allèle maternel. Chez des patients atteints du syndrome de Beckwith-Wiedemann (BWS), la méthylation est présente sur les deux allèles ce qui entraîne une expression d’IGF2 à partir des deux allèles et la non expression de H19. À l’inverse, chez la moitié des patients atteints du syndrome de Silver-Russell (SRS), la méthylation n’est présente sur aucun des deux allèles ce qui entraîne la non expression d’IGF2 et l’expression de H19 à partir des deux allèles. |
| Dans le texte | |
 |
Figure 3. Méthylation de novo au cours de la tumorigenèse. Dans les cellules tumorales, un parallèle peut être fait entre l’acquisition de la méthylation d’ADN (ronds noirs) au niveau des ICR des gènes soumis à l’empreinte tels que celui du locus IGF2-H19 et celle qui s’effectue au niveau des promoteurs des gènes suppresseurs de tumeur tels que p16 (INK4a). Au niveau de ces locus, on trouve notamment des marques chromatiniennes répressives telles que la triméthylation de la lysine 9 de l’histone H3. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




