| Issue |
Med Sci (Paris)
Volume 23, Number 11, Novembre 2007
|
|
|---|---|---|
| Page(s) | 1008 - 1013 | |
| Section | M/S revues | |
| DOI | https://doi.org/10.1051/medsci/200723111008 | |
| Published online | 15 November 2007 | |
Effets fondateurs et variabilité génétique au Québec
Founder effects and genetic variability in Quebec
1
Centre de recherche, CHU Sainte-Justine, 3175, chemin de la Côte Sainte-Catherine, Montréal, Québec, H3T 1C5, Canada
2
Département de pédiatrie, Université de Montréal, Montréal (Québec) Canada
3
Groupe de recherche interdisciplinaire en démographie et épidémiologie génétique (GRIG), Département des sciences humaines, Université du Québec à Chicoutimi, 555, boulevard de l’Université, Chicoutimi (Québec) G7H 2B1, Canada
Au Québec, l’effet fondateur et ses conséquences sont souvent évoqués dans les études génétiques. La caractérisation de cet effet fondateur s’est beaucoup appuyée sur la distribution observée des maladies monogéniques : une grande partie des travaux portant sur la région du Saguenay. Pour obtenir une image plus globale de la diversité génétique, nous avons entrepris un programme de recherche sur l’ensemble des populations régionales du Québec visant l’examen de marqueurs neutres de plusieurs systèmes génétiques. Les résultats présentés ici indiquent que la population du Québec ne semble ni plus homogène ni très différente des populations européennes. On y observe cependant une série d’effets fondateurs régionaux surtout visibles au sein des systèmes génétiques qui contiennent des variants peu fréquents et plus ou moins marqués selon les caractéristiques des histoires démographiques régionales.
Abstract
Knowledge of the genetic population structure lies at the heart of mapping studies aiming genes responsible for Mendelian and complex traits. The Quebec population, which is of mostly French descent, is considered an excellent model for such genetic epidemiological endeavours because it is a young founder population. Yet, the assessment of the founder effect has relied mostly on the observed distribution of monogenic diseases and on the analysis of the underlying mutations with investigations focusing on the Saguenay region. To eliminate this clinical bias and to obtain a more complete image of the genetic diversity, different regional populations of Quebec were investigated by analysing neutral markers that represent maternal, paternal and X chromosome lineages. Results indicate that Quebec does not appear more homogeneous nor significantly different from European populations. However, a series of regional founder effects, particularly visible at the level of rare variants, are observed. These effects can be explained by the successive migrations of descendants of the first immigrants from the initial sites of settlement towards the outer regions. Depending on the number of founders and their diversity, as well as on the degree of isolation and the magnitude of the interbreeding with the neighbouring or local populations, such as Amerindians or later migrants, the consequences of these regional founder effects are more or less detectable in the contemporary population.
© 2007 médecine/sciences - Inserm / SRMS
Au Québec, l’histoire du peuplement et ses conséquences sur le patrimoine génétique de la population contemporaine suscitent beaucoup d’intérêt. La formation de la population d’origine européenne remonte à seulement quelques siècles et certaines populations régionales se sont constituées dans un relatif isolement. L’existence de données généalogiques de qualité exceptionnelle offre un contexte favorable pour étudier la relation entre l’histoire démographique du Québec et sa diversité génétique. Jusqu’à maintenant, les travaux se sont beaucoup appuyés sur l’étude des maladies monogéniques et se sont concentrés sur la région du Saguenay-Lac-St-Jean (appelée Saguenay dans la suite du texte) [1–3]. Pour obtenir une image plus globale de la diversité génétique, nous avons entrepris un programme de recherche portant sur l’ensemble des populations régionales du Québec en débutant avec l’étude de la Gaspésie, une région maritime qui se caractérise par la diversité des fondateurs d’origine européenne venus s’y installer. Nous présentons ici des résultats provenant de cette région, ainsi que de celles du Saguenay et de Montréal. Des comparaisons ont aussi été effectuées avec des échantillons français et européens.
Des histoires de peuplement diversifiées : la Gaspésie, le Saguenay et Montréal
La population québécoise contemporaine compte environ 6 millions de francophones qui sont en grande majorité des descendants de quelque 10 000 immigrants, pour la plupart venus de France, et installés en Nouvelle-France entre la fondation de Québec, en 1608, et la conquête anglaise de 1760 [4]. Ce nombre se restreint à 8 500 si l’on compte seulement ceux qui ont laissé au moins un enfant marié dans la colonie (B. Desjardins, résultats non publiés). L’occupation du territoire s’est faite progressivement dans les régions situées de part et d’autre du fleuve Saint-Laurent.
Après la Conquête, l’immigration française au Québec a pratiquement cessé mais les Canadiens-français ont connu une forte croissance démographique grâce à un taux de fécondité élevé [5]. Au cours du XIXe siècle, cette croissance a entraîné l’ouverture de nouvelles régions telles que le Saguenay situé à 200 kilomètres au nord de Québec (Figure 1). Son peuplement a débuté vers 1840 avec l’arrivée de pionniers provenant de la région voisine de Charlevoix et, par la suite, d’autres régions de la vallée laurentienne. La population est ainsi passée d’environ 5 000 habitants en 1850 à 275 000 aujourd’hui [6, 7].
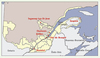 |
Figure 1. Représentation des principales zones d’occupation du territoire québécois (en jaune) et localisation des régions étudiées. |
En ce qui concerne la Gaspésie, région située à l’est de la province (Figure 1), l’occupation permanente du territoire a commencé après la conquête anglaise de 1760. En effet, quelques familles acadiennes s’y sont réfugiées après leur déportation en 1755. Les Acadiens descendent des pionniers français qui se sont installés aux XVIIe et XVIIIe siècles en Acadie, aujourd’hui la Nouvelle-Écosse et une partie du Nouveau-Brunswick. Puis, avec la guerre d’indépendance américaine, des Loyalistes y ont à leur tour trouvé asile. Au cours de la même période, l’industrie de la pêche a attiré plusieurs centaines d’habitants - surtout des hommes - provenant des îles Anglo-Normandes. Enfin, tout au long du XIXe siècle, des Canadiens-français de la vallée du Saint-Laurent s’installent en Gaspésie, ainsi que des Écossais, des Irlandais et des Anglais venus avec le développement de l’industrie navale et du commerce du bois [8]. Aujourd’hui, la Gaspésie compte environ 80 000 habitants dont 87 % déclarent le français comme langue maternelle [9].
Ces deux régions, la Gaspésie et le Saguenay, évoquent donc des scénarios de peuplement très différents. L’une a été colonisée sur une assez longue période par des fondateurs d’origines diverses et l’autre a été fondée sur une période plus restreinte par des pionniers d’origine française issus d’une région adjacente. Quant à la région métropolitaine de Montréal, son peuplement a débuté avec la fondation de la ville, en 1642 ; il s’agit aujourd’hui d’une métropole de plus de 3 millions d’habitants provenant du monde entier. Parmi les Canadiens-français on retrouve des descendants des pionniers installés sous le Régime français mais aussi des habitants de toutes les régions du Québec venus y vivre au cours des XIXe et XXe siècles.
L’effet fondateur : mythes et réalités
L’effet fondateur est lié à l’établissement d’une nouvelle population sur un territoire neuf. Il se caractérise par un changement du profil des fréquences alléliques du groupe migrant par rapport à sa population d’origine [10]. Il s’explique par la stochastique de l’échantillonnage des chromosomes et devient d’autant plus important lorsque le nombre des migrants est petit et que le groupe des migrants est formé de fratries ou de familles [11]. Au cours des générations qui suivent la migration, la dérive génétique peut aussi modifier les fréquences alléliques et l’ampleur de son effet sera fonction de l’effectif et de la dynamique démographique de la nouvelle population. Quand on parle de l’effet fondateur au Québec, on pense d’abord aux maladies mendéliennes qui y sont particulièrement fréquentes par rapport aux autres populations [1–3]. Ainsi, une douzaine de maladies héréditaires ont été observées dans la région du Saguenay et leur identification est à l’origine du paradigme saguenéen de l’effet fondateur québécois.
Mais ce paradigme saguenéen est-il applicable à l’ensemble du Québec ? L’effet fondateur comporte dans sa définition la notion de goulot d’étranglement démographique qui indique une importante réduction de taille de la population fondatrice par rapport à la population mère [10]. Cette notion et les conséquences qu’on lui attribue ont pu mener au mythe d’homogénéité et de réduction de la diversité génétique des Canadiens-français [12]. Cependant, avec quelques milliers de fondateurs, une perte importante de diversité génétique en quelques générations est inconcevable. En revanche, le peuplement des régions éloignées des pôles de colonisation initiale a été le fait dans plusieurs cas d’un nombre plus restreint de pionniers qui, de surcroît, ont fréquemment migré par familles [13, 14]. Ainsi, au Saguenay, les immigrants en provenance de Charlevoix sont arrivés les premiers, ont été plus nombreux à s’installer définitivement dans la région et ont réussi à y implanter un plus grand nombre de leurs descendants [15, 16]. Il a aussi été démontré que les comportements reproductifs associés à une plus grande fécondité ont favorisé certaines familles d’une génération à l’autre renforçant ainsi l’effet fondateur [17]. Ces caractéristiques du peuplement ont permis à certains allèles rares de s’exprimer alors qu’ils étaient demeurés masqués parmi les immigrants d’origine [18, 19]. Ainsi, au Saguenay et à Charlevoix, on observe des allèles récessifs d’une fréquence de l’ordre de 1/50 ou moins (taux des porteurs ≤ 1/25), qui sont devenus les témoins de l’effet fondateur à cause de leur impact clinique [1, 2].
On observe aussi des concentrations de certaines maladies héréditaires ailleurs au Québec, comme la dystrophie oculopharyngée dans la région de Montmagny-L’Islet [20], la névrite héréditaire causée par deux mutations distinctes dans Lanaudière [21] ou la maladie de Tay-Sachs dans le Bas St-Laurent [22]. Cette régionalisation de l’effet fondateur, vue sous l’angle des maladies héréditaires, permet donc de se questionner sur l’homogénéité de la constitution génétique des Québécois d’ascendance française. Comme le mentionne C.R. Scriver [2], la distribution des mutations responsables des maladies mendéliennes plus « communes », comme la phénylcétonurie, l’hypercholestérolémie familiale ou la déficience en protéine lipase, va aussi à l’encontre de l’isolement génétique de cette population. Tout ceci nous amène à croire qu’au Québec, la distribution des maladies mendéliennes et des mutations qui les expliquent ressemble plutôt à la mosaïque des effets fondateurs observée en Finlande [23]. Au lieu d’évoquer l’effet fondateur panquébécois afin de justifier cette mosaïque non aléatoire des mutations particulières, il faut plutôt considérer des séquences migratoires telles que France – région de Québec – Charlevoix – Saguenay ou alors France – Acadie – Gaspésie.
Par ailleurs, les répercussions de l’effet fondateur sur le pool génique d’une population dépendent aussi de la diversité initiale du système génétique examiné. La variance d’échantillonnage est beaucoup plus grande pour des allèles rares. L’effet fondateur pourrait ainsi être resté sans conséquence sur la fréquence des polymorphismes communs dont l’importance dans l’étiologie des phénotypes associés aux traits complexes ou à la réponse thérapeutique au traitement est souvent soulevée.
La variabilité génétique au Québec
L’étude de la distribution des maladies mendéliennes offre donc un tableau incomplet de la diversité génétique au Québec. Pour éliminer un tel biais clinique et obtenir une image plus globale de la variabilité génétique, il est nécessaire d’examiner des marqueurs neutres représentant l’ensemble du spectre des fréquences alléliques. Plutôt que de considérer différents segments autosomiques pour caractériser la diversité, nous avons choisi d’exploiter les systèmes modèles. Ainsi, nous avons comparé nos échantillons gaspésiens, saguenéens et montréalais avec des données provenant de France, d’Angleterre et d’Irlande, pour la portion hypervariable de l’ADN mitochondrial, qui représente les lignées maternelles, et pour l’haplotype composé de microsatellites du chromosome Y, transmis par les pères à leurs fils [24]. De plus, nous avons examiné les échantillons québécois et français pour la variabilité d’un microsatellite complexe du chromosome X, DXS1238 et d’un haplotype adjacent composé de 35 polymorphismes bi-alléliques, dys-44 [25–27]. La variabilité génétique a été évaluée à l’aide de la diversité génique (hétérozygotie des haplotypes), du nombre d’haplotypes et du paramètre de diversité Θ (estimé à partir de l’homozygotie haplotypique) [28, 29]. Le Tableau I indique que cette diversité est similaire dans les populations du Québec et de l’Europe. De même, la distribution des haplotypes selon leur fréquence (nombre d’apparitions dans un échantillon) est très semblable dans les populations québécoises et françaises pour les quatre systèmes étudiés, tel que l’illustre la Figure 2 pour l’haplotype combiné dys-44-DXS1238. La principale différence que l’on observe sur ce graphique est un léger déplacement vers la droite de l’histogramme québécois où les deux haplotypes les plus fréquents sont observés à 49 et 23 reprises (18 % et 8 %) tandis qu’on ne compte que 38 et 18 copies (15 % et 7 %) des haplotypes correspondants en France. On a aussi fait la distinction entre les haplotypes observés dans les deux populations et ceux qui sont exclusifs à une population. Les haplotypes spécifiques se retrouvent essentiellement parmi les singletons, mais aussi parmi les classes d’haplotypes qui surviennent deux ou trois fois. Dans son ensemble, le Québec n’apparaît donc ni plus homogène en ce qui a trait au nombre et à la fréquence des variants génétiques, ni très différent de la France considérant que la majorité des variants présents à plus d’une copie est partagée par les deux populations.
Statistiques descriptives de la diversité génétique pour le segment hypervariable 1, SHV1, de l’ADN mitochondrial, l’haplotype composé de sept microsatellites, STR, du chromosome Y et les deux marqueurs du chromosome X. Les échantillons québécois analysés pour SHV1 provenaient du Saguenay (n = 169), de Montréal (n = 63) et de la Gaspésie représentée par les descendants des Acadiens (n = 97), des Canadiens-français (n = 95), des Loyalistes (n = 85) et des Anglo-normands (n = 88). Les mêmes groupes étaient représentés dans l’analyse du chromosome Y (n = 95, 96, 44, 35, 42 et 54 respectivement) ainsi que des marqueurs DXS1238 (n = 83, 65, 45, 32, 40 et 54) et dys-44 (n = 69, 36, 45, 33, 40 et 54).
 |
Figure 2. Histogramme des haplotypes composés des deux marqueurs du chromosome X en France (n = 257) et au Québec (n = 276), regroupés selon leur fréquence absolue dans chaque échantillon. Les couleurs pâles indiquent la portion des haplotypes exclusifs, retrouvés seulement au Québec ou seulement en France. L’haplotype le plus fréquent est le même dans les deux populations mais il apparaît un peu plus souvent au Québec (49) qu’en France (38). |
La comparaison des échantillons du Saguenay, de Montréal et des quatre populations gaspésiennes fait ressortir des différences à l’échelle des régions québécoises (Figures 3 et 4). Ces différences sont plus marquées si l’on considère la variabilité de l’ADN mitochondrial et celle du chromosome Y car ces deux systèmes sont plus sensibles à l’effet fondateur et à la dérive génétique que les marqueurs du chromosome X ou autosomiques. La distribution des haplotypes du chromosome Y regroupés selon leur fréquence dans les échantillons de Montréal et du Saguenay montre une diminution des singletons dans la région saguenéenne et, par conséquent, un plus grand nombre d’haplotypes à des fréquences plus élevées (Figure 3). Cependant, ces différences ne s’avèrent pas significatives (test de Kolmogorov-Smirnov).
 |
Figure 3. Histogramme des haplotypes composés de sept marqueurs microsatellites du chromosome Y pour les régions de Montréal (n = 96) et du Saguenay (n = 95), regroupés selon leur fréquence absolue dans chaque échantillon. Le nombre des haplotypes uniques (fréquence de 1) est beaucoup plus bas chez les Saguenéens déplaçant ainsi l’histogramme vers la droite avec l’haplotype le plus fréquent représenté en 21 copies. |
 |
Figure 4. Mesure de distance génétique Fst par paires de populations québécoises. Mesure représentée (voir http://cmpg.unibe.ch/software/arlequin3) sur un graphique MDS (MultiDimensional Scaling ; voir Statistica – http://www.statsoft.com) pour les trois systèmes génétiques étudiés chez les Gaspésiens d’origine anglo-normande (IAN), d’origine acadienne (Ac), d’origine loyaliste (Loy) et d’origine canadienne-française (C-f) ainsi que chez les Canadiens-français de Montréal (Mtl) et du Saguenay (SLSJ). Les Saguenéens se différencient significativement (*) pour tous les systèmes, les Acadiens pour l’ADN mt et le chromosome Y et les Loyalistes pour le chromosome Y seulement. |
Les distances génétiques entre les populations prises deux à deux ont été mesurées à l’aide de l’index de différenciation FST [24, 29, 30] pour les trois systèmes étudiés (Figure 4). Comme on pouvait s’y attendre considérant les caractéristiques de son histoire démographique, le Saguenay se distingue des autres régions pour tous les systèmes représentés. Les Acadiens de Gaspésie se démarquent aussi, mais seulement pour l’ADN mitochondrial et le chromosome Y. Les Acadiens sont souvent venus en Gaspésie en famille et, par la suite, ces familles semblent s’être moins mêlées aux autres groupes ce qui pourrait expliquer ces différences. Les descendants des Loyalistes se distinguent, quant à eux, seulement pour le chromosome Y. Les deux autres groupes gaspésiens, soit les Canadiens-français et les descendants des Anglo-normands, sont similaires sur le plan génétique. Ici encore, nos résultats génétiques sont appuyés par l’histoire démographique puisque les immigrants anglo-normands étaient principalement des hommes et devaient donc trouver leurs partenaires dans la population locale [8]. On observe donc des différences marquées parmi les populations régionales étudiées. Une certaine stratification se manifeste au sein d’une région comme la Gaspésie, lorsqu’on regroupe les individus représentant les descendants des différents groupes de fondateurs. Fait intéressant, on retrouve dans l’échantillon gaspésien 10 % de mitochondries d’origine amérindienne provenant principalement de deux femmes ayant vécu au XVIIIe siècle tandis que dans les autres régions, on en dénombre moins de 1 %. Chez les Gaspésiens, nous avons aussi identifié deux copies d’un haplotype dys-44 d’origine africaine qui témoigne, encore une fois, d’un enrichissement de la variabilité génétique au sein du pool génique.
Conclusion
Au Québec, on observe une série d’effets fondateurs régionaux survenus à la suite du peuplement français initial. Ces effets sont surtout visibles au sein des systèmes génétiques qui contiennent des variants peu fréquents. Les migrations successives des descendants des premiers pionniers vers les régions nouvellement ouvertes sont à l’origine de ce phénomène de régionalisation. Cependant, selon le nombre de fondateurs et leur diversité, selon le degré d’isolement et l’ampleur du métissage avec d’autres populations locales comme les Amérindiens ou avec des migrants arrivés plus tardivement, les conséquences des effets fondateurs régionaux sont observables à des degrés divers dans la population contemporaine. La caractérisation des bassins génétiques régionaux se poursuit mais elle suggère d’ores et déjà que les spécificités régionales devraient être prises en compte dans les études épidémiogénétiques portant sur des maladies mendéliennes ou complexes.
Remerciements
Les auteurs tiennent à remercier les participants des diverses régions du Québec ainsi que Sylvie Bourthoumieu, Pascale Gerbault, Peter de Knijff, Catherine Laprise, Jean-Paul Moisan, Daniel Sinnett pour leur collaboration au projet dans la collecte des données ou dans les travaux de séquençage et de génotypage. Ils offrent aussi leurs remerciements aux évaluateurs anonymes pour leurs commentaires et leurs suggestions fort appréciés. Ils tiennent enfin à souligner le soutien financier du Réseau de médecine génétique appliquée du Fonds de recherche en santé du Québec.
Références
- Laberge AM, Michaud J, Richter A, et al. Population history and its impact on medical genetics in Quebec. Clin Genet 2005; 68 : 287–301. [Google Scholar]
- Scriver CR. Human genetics: lessons from Quebec populations. Annu Rev Genomics Hum Genet 2001; 2: 69–101. [Google Scholar]
- Yotova V, Labuda D, Zietkiewicz E, et al. Anatomy of a founder effect: myotonic dystrophy in Northeastern Quebec. Hum Genet 2005; 117 : 177–87. [Google Scholar]
- Charbonneau H, Desjardins B, Légaré J, Denis H. The population of the St-Lawrence Valley, 1608-1760. In : Haines MR, Steckel RH, eds. A population history of North America. New York : Cambridge University Press, 2000 : 99–142. [Google Scholar]
- Henripin J, Péron Y. La transition démographique de la province de Québec. In : Charbonneau H, ed. La population du Québec: études rétrospectives. Trois-Rivières: Éditions du Boréal Express, 1973 : 23–44. [Google Scholar]
- Pouyez C, Lavoie Y. Les Saguenayens. Introduction à l’histoire des populations du Saguenay. Québec : Presses de l’Université du Québec, 1983. [Google Scholar]
- Institut de la statistique du Québec. Gouvernement du Québec. http://www.stat.gouv.qc.ca/regions/profils/region_02_00.htm, 2006. [Google Scholar]
- Desjardins M, Frenette Y, Bélanger J, Hétu B. Histoire de la Gaspésie. Québec : Les presses de l’Université Laval, 1999 : 796 p. [Google Scholar]
- Statistique Canada. Gouvernement du Canada. http://www12.statcan.ca/francais/census01/home/Index.cfm, 2006. [Google Scholar]
- Mayr E. Animal species and evolution. Cambridge: Harvard University Press, 1963 : 812 p. [Google Scholar]
- Fix AG. Migration and colonization in human microevolution. Cambridge, UK : Cambridge University Press, 1999 : 254 p. [Google Scholar]
- Bouchard G. Information génétique et risque de stigmatisation collective. L’exemple du Saguenay-Lac-St-Jean. Med Sci (Paris) 2004; 20 : 933–4. [Google Scholar]
- Jetté R, Gauvreau D, Guérin M. Aux origines d’une région: le peuplement fondateur de Charlevoix avant 1850. In : Bouchard G, de Braekeleer M, eds. Histoire d’un génome. Population et génétique dans l’est du Québec. Québec : Presses de l’Université du Québec, 1991 : 75–106. [Google Scholar]
- Gagnon A, Heyer E. Fragmentation of the Quebec population genetic pool (Canada): evidence from the genetic contribution of founders per region in the 17th and 18th centuries. Am J Phys Anthropol 2001; 114 : 30–41. [Google Scholar]
- Gauvreau D, Guérin M, Hamel M. De Charlevoix au Saguenay : mesure et caractéristiques du mouvement migratoire avant 1911. In : Bouchard G, de Braekeleer M, eds. Histoire d’un génome. Population et génétique dans l’est du Québec. Sillery (Québec) : Presses de l’Université du Québec, 1991 : 145–59. [Google Scholar]
- Lavoie EM, Tremblay M, Houde L, Vezina H. Demogenetic study of three populations within a region with strong founder effects. Community Genet 2005; 8 : 152–60. [Google Scholar]
- Austerlitz F, Heyer E. Social transmission of reproductive behavior increases frequency of inherited disorders in a young-expanding population. Proc Natl Acad Sci USA 1998; 95 : 15140–4. [Google Scholar]
- Labuda M, Labuda D, Korab-Laskowska M, et al. Linkage disequilibrium analysis in young populations: pseudo-vitamin D- deficiency rickets and the founder effect in French Canadians. Am J Hum Genet 1996; 59 : 633–43. [Google Scholar]
- Labuda D, Zietkiewicz E, Labuda M. The genetic clock and the age of the founder effect in growing populations: a lesson from French Canadians and Ashkenazim. Am J Hum Genet 1997; 61 : 768–71. [Google Scholar]
- Brais B, Bouchard JP, Xie YG, et al. Short GCG expansions in the PABP2 gene cause oculopharyngeal muscular dystrophy. Nat Genet 1998; 18 : 164–7. [Google Scholar]
- Roddier K, Thomas T, Marleau G, et al. Two mutations in the HSN2 gene explain the high prevalence of HSAN2 in French Canadians. Neurology 2005; 64 : 1762–7. [Google Scholar]
- Hechtman P, Kaplan F, Bayleran J, et al. More than one mutant allele causes infantile Tay-Sachs disease in French-Canadians. Am J Hum Genet 1990; 47 : 815–22. [Google Scholar]
- Kere J. Human population genetics: lessons from Finland. Annu Rev Genomics Hum Genet 2001; 2 : 103–28. [Google Scholar]
- Jobling MA, Hurles ME, Tyler-Smith C. Human evolutionary genetics: origins, peoples and disease. Garland Science Publishing, 2003 : 458 p. [Google Scholar]
- Gerbault P. Régionalisation de l’effet fondateur au Québec. Mémoire de maîtrise, Université de Montréal, 2006. [Google Scholar]
- Yotova V, Lefebvre JF, Kohany O, et al. Complex structure of the ancestral human population or earlier Speciation of Modern Humans. Am J Hum Genet 2007 (sous presse). [Google Scholar]
- Zietkiewicz E, Yotova V, Gehl D, et al. Haplotypes in the dystrophin DNA segment point to a mosaic origin of modern humans’ diversity. Am J Hum Genet 2003; 73 : 994–1015. [Google Scholar]
- Chakraborty R. Mitochondrial DNA polymorphism reveals hidden heterogeneity within some Asian populations. Am J Hum Genet 1990; 47 : 87–94. [Google Scholar]
- Excoffier L, Laval G, Schneider S. Arlequin ver. 3.0: an integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online 2005; 1 : 47-50. http://cmpg.unibe.ch/software/arlequin3/. [Google Scholar]
- Wright S. The genetical structure of populations. Ann Eugen 1951; 15 : 323–54. [Google Scholar]
- Piercy R, Sullivan KM, Benson N, Gill P. The application of mitochondrial DNA typing to the study of white Caucasian genetic identification. Int J Legal Med 1993; 106 : 85–90. [Google Scholar]
- Richards M, Côrte-Real H, Forster P, et al. Paleolithic and neolithic lineages in the European mitochondrial gene pool. Am J Hum Genet 1996; 59 : 185–203. [Google Scholar]
- Rousselet F, Mangin P. Mitochondrial DNA polymorphisms: a study of 50 French Caucasian individuals and application to forensic casework. Int J Legal Med 1998; 111: 292–8. [Google Scholar]
- Helgason A, Hickey E, Goodacre S, et al. mtDna and the islands of the North Atlantic: estimating the proportions of Norse and Gaelic ancestry. Am J Hum Genet 2001; 68 : 723–37. [Google Scholar]
- Dubut V, Chollet L, Murail P, et al. mtDNA polymorphisms in five French groups: importance of regional sampling. Eur J Hum Genet 2004; 12 : 293–300. [Google Scholar]
- Roewer L, Croucher P J, Willuweit S, et al. Signature of recent historical events in the European Y-chromosomal STR haplotype distribution. Hum Genet 2005; 116 : 279–91. [Google Scholar]
Liste des tableaux
Statistiques descriptives de la diversité génétique pour le segment hypervariable 1, SHV1, de l’ADN mitochondrial, l’haplotype composé de sept microsatellites, STR, du chromosome Y et les deux marqueurs du chromosome X. Les échantillons québécois analysés pour SHV1 provenaient du Saguenay (n = 169), de Montréal (n = 63) et de la Gaspésie représentée par les descendants des Acadiens (n = 97), des Canadiens-français (n = 95), des Loyalistes (n = 85) et des Anglo-normands (n = 88). Les mêmes groupes étaient représentés dans l’analyse du chromosome Y (n = 95, 96, 44, 35, 42 et 54 respectivement) ainsi que des marqueurs DXS1238 (n = 83, 65, 45, 32, 40 et 54) et dys-44 (n = 69, 36, 45, 33, 40 et 54).
Liste des figures
 |
Figure 1. Représentation des principales zones d’occupation du territoire québécois (en jaune) et localisation des régions étudiées. |
| Dans le texte | |
 |
Figure 2. Histogramme des haplotypes composés des deux marqueurs du chromosome X en France (n = 257) et au Québec (n = 276), regroupés selon leur fréquence absolue dans chaque échantillon. Les couleurs pâles indiquent la portion des haplotypes exclusifs, retrouvés seulement au Québec ou seulement en France. L’haplotype le plus fréquent est le même dans les deux populations mais il apparaît un peu plus souvent au Québec (49) qu’en France (38). |
| Dans le texte | |
 |
Figure 3. Histogramme des haplotypes composés de sept marqueurs microsatellites du chromosome Y pour les régions de Montréal (n = 96) et du Saguenay (n = 95), regroupés selon leur fréquence absolue dans chaque échantillon. Le nombre des haplotypes uniques (fréquence de 1) est beaucoup plus bas chez les Saguenéens déplaçant ainsi l’histogramme vers la droite avec l’haplotype le plus fréquent représenté en 21 copies. |
| Dans le texte | |
 |
Figure 4. Mesure de distance génétique Fst par paires de populations québécoises. Mesure représentée (voir http://cmpg.unibe.ch/software/arlequin3) sur un graphique MDS (MultiDimensional Scaling ; voir Statistica – http://www.statsoft.com) pour les trois systèmes génétiques étudiés chez les Gaspésiens d’origine anglo-normande (IAN), d’origine acadienne (Ac), d’origine loyaliste (Loy) et d’origine canadienne-française (C-f) ainsi que chez les Canadiens-français de Montréal (Mtl) et du Saguenay (SLSJ). Les Saguenéens se différencient significativement (*) pour tous les systèmes, les Acadiens pour l’ADN mt et le chromosome Y et les Loyalistes pour le chromosome Y seulement. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




