| Issue |
Med Sci (Paris)
Volume 22, Number 11, Novembre 2006
|
|
|---|---|---|
| Page(s) | 995 - 998 | |
| Section | Repères | |
| DOI | https://doi.org/10.1051/medsci/20062211995 | |
| Published online | 15 November 2006 | |
Prix Nobel de Chimie 2006 Roger Kornberg : un Nobel pour la transcription des gènes
The Nobel goes to gene transcription
Membre de l’Académie des Sciences Européenne, Service de Biochimie et de Génétique Moléculaire (SBGM), CEA-Saclay, 91191, Gif-sur-Yvette Cedex, France
Roger Kornberg, 59 ans, Américain, né en 1947 à Saint-Louis (Missouri, États-Unis), est professeur de médecine à l’Université de Stanford (Californie, États-Unis). Il a 12 ans lorsqu’il accompagne son père Arthur Kornberg à Stockholm, où celui-ci se voit remettre le Prix Nobel de médecine. C’est à Stanford qu’il a fait l’essentiel de ses études et de sa carrière, si l’on excepte une parenthèse à l’université britannique de Cambridge entre 1978 et 1984. Roger Kornberg avait auparavant reçu plusieurs distinctions dont le Prix Charles Leopold Mayer en 2002, ainsi que le Prix Gairdner en 1995.
© 2006 médecine/sciences - Inserm / SRMS
Le prix Nobel de chimie vient d’être décerné à Roger Kornberg pour l’élucidation des fondements moléculaires de la transcription des gènes dans les cellules eucaryotes. La transcription de l’information génétique en ARN, codant pour des protéines ou non codants, est effectuée par des enzymes complexes, les ARN polymérases. C’est un processus vital qui sous-tend des phénomènes aussi essentiels que le développement, la différenciation cellulaire et l’adaptation des cellules à un environnement changeant. L’attribution de ce prix Nobel manifeste la conviction du Comité Nobel que ce domaine de recherche, qui a tant passionné de si nombreux laboratoires depuis presque un demi-siècle, est arrivé à maturité : les acteurs moléculaires essentiels sont identifiés et les mécanismes moléculaires établis. La contribution de Roger Kornberg dans cette immense tâche est tout à fait exceptionnelle.
Pour bien comprendre, a posteriori, les défis scientifiques, revenons brièvement sur le déroulement de ces recherches riches en découvertes et rebondissements. Comme une famille de poupées russes emboîtées les unes dans les autres, chaque découverte en appelle une autre.
Le paradigme bactérien
Tout commence en 1959 sous le signe de la chimie par une communication de Weiss et Gladstone dans le Journal of American Chemical Society. Ils y décrivent la découverte d’une activité ARN polymérase dans des noyaux de foie de rat. Le mécanisme de la réaction ressemble à celui décrit peu auparavant (1957) par Arthur Kornberg (son père, Prix Nobel de Médecine en 1959) pour la réplication de l’ADN.
Dans la décennie qui suit, l’essentiel des mécanismes fondamentaux est établi, pour des raisons pratiques, en utilisant l’ARN polymérase bactérienne, purifiée dès 1960 : l’ARN est synthétisé dans la direction 5’ vers 3’ à partir de ribonucléosides triphosphates, et la séquence de l’ARN est complémentaire au brin d’ADN servant de matrice. La question centrale de la sélection des sites de démarrage et de terminaison de la transcription est résolue pour l’essentiel avec la découverte du facteur d’initiation sigma et du facteur de terminaison rho (1969). La même année est élucidée la structure multimérique de l’ARN polymérase procaryotique (β’βα2). La première séquence de promoteur est publiée en 1974 (un exploit technique à cette époque pré-génomique). À ce moment, l’impression générale était que les mécanismes gouvernant la sélectivité et la fidélité de la transcription des gènes procaryotes étaient compris, tout comme les principes de la régulation de l’activité des gènes couronnés par le prix Nobel décerné à François Jacob et à Jacques Monod en 1965.
Des formes multiples d’ARN polymérases chez les eucaryotes
Pour aussi naïve qu’elle paraisse aujourd’hui, la question de la présence ou non de facteurs de type sigma dans les cellules eucaryotes était dans tous les esprits. Il en était de même pour le concept de séquences promotrices. Les mécanismes assurant la sélectivité de l’initiation de la transcription chez les procaryotes étaient-ils conservés dans les cellules nucléées ? C’est dans ce contexte que Robert Roeder et, indépendamment, Pierre Chambon découvrent que les eucaryotes ont plusieurs formes d’ARN polymérases nucléaires (1970). Cette découverte a un profond impact. Dans la décennie qui suit, nombre de laboratoires se lancent dans la purification de ces enzymes pour établir, sur des bases structurales et fonctionnelles, l’existence de trois formes distinctes d’ARN polymérases (I, II et III) transcrivant des classes de gènes différentes. Les enzymes de la levure Saccharomyces cerevisiae sont caractérisées en détail, en particulier dans mon laboratoire, à Saclay. Leur complexité moléculaire (12, 14 et 17 sous-unités) surprend. La présence de cinq sous-unités conservées dans les trois enzymes suggère une parenté évolutive confirmée par l’analyse immunologique. Ces trois formes d’enzymes sont apparentées à l’ARN polymérase des archaebactéries. En dépit de leur complexité, les ARN polymérases eucaryotes se révèlent incapables d’initier correctement la transcription. Ce constat décevant ouvre une nouvelle vague de dix années de recherches, en quête des maillons manquants.
Découverte des facteurs généraux de transcription
Un retour aux extraits cellulaires s’imposait. En 1979, Roeder obtient la transcription correcte d’un gène viral en utilisant un extrait protéique comme source de protéines auxilliaires. La purification et la caractérisation des facteurs généraux de transcription est lancée. De nombreux et grands laboratoires s’y consacrent : Roeder, Chambon, Egly, Sharp, les Conaway, Greenblatt, Tjian, Reinberg et d’autres encore. Les travaux révèlent l’énorme complexité des facteurs généraux requis par l’ARN polymérase II pour initier correctement la synthèse des ARNm. Au total six facteurs sont requis dont deux, TFIID (14 sous-unités) et TFIIH (9 sous-unités) sont aussi grands que l’ARN polymérase II (12 sous-unités). Le cas du facteur de transcription TFIIH mérite une mention spéciale car ce facteur intervient à la fois dans la transcription et dans la réparation de l’ADN (travaux de Jean-Marc Egly et de Roger Kornberg). Des mutations dans le TFIIH humain causent des syndromes graves.
Des facteurs différents dirigent la transcription par les ARN polymérases I et III. Mon laboratoire purifie et clone les facteurs de levure qui dirigent la transcription des gènes d’ARNt, et décrit la cascade d’interactions protéines-ADN et protéine-protéine qui conduit au recrutement de l’ARN polymérase III, à la transcription précise du gène et au recyclage de l’enzyme. Au total, 26 protéines sont impliquées.
Découverte des éléments promoteurs et des enhancers
Dans le même temps, des travaux menés pour beaucoup dans le laboratoire de Pierre Chambon révèlent l’existence et la nature des promoteurs eucaryotes. En 1979, on s’interrogeait encore sur l’existence de séquences promotrices dispersées dans l’immensité des génomes animaux. Les premiers éléments promoteurs décrits concernent les gènes d’ARNt transcrits par l’ARN polymérase III. De façon surprenante, ils sont intragéniques ! Peu après, les nouvelles méthodes de séquençage de l’ADN révèlent une courte séquence de type TATA en amont des premiers gènes d’ARN messager séquencés. Chambon montre, par mutagenèse dirigée, que cette « boîte TATA » joue un rôle déterminant in vitro. Mais ce n’est pas tout : des séquences bien plus en amont, parfois très éloignées, augmentent considérablement l’efficacité de la transcription. Ce sont les séquences enhancer, découvertes par Schaffner et par Chambon, indépendamment (1981). Les enhancers fonctionnent dans les deux orientations (contrairement à la boîte TATA) et à grande distance, jusqu’à plusieurs milliers de paires de bases. Cette observation, sans précédent chez les procaryotes, déclenche la recherche de séquences fonctionnellement homologues dans divers organismes. L’analyse détaillée d’un grand nombre de promoteurs de cellules animales ou de levures révèle d’autres éléments promoteurs proches du site d’initiation. Toutefois, la boîte TATA reste conceptuellement l’élément central qui lie TBP (TATA-binding protein), une protéine extrêmement conservée des archaebactéries à l’homme. En 1989 (trente ans après Weiss et Gladstone), TBP de levure est purifié et cloné, étape décisive qui entraîne la découverte par Tjian des TAF (TBP-associated factors) qui forment avec TBP un énorme complexe protéique qui correspond, en fait, à TFIID.
Activateurs et co-activateurs : le concept de « Médiateur »
La découverte des éléments enhancers a ouvert un champ immense d’investigation à la recherche des « facteurs spécifiques de transcription » se liant à l’ADN. La récolte est abondante. Tjian purifie Sp1, le premier d’une longue liste d’activateurs, et premier membre d’une nombreuse famille de protéines à « doigt à zinc ». Une autre célèbre famille de facteurs contient un motif de liaison dit leucine zipper (Steve McKnight) qui peut former un hétérodimère avec une autre protéine de même type. L’implication évidente de l’hétérodimérisation était la création d’une diversité de liaison de type combinatoire. Chambon décortique la structure modulaire, multi-sites, de l’enhancer du virus SV40 et souligne que cela accroît considérablement les possibilités combinatoires d’activation ou de répression des gènes. Le cas de la superfamille des récepteurs hormonaux nucléaires est un extraordinaire exemple de complexité combinatoire (Chambon et Evans). On commence à comprendre comment un nombre nécessairement limité de facteurs se liant à l’ADN peut réguler un si grand nombre de gènes dans différentes conditions, spatiales, temporelles ou environnementales.
Le paradigme bactérien suggérait que les activateurs (ou répresseurs) contactent directement la machinerie de transcription. Plusieurs expériences in vitro allaient d’ailleurs dans ce sens. Cependant, des études indépendantes de Tjian, Kornberg, Guarente et Roeder concluent à l’existence d’une entité, appelée médiateur, co-activateur ou adaptateur, nécessaire pour l’activation des gènes, entité qui ferait le pont entre les activateurs (ou répresseurs) liés à l’ADN et la machinerie de transcription. Tjian propose, en s’appuyant sur des expériences spectaculaires, que les TAF jouent le rôle de co-activateurs. Cette hypothèse est contestée car l’activation de gènes de levure peut intervenir en absence de TAF. En fait, c’est le grand mérite de Kornberg de réussir à purifier l’activité « Médiateur » qu’il avait découverte dans la levure.
« Médiateur » est un complexe géant regroupant 20 protéines dont les deux tiers étaient déjà connus des généticiens de la levure comme étant des activateurs ou des répresseurs. Cette magnifique intégration d’études génétiques et biochimiques établit le rôle central de Médiateur dans la régulation des gènes de levure. Kornberg et Young montrent que Médiateur se lie à l’ARN polymérase II pour former ce qu’ils ont appelé l’holoenzyme. Médiateur étant conservé de la levure à l’homme, les fondements des mécanismes de la régulation des gènes eucaryotes étaient établis. Pour arriver à ce résultat spectaculaire, notons que Kornberg a dû mettre au point un système de transcription in vitro reconstitué avec tous les facteurs généraux de transcription de levure purifiés à homogénéité.
Structure de l’ARN polymérase II en action
Comme on l’a vu, la transcription des gènes fait intervenir des complexes protéiques géants dont la taille décourageait a priori toute analyse structurale de la machinerie de transcription. Toutefois, un premier pas important est réalisé par la cristallisation des complexes TBP-ADN et TFIIB-TBP-ADN. TBP, avec sa forme en selle de cheval, courbe l’ADN au niveau de la boîte TATA. La liaison de TFIIB bloque cette structure en exposant des surfaces d’interaction pour d’autres composants (Burley et Sigler).
L’avancée la plus extraordinaire dans ce domaine, que je ne pensais pas connaître de mon vivant scientifique, nous la devons à Kornberg. En 2001, dans deux papiers publiés dans Science, et d’autres publiés depuis, il décrit la structure de l’ARN polymérase II de levure à 2,8 Å de résolution, ainsi que celle du complexe d’élongation constitué de l’enzyme, de l’ADN et de l’ARN produit. La structure de l’enzyme cristallisée dans l’acte de transcription montre clairement le double brin d’ADN engagé dans une profonde crevasse formée à l’interface des deux grandes sous-unités de l’enzyme ; on distingue les 3 paires de bases d’ADN ouvertes et exposées au site actif, les 9 paires de bases d’hybride ADN.ARN formant la bulle de transcription et l’ion Mg2+ au site actif ; une ouverture, en forme d’entonnoir, débouche par un pore au site actif et sert probablement à collecter les nucléosides triphosphates (Figure 1). De multiples contacts protéines-acides nucléiques rendent compte de la séparation locale des brins d’ADN et de l’ARN synthétisé, ainsi que de l’étape de translocation après chaque ajout d’un nucléotide à la chaîne d’ARN. Kornberg nous montre comment marche la transcription au niveau atomique !
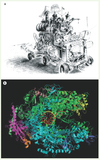 |
Figure 1. A. ARN polymérase II avant Kornberg. B. Structure de l’ARN polymérase II en action. |
Ces résultats spectaculaires couronnent quinze années de travail. Ils permettent d’interpréter les résultats biochimiques et génétiques accumulés pendant 20 ans, et ouvrent la voie à l’analyse du complexe d’initiation contenant facteurs généraux de transcription et médiateur.
L’attribution du prix Nobel à Roger Kornberg souligne la portée exceptionnelle de ses contributions à l’un des plus fascinants domaines de la biologie moléculaire des cellules eucaryotes. De grands laboratoires, d’immenses chercheurs ont contribué à des avancées essentielles au cours de ces quarante dernières années. J’ai voulu les évoquer à cette occasion pour qu’en admirant la dernière poupée russe assemblée, on garde à l’esprit une vision d’ensemble.
Liste des figures
 |
Figure 1. A. ARN polymérase II avant Kornberg. B. Structure de l’ARN polymérase II en action. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




