| Issue |
Med Sci (Paris)
Volume 20, Number 6-7, Juin-Juillet 2004
|
|
|---|---|---|
| Page(s) | 617 - 619 | |
| Section | Le Magazine : Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2004206-7617 | |
| Published online | 15 June 2004 | |
Eya, un activateur transcriptionnel à activité tyrosine-phosphatase
The transcriptional activator Eya is a tyrosine phosphatase
Département Génétique, Développement et Pathologie Moléculaire, Institut Cochin, Inserm U.567, Cnrs 8104, Université Paris V, 24, rue du Faubourg Saint-Jacques, 75014 Paris, France
Le gène Eya1 est connu des cliniciens pour son implication dans le syndrome BOR1 (branchio-oto-rénal) caractérisé par des atteintes rénales, de l’oreille interne et des dérivés des arcs branchiaux [1]. Des mutations de Eya1 (eya du nom du gène eyes absent, voir ci-dessous) ont également été décrites chez les patients BO [2], et le gène Eya4, situé au locus DFNA10, est associé à une surdité chez l’homme [3]. L’invalidation fonctionnelle du gène Eya1 chez la souris reproduit l’ensemble des phénotypes des patients BOR et l’étude de ce modèle animal suggère que Eya1 participe au contrôle de la prolifération cellulaire (directement ou en protégeant les cellules de l’apoptose) [4, 5]. Jusqu’à présent, on ne connaissait de la fonction moléculaire des protéines Eya que leur rôle de co-activateurs transcriptionnels associés aux protéines Six. Des résultats récents montrent que les protéines Eya possèdent également une fonction enzymatique tyrosine-phosphatase dont l’importance physiologique a été démontrée chez la drosophile. Cette découverte ouvre de nouvelles perspectives quant à l’interprétation du rôle de Eya au cours du développement embryonnaire normal, mais aussi dans l’étiologie du syndrome BOR. Peut-être qu’une fois encore, la recherche chez la mouche permettra la mise au point de nouvelles stratégies thérapeutiques pour l’homme.
Ce que l’on savait : le rôle de Eya en tant que co-activateur transcriptionnel associé aux protéines Six
Initialement le gène eyes absent (eya) a été caractérisé chez la drosophile, où il joue un rôle crucial dans le développement de l’œil. Eya agit au sein d’un réseau de gènes liés par des boucles de régulation transcriptionnelle positives et réciproques, qui inclut eyeless (ey, homologue de Pax6), sine oculis (so/Six) et dachshund (dach). Des mutations de chacun de ces gènes entraînent une absence, ou un développement anormal, de l’œil composé chez l’insecte. Ces gènes codent pour des facteurs de transcription (Ey et So lient directement l’ADN sur des sites spécifiques) ou des cofacteurs (Eya et Dach) qui agissent en synergie, via des interactions protéiques directes. En particulier, des interactions Eya-So et Eya-Dach ont été caractérisées. Ces gènes ont été qualifiés de master genes car lorsqu’ils sont exprimés dans des territoires ectopiques, tels que le disque imaginal de la patte ou de l’antenne dans l’embryon de drosophile, ils commandent la formation d’un œil additionnel [6].
Par homologie de séquence, des homologues de eya ont été identifiés chez les vertébrés (quatre gènes chez la souris et l’homme). Les domaines protéiques impliqués dans les interactions avec Six et Dach ont été particulièrement bien conservés. D’ailleurs, des synergies fonctionnelles ont été mises en évidence entre les protéines de souris [7, 8], indiquant que les mécanismes moléculaires décrits chez la drosophile pourraient être actifs chez les vertébrés. Néanmoins, le rôle activateur des cofacteurs Dach semble remis en cause (un effet répresseur de Dach1 a été observé [9, 10], alors que Dach2, stabilisé sur le complexe Six-Eya par la protéine CBP, joue un rôle activateur (Figure 1C) [11]). Par ailleurs, des observations surprenantes ont été faites. Par exemple, l’absence de localisation nucléaire des protéines Eya en l’absence de leur partenaire Six dans les cellules COS [8], ou encore l’interaction de Eya avec les petites protéines G monomériques activées (Galphai et GalphaZ) qui la retient à la membrane plasmique (Figure 1C) [12]. Ces observations posent la question d’un éventuel rôle de Eya dans la transduction d’un signal extracellulaire.
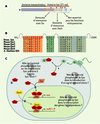 |
Figure 1. Structure et fonction de la protéine Eya. A. Représentation schématique des différents domaines fonctionnels des protéines Eya. Le domaine aminoterminal est responsable de l’activité transcriptionnelle, alors que le domaine carboxyterminal participe aux interactions avec les partenaires protéiques (Six, Dach, protéines G). Les 271 aa carboxyterminaux sont particulièrement bien conservés au sein de la famille Eya et définissent un domaine protéique particulier nommé domaine Eya. C’est dans cette région qu’a été identifié le site catalytique tyrosine phosphatase (rectangles orange, vert et bleu). Pour indication, quelques sites de mutations ponctuelles identifiées chez les patients BOR ont été reportés sur la figure (balises noires). B. Séquence des trois motifs caractérisant les enzymes HAD (haloacid dehalogenase hydrolases). Les aa colorés sont identiques dans toutes les protéines Eya et constituent la signature biochimique des enzymes HAD. C. Modèle résumant les interactions protéiques connues entre Eya et ses partenaires. 1. Eya, seul, reste localisé dans le cytoplasme ; 2. En présence d’homéoprotéine Six, Eya est transloquée dans le noyau, où le complexe Six-Eya fonctionne en synergie pour activer la transcription de gènes cibles : la protéine Six permet l’ancrage du complexe à l’ADN au niveau de séquences spécifiques et Eya active fortement la transcription des gènes cibles. 3. Eya peut également interagir avec Dach, mais le rôle activateur et/ou répresseur de ce troisième partenaire transcriptionnel est encore controversé. Le complexe Six-Eya-Dach est stabilisé par la protéine CBP. 4. Eya est capable d’interagir avec les petites protéines Galphai et GalphaZ monomériques activées ; cette interaction retient Eya à la membrane plasmique et suggère que Eya participe à la transduction d’un signal extracellulaire, encore non identifié. La découverte de l’activité tyrosine phosphatase de Eya amène à reconsidérer ces interactions protéiques sous un nouvel angle, puisque chaque partenaire pourrait également être un substrat de Eya. Enfin, il est très tentant, mais également très spéculatif à ce jour, d’imaginer un modèle où Eya jouerait un rôle d’interrupteur, permettant d’allumer ou d’éteindre l’expression de gènes en réponse à un signal extra-cellulaire : l’activation de la transcription ne serait possible que dans les cellules exprimant les protéines Six qui permettent la translocation nucléaire de Eya et son ancrage à l’ADN ; en revanche, la présence d’un signal extra-cellulaire faisant intervenir l’activation des protéines Galphai et GalphaZ monomériques inhiberait la transcription des gènes cibles en maintenant Eya au niveau de la membrane plasmique. |
La grande nouveauté : Eya possède une activité tyrosine-phosphatase
Trois articles parus dans Nature au mois de décembre 2003 décrivent l’activité phosphatase des protéines Eya [10, 13, 14]. Non seulement, la région carboxyterminale de ces protéines est très conservée, mais en plus elle présente trois motifs caractéristiques des enzymes de la classe des haloacid dehalogenase hydrolases (HAD) (Figure 1B). L’analyse biochimique a révélé que ce domaine protéique possède une activité tyrosine-phosphatase dépendante du magnésium. Les différents membres de la famille Eya présentent des activités enzymatiques différentes et variables selon les substrats : l’isoforme dont le gène a été cloné chez la plante Arabidopsis thaliana est la plus efficace in vitro sur le substrat synthétique pNPP (p-nitrophényl phosphate) ; viennent ensuite les isoformes de souris et la protéine de drosophile. Ces résultats non seulement constituent la première démonstration d’une activité phosphatase au sein de la famille HAD, mais surtout, Eya est à ce jour le seul activateur transcriptionnel connu qui possède une activité phosphatase intrinsèque. En effet, il existe de nombreux exemples de facteurs de transcription dont l’activité est modulée par des modifications post-traductionnelles de type phosphorylation/déphosphorylation. Cependant, jusqu’à présent, les phosphatases impliquées n’ont pas été décrites comme des acteurs transcriptionnels directs.
Afin de valider la signification physiologique de cette activité enzymatique, des tests fonctionnels ont été réalisés in vivo chez la drosophile, avec des formes de eya mutées dont l’activité phosphatase est perturbée in vitro. Ces travaux démontrent l’importance de l’activité phosphatase pour la fonction de Eya au cours du développement de l’œil d’insecte. Néanmoins, un petit nombre d’ommatidies2 se forme correctement dans les deux cas, suggérant que l’activité phosphatase de Eya, bien que nécessaire, n’est pas indispensable pour induire la différenciation oculaire. À ce stade, plusieurs hypothèses peuvent être formulées : soit les mutations étudiées affectent l’activité phosphatase in vitro, mais pas, ou peu, in vivo ; soit la protéine Eya possède deux fonctions distinctes, l’une liée à l’activité phosphatase, l’autre à l’activation transcriptionnelle, et toutes deux sont requises au cours de l’organogenèse oculaire.
Bien que remarquablement concordantes, les informations révélées par ces trois articles présentent quelques divergences, notamment en ce qui concerne l’interaction Eya-So [14], et à ce jour la question reste entière de savoir si les protéines Six sont des substrats de Eya. D’une manière générale, il sera essentiel d’identifier les protéines déphosphorylées par Eya afin connaître le rôle joué par cette activité phosphatase dans la cellule.
Les questions
Cette nouvelle découverte soulève de nombreuses questions (Figure 1C). Tout d’abord, l’activité phosphatase de Eya est-elle constitutive dans la cellule ou se manifeste-t-elle uniquement dans un compartiment sub-cellulaire particulier (noyau ou cytoplasme) ? Cette activité enzymatique a-t-elle un lien avec l’interaction de Eya, d’une part avec les petites protéines G au niveau de la membrane plasmique, d’autre part avec les facteurs de transcription Six et Dach dans le noyau ? En d’autres termes, ces partenaires protéiques sont-ils des substrats de Eya, et leur état phosphorylé versus déphosphorylé conditionne-t-il leur affinité pour Eya ? Surtout, l’activité phosphatase de Eya influence-t-elle directement ses propres caractéristiques transcriptionnelles. Une activité auto-catalytique de Eya a été suggérée par T.L. Tootle et al. du fait de la capacité de Eya à former des homodimères [15]. Si c’est le cas, l’identification des sites phosphorylés de Eya et des kinases responsables de cette phosphorylation, devrait permettre de rattacher Eya à une voie de signalisation et ainsi de mieux comprendre le rôle intégrateur de Eya au cours du développement embryonnaire.
Enfin, chez l’homme, la plupart des mutations de Eya1 identifiées chez les patients BOR sont des mutations ponctuelles qui touchent des acides aminés de la région carboxyterminale (Figure 1A) [16], mais aucune ne touche les acides aminés du site catalytique de Eya. Des études complémentaires seront nécessaires pour établir si ces mutations affectent, ou non, l’activité phosphatase, et donc si le dérèglement de cette fonction enzymatique peut être responsable de certains aspects de la pathologie. Les protéines Eya, et en particulier Eya1, participent à de nombreux processus de différenciation au cours du développement embryonnaire [4, 5]. Ces fonctions ont jusqu’à présent été corrélées à l’effet transcriptionnel des co-activateurs Eya, cependant l’activité phosphatase de ces protéines ouvre de nouvelles perspectives à explorer.
Remerciements
Je tiens à remercier Pascal Maire pour sa lecture critique du manuscrit.
Le syndrome BOR associe des anomalies des arcs branchiaux (fentes, fistules ou kystes branchiaux), des anomalies auditives (malformation du pavillon de l’oreille avec orifices pré-auriculaires, surdité de transmission ou surdité neurosensorielle) et des anomalies rénales (malformation de l’arbre urinaire, hypoplasie ou agénésie rénale, dysplasie rénale). L’atteinte rénale peut être responsable d’une insuffisance rénale chronique. L’expression de la maladie varie d’une famille à l’autre, certaines ne présentant pas d’anomalies rénales ou d’anomalies de l’arbre urinaire. Le syndrome BOR est transmis selon le mode autosomique dominant. Sa prévalence est de 1/40 000. [source Pr P. Niaudet orphanet].
Les ommatidies sont les unités structurales composant les yeux à facettes des insectes : une ommatidie est formée d’une cornée transparente (lentille biconvexe), d’un cône cristallin entouré de cellules pigmentaires et d’un prolongement nerveux, le rhabdome, s’unissant à ceux des autres ommatidies en une rétinula qui entre en connexion avec les lobes optiques du cerveau.
Références
- Abdelhak, S., Kalatzis V, Heilig R, et al. A human homologue of the Drosophila eyes absent gene underlies branchio-oto-renal (BOR) syndrome and identifies a novel gene family. Nat Genet 1997; 15 : 157–64. [Google Scholar]
- Vincent C, Kalatzis V, Abdelhak S, et al. BOR and BO syndromes are allelic defects of EYA1. Eur J Hum Genet 1997; 5 : 242–6. [Google Scholar]
- Wayne S, Robertson NG, DeClau F, et al. Mutations in the transcriptional activator EYA4 cause late-onset deafness at the DFNA10 locus. Hum Mol Genet 2001; 10 : 195–200. [Google Scholar]
- Xu PX, Adams J, Peters H, et al. Eya1-deficient mice lack ears and kidneys and show abnormal apoptosis of organ primordia. Nat Genet 1999; 23 : 113–7. [Google Scholar]
- Xu PX, Zheng W, Laclef C, et al. Eya1 is required for the morphogenesis of mammalian thymus, parathyroid and thyroid. Development 2002; 129 : 3033–44. [Google Scholar]
- Wawersik S, Maas RL. Vertebrate eye development as modeled in Drosophila. Hum Mol Genet 2000; 9 : 917–25. [Google Scholar]
- Heanue TA, Reshef R, Davis RJ, et al. Synergistic regulation of vertebrate muscle development by Dach2, Eya2, and Six1, homologs of genes required for Drosophila eye formation. Genes Dev 1999;13 : 3231–43. [Google Scholar]
- Ohto H, Kamada S, Tago K, et al. Cooperation of six and eya in activation of their target genes through nuclear translocation of Eya. Mol Cell Biol 1999; 19 : 6815–24. [Google Scholar]
- Li X, Perissi V, Liu F, et al. Tissue-specific regulation of retinal and pituitary precursor cell proliferation. Science 2002; 297 : 1180–3. [Google Scholar]
- Li X, Oghi KA, Zhang J, et al. Eya protein phosphatase activity regulates Six1-Dach-Eya transcriptional effects in mammalian organogenesis. Nature 2003; 426 : 247–54. [Google Scholar]
- Ikeda K, Watanabe Y, Ohto H, Kawakami K. Molecular interaction and synergistic activation of a promoter by Six, Eya, and Dach proteins mediated through CREB binding protein. Mol Cell Biol 2002; 22 : 6759–66. [Google Scholar]
- Fan X, Brass LF, Poncz M, et al. The alpha subunits of Gz and Gi interact with the eyes absent transcription cofactor Eya2, preventing its interaction with the six class of homeodomain-containing proteins. J Biol Chem 2000; 275 : 32129–34. [Google Scholar]
- Rayapureddi JP, Kattamuri C, Steinmetz BD, et al. Eyes absent represents a class of protein tyrosine phosphatases. Nature 2003; 426 : 295–8. [Google Scholar]
- Tootle TL, Silver SJ, Davies EL, et al. The transcription factor Eyes absent is a protein tyrosine phosphatase. Nature 2003; 426 : 299–302. [Google Scholar]
- Silver SJ, Davies EL, Doyon L, Rebay I. Functional dissection of eyes absent reveals new modes of regulation within the retinal determination gene network. Mol Cell Biol 2003; 23 : 5989–99. [Google Scholar]
- Ozaki H, Watanabe Y, Ikeda K, Kawakami K. Impaired interactions between mouse Eyal harboring mutations found in patients with branchio-oto-renal syndrome and Six, Dach, and G proteins. J Hum Genet 2002; 47 : 107–16. [Google Scholar]
© 2004 médecine/sciences - Inserm / SRMS
Liste des figures
 |
Figure 1. Structure et fonction de la protéine Eya. A. Représentation schématique des différents domaines fonctionnels des protéines Eya. Le domaine aminoterminal est responsable de l’activité transcriptionnelle, alors que le domaine carboxyterminal participe aux interactions avec les partenaires protéiques (Six, Dach, protéines G). Les 271 aa carboxyterminaux sont particulièrement bien conservés au sein de la famille Eya et définissent un domaine protéique particulier nommé domaine Eya. C’est dans cette région qu’a été identifié le site catalytique tyrosine phosphatase (rectangles orange, vert et bleu). Pour indication, quelques sites de mutations ponctuelles identifiées chez les patients BOR ont été reportés sur la figure (balises noires). B. Séquence des trois motifs caractérisant les enzymes HAD (haloacid dehalogenase hydrolases). Les aa colorés sont identiques dans toutes les protéines Eya et constituent la signature biochimique des enzymes HAD. C. Modèle résumant les interactions protéiques connues entre Eya et ses partenaires. 1. Eya, seul, reste localisé dans le cytoplasme ; 2. En présence d’homéoprotéine Six, Eya est transloquée dans le noyau, où le complexe Six-Eya fonctionne en synergie pour activer la transcription de gènes cibles : la protéine Six permet l’ancrage du complexe à l’ADN au niveau de séquences spécifiques et Eya active fortement la transcription des gènes cibles. 3. Eya peut également interagir avec Dach, mais le rôle activateur et/ou répresseur de ce troisième partenaire transcriptionnel est encore controversé. Le complexe Six-Eya-Dach est stabilisé par la protéine CBP. 4. Eya est capable d’interagir avec les petites protéines Galphai et GalphaZ monomériques activées ; cette interaction retient Eya à la membrane plasmique et suggère que Eya participe à la transduction d’un signal extracellulaire, encore non identifié. La découverte de l’activité tyrosine phosphatase de Eya amène à reconsidérer ces interactions protéiques sous un nouvel angle, puisque chaque partenaire pourrait également être un substrat de Eya. Enfin, il est très tentant, mais également très spéculatif à ce jour, d’imaginer un modèle où Eya jouerait un rôle d’interrupteur, permettant d’allumer ou d’éteindre l’expression de gènes en réponse à un signal extra-cellulaire : l’activation de la transcription ne serait possible que dans les cellules exprimant les protéines Six qui permettent la translocation nucléaire de Eya et son ancrage à l’ADN ; en revanche, la présence d’un signal extra-cellulaire faisant intervenir l’activation des protéines Galphai et GalphaZ monomériques inhiberait la transcription des gènes cibles en maintenant Eya au niveau de la membrane plasmique. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




