Figure 3.
Télécharger l'image originale
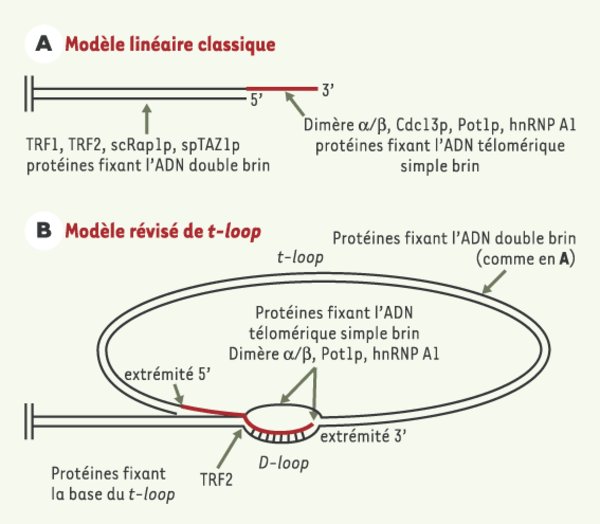
Modèles de structure des télomères. A. Modèle classique d’une extrémité chromosomique avec une courte extension en 3’ (en rouge) et un complexe protéique protégeant l’ADN chromosomique (modèle linéaire). B. Les télomères des mammifères se terminent par de larges boucles, les t-loops (telomere loops). Le double brin télomérique se replie sur lui-même, l’extension 3’ simple brin s’insère dans la portion double brin et engendre la D-loop (displacement loop). La protéine TRF2 se fixe à la base du D-loop in vitro. Les t-loops, en association avec un complexe protéique terminal, pourraient permettre de différencier les extrémités des chromosomes des cassures double brin dans l’ADN, et éviter ainsi la détection des télomères par la machinerie de réparation des cassures d’ADN. Les régions simple brin du t-loop et les extensions en 3’ sont indiquées en rouge. TRF1, 2 : telomeric repeat binding factor 1, 2 ; scRap1p : protéine télomérique RAP1 de Saccharomyces cerevisiae ; Cdc13p : telomere binding protein Cdc13p ; Pot1p : protection of telomeres 1 ; hnRNPA1 : heterogeneous ribonucleoprotein A1.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




