| Issue |
Med Sci (Paris)
Volume 39, Number 11, Novembre 2023
|
|
|---|---|---|
| Page(s) | 862 - 868 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2023163 | |
| Published online | 29 November 2023 | |
Immunité bactérienne : à la découverte d’un nouveau monde
Bacterial immunity: Uncovering a new world
Institut Pasteur, université Paris Cité, Inserm U1284, Diversité moléculaire des microbes ( Molecular Diversity of Microbes lab ), 75015 Paris, France
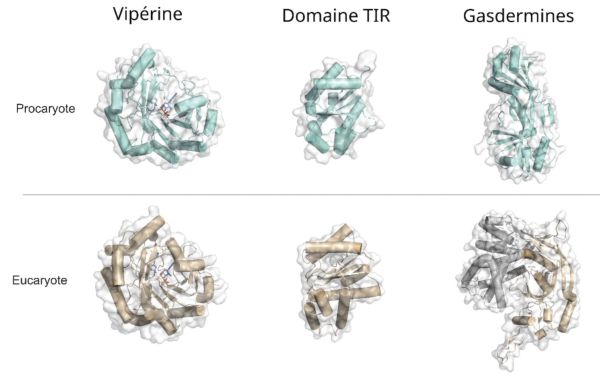
Les virus sont des parasites qui infectent tous les organismes vivants, et les bactéries n’y font pas exception. Pour se défendre contre leurs virus (les bactériophages ou phages), les bactéries se sont dotées d’un éventail de mécanismes élaborés, dont la découverte et la compréhension sont en pleine expansion. Dans les années 2000, seuls quelques systèmes de défense étaient connus et deux semblaient présents chez la plupart des bactéries. En 2018, une nouvelle méthode fondée sur l’analyse des génomes a révélé l’existence potentielle de nombreux autres. Plus de 150 nouveaux systèmes anti-phages ont été découverts au cours des cinq dernières années. On estime maintenant qu’il en existe probablement des milliers. Cette formidable diversité, qui est à mettre en parallèle avec la considérable diversité virale, s’exprime tant en termes de combinaisons de systèmes possibles dans les génomes bactériens que de mécanismes moléculaires. Une des observations les plus surprenantes qui émerge est la découverte de similarités entre certains systèmes de défense bactériens et des mécanismes antiviraux eucaryotes. Contrairement au paradigme jusqu’alors en place, des organismes aussi différents que des champignons, des plantes, des bactéries ou des êtres humains partagent certaines stratégies moléculaires pour combattre des infections virales, suggérant qu’une part sous-estimée de l’immunité antivirale eucaryote a directement évolué à partir des systèmes de défense bactériens.
Abstract
Viruses are parasites that infect all living organisms, and bacteria are no exception. To defend themselves against their viruses (phages), bacteria have developed numerous and sophisticated defense mechanisms, our understanding of which is rapidly growing. In the 2000s, only a handful of mechanisms were known and only two of them seemed to be found in most bacteria. In 2018, a new key method based on genome analysis revealed that there were likely many others. Indeed, over the past five years, more than 150 new mechanisms have been discovered. It is now estimated that there are probably thousands. This remarkable diversity, paralleled with the tremendous viral diversity, is evident both in terms of possible combinations of systems in bacterial genomes and in molecular mechanisms. One of the most surprising observations emerging from the exploration of this diversity is the discovery of striking similarities between certain bacterial defense systems and antiviral systems in humans, as well as plant (and eukaryotes in general) immune systems. Contrary to the previously accepted paradigm, organisms as diverse as fungi, plants, bacteria and humans share certain molecular strategies to fight viral infections, suggesting that an underestimated part of eukaryotic antiviral immunity could have evolved from bacterial antiviral defense systems.
© 2023 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.