| Issue |
Med Sci (Paris)
Volume 36, Number 11, Novembre 2020
|
|
|---|---|---|
| Page(s) | 980 - 983 | |
| Section | Le Magazine | |
| DOI | https://doi.org/10.1051/medsci/2020178 | |
| Published online | 05 November 2020 | |
La protéine neuronale Arc : une capside de rétrotransposon recyclée pour des fonctions clés dans les synapses
The neuronal protein Arc: a retrotransposon capsid domesticated to serve key synaptic functions
1
Inserm U1016, CNRS UMR8104, Institut Cochin, Groupe hospitalier Cochin-Port-Royal, bâtiment Cassini, 123 boulevard de Port-Royal, 75014 Paris, France.
2
UFR927 Sciences de la vie, Sorbonne Université, 4 Place Jussieu, 75005 Paris, France.
Les génomes des vertébrés contiennent un grand nombre de rétrovirus endogènes, vestiges d’infections de cellules germinales d’un ancêtre de l’hôte par des rétrovirus. Au cours de l’évolution, les rétrovirus endogènes ont très souvent (mais pas toujours) perdu leur capacité infectieuse et se transmettent donc surtout verticalement [1] (→).
(→) Voir la Synthèse de F.J. Kim et al., m/s n° 10, octobre 2004, page 876
Les génomes eucaryotes présentent aussi de nombreuses séquences dérivées de rétrotransposons à LTR (long terminal repeat), éléments mobiles étroitement apparentés aux rétrovirus. Les rétrotransposons à LTR possèdent des gènes gag et pol1 comme les rétrovirus, mais sont généralement dépourvus de gène d’enveloppe env [1, 2]. Leur cycle est donc principalement intracellulaire. Néanmoins, la limite est parfois floue puisque certains rétrovirus endogènes ont perdu leur gène env, tandis que certains rétrotransposons à LTR en ont capturé un [1]. Pour ces deux rétro-éléments, nous utiliserons ici l’adjectif « rétroviral ». Comme les rétrovirus, les rétrotransposons à LTR forment des capsides permettant la protection et le trafic de leur génome pendant leur réplication. Parmi les rétrotransposons à LTR, ceux de la famille Ty3/gypsy (metaviridae2) sont les plus proches des rétrovirus, et probablement leurs ancêtres [2].
Le gène Arc3 est rapidement induit par l’activité neuronale, ce qui a permis son identification et lui a valu ses deux noms (activity-regulated cytoskeleton-associated protein ou Arg3.1, activation-regulated gene). Chez les rongeurs, Arc joue un rôle clé dans différentes formes de plasticité synaptique4 [3-6]. Arc est ainsi indispensable pour le remodelage du cortex visuel consécutif à une occlusion mono-oculaire, ou dans la consolidation de la mémoire. Dans des situations d’apprentissage, l’induction de Arc permet d’identifier les neurones de l’hippocampe et du néocortex impliqués dans le traitement de l’information et la mémorisation [3, 7]. L’ARN messager (ARNm) Arc est transporté dans les dendrites, et la protéine s’accumule dans les synapses activées. Arc est nécessaire pour l’endocytose des récepteurs du glutamate de type AMPAR5 ou la polymérisation de l’actine, deux processus impliqués dans différentes formes de plasticité synaptique [3-7]. Néanmoins, comme la séquence d’acides aminés de Arc ne présente pas de similitude claire avec celle d’autres protéines cellulaires, l’origine de Arc et les bases moléculaires de ses activités sont longtemps demeurées énigmatiques.
Les réponses à ces questions ont été apportées par des études montrant que des homologues de Arc existent bel et bien… parmi les protéines rétrovirales. Arc présente en effet, dans sa moitié C-terminale, une structure bilobée similaire à celle de la portion CA (capside) des protéines Gag rétrovirales, les plus proches étant celles des rétrotransposons Ty3/gypsy (Figure 1) [2, 4-8]. Cette ressemblance révèle l’origine de Arc et suggère son mode de fonctionnement moléculaire. La portion CA fournit, après clivage du précurseur polypeptidique Gag, les protéines de la capside rétrovirale. De façon similaire, les protéines Arc de rat recombinantes purifiées ou surexprimées dans des cellules eucaryotes s’assemblent in vitro en structures multimériques d’environ 32 nm ressemblant à des capsides rétrovirales [9]. De plus, à la fois Arc endogène dans les neurones et Arc purifiée forment un complexe avec l’ARNm Arc, rappelant l’association entre Gag et l’ARN génomique rétroviral. Cette association protège l’ARNm Arc et facilite la formation des capsides, deux effets également observés dans l’assemblage des rétrovirus [9]. De façon encore plus remarquable, Arc peut convoyer « son » ARNm d’une cellule à l’autre. En effet, les dendrites de neurones dépourvus du gène Arc deviennent positives pour l’ARNm et la protéine Arc lorsque ces neurones sont exposés à des capsides Arc produites par des bactéries et purifiées. Ce résultat est aussi observé lorsque les neurones receveurs sont exposés à des vésicules extracellulaires de neurones primaires qui contiennent naturellement Arc [9]. Le transfert intercellulaire implique la formation de capsides chargées dans les neurones donneurs, leur relargage dans les vésicules extracellulaires, et leur endocytose dépendante de la clathrine dans les neurones receveurs (Figure 2) [9]. L’activité neuronale stimule le relargage des capsides chargées par les neurones donneurs et la traduction de l’ARNm transféré dans les neurones receveurs [9]. Les capsides Arc chargées reproduisent donc deux propriétés (rétro)virales : la capacité à sortir d’une cellule et celle d’entrer dans une autre cellule. Comme dans le cas d’une infection (rétro)virale, ces deux étapes permettent le transfert intercellulaire d’une protéine et d’une information génétique codant cette protéine. Dans le cas des capsides Arc, les mécanismes de ce transfert restent cependant à éclaircir (Figure 2) [5, 9].
 |
Figure 1. Ressemblances entre les protéines Arc, dArc1, dArc2 et les protéines Gag de rétrovirus et de rétrotransposons à LTR. La polyprotéine Gag du rétrovirus de l’immunodéficience humaine (VIH) est clivée (triangles) en 4 protéines : matrice (MA) qui cible les membranes, capside (CA) qui s’oligomérise en capside, nucléocapside (NC) qui interagit avec les ARN génomiques, P6 impliquée dans la libération des virus hors des cellules. CA se compose de deux sous-domaines (CANt et CACt) présentant certaines similitudes entre eux (hachures) et résultant vraisemblablement d’une duplication [4, 7, 8]. La partie CACt est plus constante parmi les rétrovirus [2]. La protéine Arc des tétrapodes (celle du rat est donnée en exemple) dérive de Gag d’un rétrotransposon de type Ty3/gypsy. Arc a conservé les deux sous-domaines de CA qui présentent une ressemblance plus grande avec le domaine CACt du HIV [2, 7, 8]. Arc et Gag de certains rétrotransposons de type Ty3/gypsy (un exemple ici avec D.r.Gypsy10 du poisson Danio rerio [8]) possèdent un domaine N-terminal présentant des similitudes de séquence avec la région MA de Gag du HIV [4, 7, 8]. La ressemblance est limitée, et la fonction du domaine MA présumé de Arc reste à éclaircir. dArc1 dérive elle aussi de la protéine Gag d’un rétrotransposon Ty3/gypsy, et présente également les deux sous-domaines de CA. dArc1 possède de plus, dans sa partie C-terminale, un doigt à zinc de structure CX2CX4HX4C (en rouge) dans un contexte basique (petits carrés) [7, 8, 10]. Cette structure est rencontrée dans la région NC de nombreux rétrovirus et rétrotransposons Ty3/gypsy, mais pas tous. Elle est présente en deux exemplaires dans le HIV et en un seul dans D.r.Gypsy10 ou S.c.Ty3. Elle est absente dans Arc, ce qui indique, avec d’autres arguments, que Arc et darc1 correspondent à deux domestications indépendantes de rétrotransposons apparentés mais distincts [7-10]. Un second gène darc2 code, chez la drosophile, une protéine très similaire à dArc1 mais sans le doigt à zinc CX2CX4HX4C. Comme dArc1, dArc2 est relarguée dans des vésicules extracellulaires, probablement sous forme multimérique, mais son ARNm, dépourvu de la région 3’UTR de l’ARNm darc1 n’est pas encapsidé [10]. De plus, un pseudo-gène résultant d’une duplication partielle de darc1 s’intercale entre darc1 et darc2 dans au moins une souche de drosophile. Cette duplication altère l’expression de darc1, et, par conséquent, le développement des synapses neuro-musculaires et le phénotype des mutants darc1 [10]. Les clivages produisant de petits peptides (spacers) ne sont pas indiqués ; les clivages éventuels de Gag de D.r.Gypsy10 ne sont pas connus. |
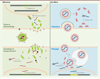 |
Figure 2. Transfert intercellulaire des particules Arc et dArc1 et comparaison avec le cycle des rétrovirus. À gauche, le cycle simplifié d’un rétrovirus. En général, le transfert d’un rétrovirus implique son assemblage et bourgeonnement au niveau de la membrane plasmique, sa maturation (clivage protéolytique des polyprotéines Gag et Gag-Pol), puis l’interaction entre ses protéines d’enveloppe et un récepteur. Le plus souvent, cette interaction permet la fusion entre les membranes virale et cellulaire et l’entrée de la capside, son désassemblage plus ou moins poussé, la rétro-transcription des ARN rétroviraux, puis l’intégration de l’ADN issu de cette rétro-transcription dans le génome de l’hôte. Ce scénario connaît des variantes selon le type de rétrovirus. Dans le cas des particules Arc et dArc1, la sortie s’opérerait via des exosomes relargués par fusion de corps multivésiculaires (multivesicular bodies) à la membrane plasmique et l’entrée par endocytose de ces exosomes. Les particules dArc1 sont transférées des motoneurones aux cellules musculaires à travers la jonction neuro-musculaire, tandis que les particules Arc voyagent vraisemblablement entre les dendrites [9, 10]. Les ARNm Arc ou darc1 transportés ne sont pas rétro-transcrits mais traduits pour fournir, sans doute avec les protéines des capsides, un stock de nouvelles protéines Arc/dArc1. Dans le cas de l’ARNm Arc, cette traduction est stimulée par l’activation des récepteurs du glutamate mGluR1/5 [9]. Notons qu’une sortie via des exosomes et/ou une entrée par endocytose a également été proposée pour certains rétrovirus dans différentes cellules. De nombreux aspects du transfert des particules Arc/dArc1 sont encore mal connus, notamment le site de leur assemblage dans la cellule émettrice, leur sortie des endosomes et la libération des ARN dans le cytoplasme des cellules receveuses. Le nombre et la nature des ARN transportés par les particules Arc et dArc1 restent à préciser. Les modalités du trafic des particules Arc et dArc1 sont éventuellement différentes. |
Des résultats proches ont été obtenus avec la protéine dArc1 de la mouche drosophile (Drosophila melanogaster) (Figure 1) [10]. En effet, dArc1 dérive de la région Gag d’un rétrotransposon Ty3/gypsy et forme des capsides capables de s’associer à l’ARNm darc1 et de sortir des cellules via des vésicules extracellulaires. In vivo, ces capsides chargées sont transférées des motoneurones vers le muscle à la jonction neuromusculaire. Ce transfert trans-synaptique unidirectionnel concentre dArc1 dans le compartiment post-synaptique de la fibre musculaire, et apparaît nécessaire à la plasticité de la synapse neuro-musculaire [10].
En résumé, Arc et dArc1 ont conservé de leur origine la propriété de former des particules capables de transférer leur ARNm d’une cellule à l’autre, propriété utilisée par l’hôte pour servir ses propres fonctions. Arc et dArc1 rejoignent donc la liste croissante des reliques rétrovirales « domestiquées6 » au cours de l’évolution. Pour Arc/dArc1, comme pour les autres exemples, la domestication concerne un seul composant rétroviral, séquence régulatrice ou protéine [2, 4-12] (→).
(→) Voir les Synthèses de A. Dupressoir et T. Heidmann, m/s n° 2, février 2011, page 163, et de V. Adoue et O. Joffre, m/s n° 3, mars 2020, page 253
La particularité remarquable du cas de Arc/dArc1 est que la domestication d’un seul composant s’accompagne du recyclage d’une partie du cycle rétroviral (Figure 2).
Quelles autres propriétés de Gag ont-elles pu être recyclées dans Arc ? Arc régule l’endocytose des récepteurs AMPAR en interagissant avec leurs partenaires, la machinerie d’endocytose et le cytosquelette. Le domaine N-terminal de la région CA de Arc (CANt, Figure 1 ) s’associe avec des partenaires des AMPAR et des régulateurs du cytosquelette. Si l’interaction avec le cytosquelette est possiblement une propriété ancestrale de Gag, l’association avec les partenaires des AMPAR est spécifique de Arc [6, 7]. Le domaine N-terminal de la région CA de Arc a donc été refaçonné par l’évolution pour servir les fonctions de l’hôte. Il constitue une cible potentielle pour des molécules destinées à traiter les désordres neurologiques peut-être liés à une suractivité de Arc [6, 7]. La régulation des AMPAR dépend aussi de la moitié N-terminale de Arc qui présente quelques similitudes avec la portion MA (matrice) de Gag des rétrovirus [4, 6] (Figure 1) : cette région pourrait avoir conservé la fonction de ciblage membranaire de MA, et acquis une capacité à interagir avec la machinerie d’endocytose au cours de la domestication [6]. Toutefois l’origine de cette région, absente dans dArc1, reste incertaine [7, 8].
L’histoire des gènes Arc est d’ailleurs intrigante puisque, malgré leurs ressemblances structurales et fonctionnelles, Arc et darc1 ne dérivent pas d’un ancêtre commun mais résultent, chez les tétrapodes et chez les mouches7, de deux domestications indépendantes de deux lignages distincts de rétrotransposons Ty3/gypsy [5, 7-10]. Une telle convergence évolutive a été rapportée pour la domestication d’autres protéines rétrovirales [5, 11]. Ce scénario évolutif se traduit par l’absence de gène Arc identifiable chez de nombreuses espèces animales, poissons ou insectes distincts des mouches par exemple [7, 9, 10]. Comment ces espèces se passent-elles de Arc ? Ont-elles domestiqué d’autres protéines Gag pour réaliser des fonctions similaires ?
D’autres questions importantes demeurent, par exemple, la diversité exacte des ARN chargés par les capsides Arc et dArc1 [5]. Les capsides Arc produites en bactérie sont capables de charger n’importe quel ARN fortement exprimé. In vivo, il est donc probable qu’elles s’associent préférentiellement avec les ARN les plus représentés dans les dendrites, parmi lesquels l’ARNm Arc [9]. Les capsides dArc1 se montrent, quant à elles, plus sélectives puisque l’encapsidation in vivo dépend de la partie 3’ non traduite de l’ARNm darc1 [10]. Il serait intéressant de savoir si la séquence ou la longueur de cette partie 3’ non traduite est le paramètre déterminant, et si la sélectivité implique la région C-terminale de dArc1, absente dans Arc, et ressemblant à la région NC (nucléocapside) des protéines Gag rétrovirales (Figure 1) [7-10]. Par ailleurs, si le transfert intercellulaire des particules dArc1 est clair, celui des particules Arc reste à établir formellement. L’existence d’un transfert intercellulaire de Arc est en effet déduite d’expériences utilisant des particules purifiées ou des vésicules extracellulaires, mais n’a pas encore été directement montrée entre deux neurones [9]. De plus, l’absence de Arc dans les terminaisons axonales suggère que Arc sert la communication entre dendrites, plutôt qu’entre neurones. Cette communication permettrait la propagation des modifications fonctionnelles contrôlées par Arc dans les dendrites [3, 10]. Si le transfert intercellulaire de Arc était confirmé, il pourrait s’accompagner de la transmission d’états transcriptionnels entre neurones [13]. De tels phénomènes pourraient aider à comprendre le rôle de Arc dans la mémorisation [13], et pourraient même avoir une portée plus générale s’il s’avère que d’autres protéines Gag ont été domestiquées pour un usage similaire dans d’autres types cellulaires [5, 8-10].
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Les auteurs remercient le Dr Cédric Feschotte pour sa relecture du manuscrit et ses suggestions et corrections.
La famille des metaviridae Ty3/gypsy comporte trois genres : les métavirus, tels que le rétrotransposon Ty3 de la levure Saccharomyces cerevisiae, qui ne possèdent pas de gène env ; les errantivirus, tels que le rétrotransposon gypsy de la mouche drosophile, qui possèdent un gène env ; et les chromovirus, présents chez de nombreux eucaryotes, qui possèdent un chromodomaine dans l’intégrase.
Références
- Kim FJ, Manel N, Battini JL, Sitbon M. La capture d’enveloppe par les rétrovirus. Med Sci (Paris) 2004 ; 20 : 876–881. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Dodonova SO, Prinz S, Bilanchone V, et al. Structure of the Ty3/Gypsy retrotransposon capsid and the evolution of retroviruses. Proc Natl Acad Sci USA 2019 ; 116 : 10048–10057. [CrossRef] [Google Scholar]
- Shepherd JD, Bear MF. New views of Arc, a master regulator of synaptic plasticity. Nat Neurosci 2011 ; 14 : 279–284. [CrossRef] [PubMed] [Google Scholar]
- Shepherd JD. Arc: an endogenous neuronal retrovirus?. Semin Cell Dev Biol 2018 ; 77 : 73–78. [CrossRef] [PubMed] [Google Scholar]
- Parrish NF, Tomonaga K. A viral (Arc)hive for metazoan memory. Cell 2018 ; 172 : 8–10. [CrossRef] [PubMed] [Google Scholar]
- Campioni MR. Finkbeiner, S. Going retro: ancient viral origins of cognition. Neuron 2015 ; 86 : 346–348. [CrossRef] [PubMed] [Google Scholar]
- Zhang W, Wu J, Ward MD, et al. Structural basis of Arc binding to synaptic proteins: implications for cognitive disease. Neuron 2015 ; 86 : 490–500. [CrossRef] [PubMed] [Google Scholar]
- Campillos M, Doerks T, Shah PK, Bork P. Computational characterization of multiple Gag-like human proteins. Trends Genet 2006 ; 22 : 585–589. [CrossRef] [PubMed] [Google Scholar]
- Pastuzyn ED, Day CE, Kearns RB, et al. The neuronal gene Arc encodes a repurposed retrotransposon Gag protein that mediates intercellular RNA transfer. Cell 2018 ; 172 : 275–288. [CrossRef] [PubMed] [Google Scholar]
- Ashley J, Cordy B, Lucia D, et al. Retrovirus-like Gag protein Arc1 binds RNA and traffics across synaptic boutons. Cell 2018 ; 172 : 262–274. [CrossRef] [PubMed] [Google Scholar]
- Dupressoir A, Heidmann T. Les syncytines : des protéines d’enveloppe rétrovirales capturées au profit du développement placentaire. Med Sci (Paris) 2011 ; 27 : 163–169. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Adoue V, Joffre O. Les rétrovirus endogènes : un rôle clé dans la programmation des lymphocytes T CD4. Med Sci (Paris) 2020; 36 : 253–60. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Abraham WC, Jones OD, Glanzman DL. Is plasticity of synapses the mechanism of long-term memory storage?. NPJ Sci Learn 2019 ; 4 : 9. [CrossRef] [PubMed] [Google Scholar]
© 2020 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Ressemblances entre les protéines Arc, dArc1, dArc2 et les protéines Gag de rétrovirus et de rétrotransposons à LTR. La polyprotéine Gag du rétrovirus de l’immunodéficience humaine (VIH) est clivée (triangles) en 4 protéines : matrice (MA) qui cible les membranes, capside (CA) qui s’oligomérise en capside, nucléocapside (NC) qui interagit avec les ARN génomiques, P6 impliquée dans la libération des virus hors des cellules. CA se compose de deux sous-domaines (CANt et CACt) présentant certaines similitudes entre eux (hachures) et résultant vraisemblablement d’une duplication [4, 7, 8]. La partie CACt est plus constante parmi les rétrovirus [2]. La protéine Arc des tétrapodes (celle du rat est donnée en exemple) dérive de Gag d’un rétrotransposon de type Ty3/gypsy. Arc a conservé les deux sous-domaines de CA qui présentent une ressemblance plus grande avec le domaine CACt du HIV [2, 7, 8]. Arc et Gag de certains rétrotransposons de type Ty3/gypsy (un exemple ici avec D.r.Gypsy10 du poisson Danio rerio [8]) possèdent un domaine N-terminal présentant des similitudes de séquence avec la région MA de Gag du HIV [4, 7, 8]. La ressemblance est limitée, et la fonction du domaine MA présumé de Arc reste à éclaircir. dArc1 dérive elle aussi de la protéine Gag d’un rétrotransposon Ty3/gypsy, et présente également les deux sous-domaines de CA. dArc1 possède de plus, dans sa partie C-terminale, un doigt à zinc de structure CX2CX4HX4C (en rouge) dans un contexte basique (petits carrés) [7, 8, 10]. Cette structure est rencontrée dans la région NC de nombreux rétrovirus et rétrotransposons Ty3/gypsy, mais pas tous. Elle est présente en deux exemplaires dans le HIV et en un seul dans D.r.Gypsy10 ou S.c.Ty3. Elle est absente dans Arc, ce qui indique, avec d’autres arguments, que Arc et darc1 correspondent à deux domestications indépendantes de rétrotransposons apparentés mais distincts [7-10]. Un second gène darc2 code, chez la drosophile, une protéine très similaire à dArc1 mais sans le doigt à zinc CX2CX4HX4C. Comme dArc1, dArc2 est relarguée dans des vésicules extracellulaires, probablement sous forme multimérique, mais son ARNm, dépourvu de la région 3’UTR de l’ARNm darc1 n’est pas encapsidé [10]. De plus, un pseudo-gène résultant d’une duplication partielle de darc1 s’intercale entre darc1 et darc2 dans au moins une souche de drosophile. Cette duplication altère l’expression de darc1, et, par conséquent, le développement des synapses neuro-musculaires et le phénotype des mutants darc1 [10]. Les clivages produisant de petits peptides (spacers) ne sont pas indiqués ; les clivages éventuels de Gag de D.r.Gypsy10 ne sont pas connus. |
| Dans le texte | |
 |
Figure 2. Transfert intercellulaire des particules Arc et dArc1 et comparaison avec le cycle des rétrovirus. À gauche, le cycle simplifié d’un rétrovirus. En général, le transfert d’un rétrovirus implique son assemblage et bourgeonnement au niveau de la membrane plasmique, sa maturation (clivage protéolytique des polyprotéines Gag et Gag-Pol), puis l’interaction entre ses protéines d’enveloppe et un récepteur. Le plus souvent, cette interaction permet la fusion entre les membranes virale et cellulaire et l’entrée de la capside, son désassemblage plus ou moins poussé, la rétro-transcription des ARN rétroviraux, puis l’intégration de l’ADN issu de cette rétro-transcription dans le génome de l’hôte. Ce scénario connaît des variantes selon le type de rétrovirus. Dans le cas des particules Arc et dArc1, la sortie s’opérerait via des exosomes relargués par fusion de corps multivésiculaires (multivesicular bodies) à la membrane plasmique et l’entrée par endocytose de ces exosomes. Les particules dArc1 sont transférées des motoneurones aux cellules musculaires à travers la jonction neuro-musculaire, tandis que les particules Arc voyagent vraisemblablement entre les dendrites [9, 10]. Les ARNm Arc ou darc1 transportés ne sont pas rétro-transcrits mais traduits pour fournir, sans doute avec les protéines des capsides, un stock de nouvelles protéines Arc/dArc1. Dans le cas de l’ARNm Arc, cette traduction est stimulée par l’activation des récepteurs du glutamate mGluR1/5 [9]. Notons qu’une sortie via des exosomes et/ou une entrée par endocytose a également été proposée pour certains rétrovirus dans différentes cellules. De nombreux aspects du transfert des particules Arc/dArc1 sont encore mal connus, notamment le site de leur assemblage dans la cellule émettrice, leur sortie des endosomes et la libération des ARN dans le cytoplasme des cellules receveuses. Le nombre et la nature des ARN transportés par les particules Arc et dArc1 restent à préciser. Les modalités du trafic des particules Arc et dArc1 sont éventuellement différentes. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




