| Numéro |
Med Sci (Paris)
Volume 29, Numéro 5, Mai 2013
|
|
|---|---|---|
| Page(s) | 487 - 494 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2013295011 | |
| Publié en ligne | 28 mai 2013 | |
Les piARN forgent un système immunitaire pour le génome
The piRNAs forge an immune system for the genome
1
European molecular biology laboratory (EMBL), 6, rue Jules Horowitz, BP 181, 38042 Grenoble, France
2
Center for biological sequence analysis (CBS), Technical University of Denmark (DTU), Kgs. Lyngby, Danemark
*
muller.sbr@gmail.com
muller@cbs.dtu.dk
**
rrpandey@embl.fr
***
pillai@embl.fr
L’intégrité du génome des lignées germinales est essentielle pour la survie des espèces. Un mécanisme de défense basé sur l’utilisation de petits ARN appelés piARN (PIWI-interacting RNA) a évolué pour protéger ces lignées des effets délétères des éléments transposables. La machinerie piARN élabore à l’échelle du génome une réponse similaire à celle du système immunitaire. La reconnaissance de la menace est assurée par une complémentarité de séquence entre un vaste répertoire de piARN et les intrus, et déclenche une dégradation adaptée et mémorisée de la cible. Cette revue résume les connaissances actuelles de cette surprenante immunité qui protège la transmission de l’information génétique.
Abstract
Genome integrity of germline is essential for the survival of any species. A dedicated defence mechanism based on small RNA called piRNA (PIWI-interacting RNA) has evolved to protect the germline from the deleterious effects of transposon mobility in genomes such as mutations, deletions or chromosomal rearrangements. The piRNA machinery ensures genomic integrity to germ cells by setting a response similar to the immune system. The recognition of the threat is mediated by sequence complementarity between a vast repertoire of piRNAs and the intruders, and initiates a rapid and efficient degradation of the targets. Akin to acquired immunity, the response is memorized throughout generations thanks to epigenetic modifications. Investigations are progressing to unravel the mysterious mechanisms of this exciting class of non coding RNAs. This review summarizes some of the recent advances on this exceptional immunity that protects transmission of genetic information.
© 2013 médecine/sciences – Inserm
Les éléments transposables, une menace pour le génome
Le génome est menacé par de nombreux événements extérieurs, tels que des agressions physiques ou l’exposition à des facteurs mutagènes (radiations ionisantes ou réactifs toxiques) qui provoquent des mutations, voire d’importants réarrangements au sein de l’ADN. À cela s’ajoute un danger intrinsèque, porté par le génome lui-même : les séquences répétées mobiles appelées éléments transposables. Ces éléments sont constitués par les rétrotransposons et les transposons ADN qui transportent du matériel génétique d’un endroit du génome à un autre par des mécanismes de copier-coller et couper-coller, avec des conséquences potentiellement délétères pour le génome [1]. Malgré le danger qu’ils représentent, ces éléments transposables couvrent près de la moitié du génome humain. Leur mobilité et leur grand nombre ont conféré une inestimable malléabilité au remodelage des génomes au cours de l’évolution [2]. Bien que la majorité de ces éléments transposables soient inactifs (état d’ADN fossile) parce que modifiés par des mutations, certains restent actifs ou peuvent être réactivés. Par exemple, des rétrotransposons de la classe LINE1 (long interspersed elements), au nombre d’environ 500 000 copies dans les génomes de mammifères, comptent respectivement 100 et 3 000 copies actives chez l’homme et la souris [2], et certaines d’entre elles sont impliquées dans des maladies [3].
Plusieurs systèmes de défense contrecarrent l’action et l’expansion préjudiciables de ces éléments transposables. Chez les mammifères, la méthylation de l’ADN, ainsi que la modification des histones, sont indispensables pour inhiber l’expression des éléments transposables. Cette inactivation est ensuite transmise aux cellules filles via un mécanisme de contrôle dit épigénétique. Un autre niveau de contrôle est assuré par de petits ARN, de taille comprise entre 20 et 30 nucléotides (nt). Ces petits ARN semblent jouer un rôle particulièrement important dans les lignées germinales. En effet, la lignée germinale (ou sexuelle) est celle qui transmet les informations génétiques à la génération suivante. Puisque toute modification du patrimoine génétique de ces cellules sera transmise aux descendants, il est primordial que la stabilité du génome des lignées germinales, plus encore que celle des lignées somatiques (non sexuelles), soit garantie pour le bien des individus et de leur espèce. De plus, il existe chez les mammifères une fenêtre critique, de la fin de l’embryogenèse jusqu’à quelques jours après la naissance, pendant laquelle la méthylation du génome des cellules germinales est remodelée, ce qui rend ces cellules, qui sont en cours de différenciation, particulièrement vulnérables à l’envahissement par les éléments transposables. Durant cette période, le génome peut bénéficier de la présence du système de défense particulier basé sur l’utilisation de petits ARN appelés piARN (PIWI-interacting RNA) [4–7, 38, 39], dont l’action pourrait être comparée à celle d’un système immunitaire à l’échelle du génome. Cette revue résume les connaissances que nous avons de cette classe d’ARN dont la caractérisation a débuté en 2006.
Caractéristiques des piARN
Les piARN ont été détectés dans les cellules des lignées germinales de tous les animaux étudiés à ce jour et semblent apparaître très tôt au cours de l’évolution, chez l’ancêtre commun de tous les animaux [8]. Dans tous ces organismes, ils sont pressentis pour être les acteurs majeurs de la lutte contre les éléments transposables au cours de la gamétogenèse. À noter qu’une autre classe de petits ARN, les endo-siARN (small interfering RNA) endogènes semblent également impliqués dans la répression des éléments transposables. Leur action semble être importante dans les tissus somatiques [9, 10]. Bien qu’il existe des ressemblances avec les siARN et microARN (miARN), les piARN s’en démarquent par plusieurs particularités [11]. Ainsi, les piARN ont une taille comprise entre 21 et 30 nt, contre environ 21 nt pour les siARN, et environ 22 nt pour les miARN. Ces trois classes d’ARN interagissent avec des protéines Argonautes organisées en deux sous-familles : AGO et PIWI. Les protéines AGO interagissent avec les miARN et siARN [39], tandis que les protéines PIWI s’associent aux piARN. Chez les mammifères, si l’on compte de l’ordre d’un millier de séquences uniques de miARN, il en existe plus d’un million pour les piARN. Contrairement aux miARN, les séquences des piARN sont très peu conservées entre les espèces, et seules leurs positions relatives dans le génome (synthénie) semblent être conservées. Enfin la biogenèse des piARN se distingue de celle des miARN et siARN [39–41].
Biogenèse des piARN
Alors que les miARN requièrent une maturation à partir de structures en tige boucle caractéristiques et les siARN la présence de double brins d’ARN [40, 41], les piARN semblent être issus de longs précurseurs d’ARN simple brin par un processus encore énigmatique [11, 12]. Les résultats de séquençages modernes montrent que les millions de piARN différents proviennent d’un nombre limité de locus, une centaine chez les mammifères, dans lesquels la plupart sont regroupés en clusters. Dans ces clusters, les piARN sont issus d’un des deux brins, mais ne se chevauchent pas [4, 5]. Ces clusters sont majoritairement localisés dans des régions riches en répétitions ou encore dans les régions intergéniques. En effet, leur provenance peut varier au sein de différentes sous-populations de piARN propres à un stade de différenciation. Par exemple, chez les mammifères, nous pouvons distinguer des populations de piARN caractéristiques des stades de prépachytène ou du pachytène. Il est à noter également que deux à quatre protéines PIWI différentes sont systématiquement identifiées chez les animaux et interagissent avec des piARN de taille et de nature différentes. La biogenèse des piARN présente encore de nombreuses zones d’ombre. Dans un premier temps, une endonucléase appelée Zucchini génère les précurseurs ou pré-piARN (Figure 1) [13, 14]. Chez le ver C. elegans, une séquence présente en amont permet de recruter cette endonucléase [15]. Chez les autres espèces, on ne sait pas ce qui définit ces régions comme clusters de piARN. Les pré-piARN sont reconnus par une protéine PIWI qui est responsable d’un biais de sélection des candidats. En effet, les piARN primaires présentent plus fréquemment un résidu U en 5’, signature du processus primaire de genèse des piARN. Associé à cette protéine PIWI, le pré-piARN est soumis à l’action d’une exonucléase inconnue qui réduit sa taille à 21-30 nt [16]. La gêne stérique exercée par la protéine PIWI explique la différence des tailles de piARN. Ces piARN primaires doivent encore subir une étape de méthylation de leur extrémité 3’ par HEN1 (HUA enhancer 1), modification censée les protéger d’une dégradation précoce [17]. Le piARN primaire peut guider, par appariement total ou partiel, la protéine PIWI sur sa cible, par exemple l’ARNm d’un rétrotransposon. L’appariement du piARN avec sa cible conduit une protéine PIWI à cliver la cible, donnant naissance à l’extrémité 5’ d’un nouveau piARN, dit secondaire. Le clivage s’effectue toujours au même endroit, au niveau du nucléotide qui s’apparie avec le 10e nucléotide du piARN. Par conséquent, le piARN secondaire présente deux signatures : d’une part, sa séquence est complémentaire de celle du piARN primaire sur les 10 premiers nucléotides et, d’autre part, l’enrichissement en piARN affichant un U en 1re position se traduit dans les piARN secondaires par un enrichissement en A en position 10. Ce piARN secondaire peut conduire la même protéine PIWI, ou une autre protéine, à cliver sa cible (cluster de piARN), ce qui génère un nouveau piARN dit secondaire, car généré par le biais de protéines PIWI, mais semblable au piARN primaire. Puisque seule la cible du piARN est clivée, le piARN peut être réutilisé par les protéines PIWI pour diriger le clivage d’une autre cible. Ainsi, le cycle décrit revient à amplifier l’action des piARN. Ce cycle d’amplification est communément appelé cycle ping-pong [38], et contribue, en totalité ou partiellement, à la biogenèse des piARN (voir plus loin).
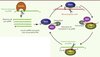 |
Figure 1. Modèle théorique de la biogenèse primaire des piARN et de l’amplification par cycle ping-pong. L’endonucléase Zucchini est responsable de la maturation de transcrits portant les clusters de piARN. Les pré-piARN sont reconnus par une protéine PIWI (ici PIWI A), qui a une préférence pour les séquences présentant un U en 1re position. Associés à PIWI A, les pré-piARN doivent encore subir une maturation par une exonucléase 3’-5’, ainsi que par HEN1 qui apporte un groupement méthyl en 2’O de l’extrémité 3’ ce qui les rend fonctionnels. Les piARN primaires ainsi générés peuvent s’engager dans le cycle ping-pong. Par complémentarité de séquence, le piARN primaire dirige le clivage du rétrotransposon ciblé (brin sens ou antisens) par la protéine PIWI A qui porte l’activité endonucléase. La nouvelle extrémité 5’ générée est reconnue par une autre protéine PIWI (ici PIWI B), mais cette protéine peut être la même que la première. Il est à remarquer que l’extrémité 3’ du piARN secondaire est maturée et modifiée comme l’est le piARN primaire. Le piARN secondaire peut diriger l’activité de clivage de PIWI B sur le transcrit portant les clusters de piARN, générant ainsi un nouveau piARN secondaire dont la séquence est identique au piARN primaire, et ainsi de suite. Les piARN dont l’extrémité 5’ est générée par l’action de Zucchini sont appelés piARN primaires, alors que les piARN dont l’extrémité 5’ est générée par les protéines PIWI sont appelés piARN secondaires. Ainsi, les piARN notés A10 représentent des piARN secondaires, alors que les piARN notés U1 peuvent être primaires ou secondaires. |
Modes d’action des piARN
Les piARN inactivent les éléments transposables par différents mécanismes. Le plus évident correspond à l’activité endonucléase des protéines PIWI, qui leur permet de cliver la cible et donc d’engager la dégradation de l’ARNm d’un rétrotransposon. Pour cela, il est nécessaire de générer un piARN primaire ou secondaire capable de s’apparier à la cible (piARN antisens), mais aussi de recruter une protéine PIWI catalytiquement active. Ce dernier point a été examiné en mutant la triade catalytique DDH des protéines PIWI et en mesurant les effets sur les populations de piARN présentant les signatures précédemment énoncées [18, 19].
Mécanisme d’action des piARN chez la souris
Chez la souris, il existe trois protéines PIWI appelées Mili, Miwi et Miwi2. Seule Mili est exprimée dans les deux sexes. Chez le mâle, Mili est exprimée du stade embryonnaire jusqu’au stade de spermatides ronds, Miwi2 est détectée du stade embryonnaire jusqu’à trois jours après la naissance, et Miwi est exprimée au stade spermatocyte du pachytène et spermatide rond (Figure 2). Lors de la spermatogenèse, ces trois protéines PIWI ont des rôles différents selon les populations de piARN du prépachytène et du pachytène auxquelles elles sont associées [39]. Dans la population des piARN du prépachytène, les piARN primaires s’associant avec Mili génèrent, après clivage de la cible, des piARN secondaires associés à Miwi2 [18, 20, 21]. Miwi2 se déplace dans le noyau et guide la méthylation de l’ADN des éléments transposables [18, 21]. Étant donné que Miwi2 ne présente pas d’activité endonucléase (contrairement à Mili et Miwi), il ne peut y avoir de cycle ping-pong entre Mili et Miwi2 [18]. À noter toutefois qu’une amplification cyclique par ping-pong est observée entre deux protéines Mili. En résumé, se met en place un transfert entre Mili qui dégrade les ARNm des transposons et peut amplifier le signal par cycle ping-pong, jusqu’à Miwi2, qui influencera à plus long terme l’expression de ce transposon, puisque les cellules filles hériteront des marques épigénétiques que sont les méthylations de l’ADN. La réponse à l’expression d’un rétrotransposon au stade prépachytène est donc de deux ordres : une répression post-transcriptionnelle rapide par Mili et, coordonnée à celle-ci, une répression épigénétique par Miwi2. La réaction impliquant la machinerie piARN est donc adaptée, rapide, amplifiée et mémorisée, rappelant celle du système immunitaire. Au stade postnatal, les piARN du pachytène s’associent avec Miwi qui est dotée d’une activité endonucléase, et répriment ainsi les rétrotranposons qui auraient échappé à la première vague de répression [19]. Miwi exercerait un rôle de surveillance, mais son interaction directe avec des ARNm, de même que le rôle des piARN au stade pachytène, restent à être explorés [22]. Notons également que Mili et Miwi interagissent avec la machinerie traductionnelle, ce qui laisse entrevoir une action de répression traductionnelle [23].
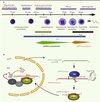 |
Figure 2. Rôle des piARN dans l’inactivation de rétrotransposons chez la souris. A. Expression des protéines PIWI et des piARN au cours de la spermatogenèse de souris. Après leur migration vers les gonades, les cellules germinales primordiales se multiplient jusqu’à l’arrêt vers 15 jpc. Commence alors l’établissement de la méthylation de novo de l’ADN. Les spermatogonies reprennent leurs divisions à 3 jpp et débutent leur méiose à 10 jpp. Les premiers spermatides ronds haploïdes apparaissent à 14 jpp. jpc : jours post-coïtum ; jpp : jours post-partum. B. Biogenèse et fonction des piARN du pachytène. Après export du noyau, le transcrit portant les clusters de piARN génère des piARN primaires qui s’associent avec Mili. L’activité endonucléase de Mili clive sa cible pour donner naissance à un piARN secondaire. Notons qu’un cycle ping-pong entre deux protéines Mili peut amplifier la production de piARN secondaires. Le piARN secondaire associé à Miwi2 peut entrer dans le noyau et guider la méthylation de l’ADN des rétrotransposons en s’appariant à un transcrit en cours d’élongation pour recruter une machinerie encore inconnue conduisant à la modification des histones et à la méthylation de l’ADN. |
Mécanisme d’action des piARN chez C. elegans
Chez le ver C. elegans, une étude récente a démontré un mode d’action original [24, 25]. En effet, la population de piARN primaires (21U-piARN) guide une protéine PIWI, PRG-1, au contact d’un transcrit correspondant à un rétrotransposon ou à un élément étranger (Figure 3). PRG-1 n’exprime aucune activité endonucléase, mais recrute un complexe ARN-polymérase ARN-dépendant qui génère, en utilisant le transcrit ciblé comme matrice, un petit ARN guide nommé 22G-ARN. Ce guide recrute la protéine Argonaute WAGO9, connue pour son implication dans le système siARN, pour réprimer le rétrotransposon par clivage et par contrôle épigénétique. Le fait qu’un piARN puisse engendrer plusieurs 22G-ARN constitue là encore une étape d’amplification du signal. Cette étude montre aussi que l’appariement entre le piARN et la cible doit être imparfait, ce qui augmente grandement le nombre de cibles potentielles. Aussi, il est intéressant d’observer que l’évolution des séquences des piARN permettrait d’éviter de cibler ses propres ARNm, issus de gènes codant pour des protéines. À moins qu’une protéine Argonaute CSR-1, également associée à des petits ARN guides, puisse reconnaître les ARNm codant pour des protéines, et donc les épargner [25] (Figure 3). Enfin, il semble que la répression initiée par les piARN soit maintenue sur plusieurs générations de vers [26]. Au final, cette distinction entre éléments du soi et du non-soi évoque les mécanismes de l’immunité innée.
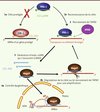 |
Figure 3. Modèle présentant le rôle des piARN dans la distinction entre les ARN du soi et du non-soi chez le ver C. elegans. Dans les lignées germinales, un complexe piARN associé à PRG-1 inspecte les ARN pour identifier des cibles avec une complémentarité imparfaite. Un système basé sur l’utilisation de la protéine CSR-1 et d’endo-siARN permet de protéger les ARNm cellulaires (1a). La reconnaissance de la cible, élément transposable ou élément étranger, par le piARN (1b) permet de recruter une ARNpolymérase- ARN-dépendante (ADAP) (2). Cette dernière génère de multiples petits ARN guides appelés 22G-ARN (3) qui s’associent à Wago9, soit pour amplifier le signal en recrutant ADAP et en générant de nouveaux 22G-ARN, soit pour inactiver le transposon. Deux mécanismes de répression sont observés ; ils interviennent dans le noyau par modifications épigénétiques (4a), et dans le cytoplasme par la dégradation de la cible (4b). |
Mécanisme d’action des piARN chez la drosophile
Chez la drosophile, des piARN primaires antisens sont générés à partir de copies d’éléments transposables dites défectives, car en partie inactives en raison de l’accumulation de mutations et de délétions. Un cycle d’amplification ping-pong permet aux piARN antisens primaires de cibler les copies d’éléments transposables fonctionnelles pour produire des piARN sens qui peuvent, à leur tour, guider les protéines PIWI vers les copies d’éléments transposables défectives pour produire des piARN antisens dits secondaires [12]. Cependant, les piARN primaires ne sont pas les seuls piARN à pouvoir déclencher le cycle d’amplification ping-pong. L’étude de l’élément I, un rétrotransposon exprimé dans la lignée germinale de l’ovaire de drosophile, montre que cette boucle d’amplification peut être enclenchée par les piARN déposés dans l’embryon. En effet, D. melanogaster a subi deux vagues d’invasion par les éléments I. Ainsi, nous observons des souches R (réactive) n’ayant subi qu’une invasion ancienne et ne présentant que des vestiges d’éléments I, et des souches I (inducer) ayant subi une seconde invasion plus récente. Le croisement de femelles R avec des mâles I engendre une progéniture stérile dont les gonades sont dystrophiques, alors que le croisement de femelles I avec des mâles R engendre une progéniture féconde. Ce phénomène, appelé dysgénésie hybride, est longtemps resté inexpliqué avant la découverte des piARN. Il est dû au fait que des piARN maternels sont déposés dans l’embryon et peuvent initier un cycle d’amplification qui permet aux lignées germinales de la génération suivante de se défendre contre les éléments I [27]. Ainsi, les femelles I transmettent à leur descendance suffisamment de piARN spécifiques de l’élément I pour inhiber son expression, alors que la quantité de piARN transmise par les femelles R est insuffisante pour induire son inhibition. Toutefois, les œufs pondus par des femelles R âgées possédent plus de piRNA que ceux pondus par des femelles R jeunes [28]. Ainsi, en vieillissant, une femelle R peut acquérir la capacité de réprimer l’élément I, même à partir de copies d’éléments transposables défectives, capacité qui peut être à son tour transmise de la mère à la fille par le dépôt de piARN dans l’embryon. Ainsi, les piARN peuvent transmettre une information épigénétique sur plusieurs générations.
Les acteurs de la machinerie piARN sont confinés dans des corps cytoplasmiques
Des études génétiques ont montré qu’en plus des protéines PIWI, de nombreux autres facteurs sont indispensables à la machinerie piARN. En effet, la machinerie piARN nécessite la formation d’un gros complexe comparable au complexe RISC (RNA-induced silencing complex) associé avec les miARN [11, 29, 39]. Les protéines PIWI se distinguent des protéines AGO par la présence de méthylations qui recrutent des protéines comportant des domaines Tudor (TDRD) spécifiques aux gonades. Ces dernières constituent une plateforme d’assemblage pour un complexe piRISC spécifique. Une différence avec le complexe miRISC est le confinement des acteurs de la machinerie piARN dans des granules cytoplasmiques observés à certains stades de différenciation des lignées germinales [30]. Ainsi, chez les mammifères, trois structures différentes rassemblent la plupart des acteurs de la machinerie piARN : les ciments intermitochondriaux, les corps P (processing bodies) et les corps chromatoïdes. Ces derniers sont périnucléaires, hébergent le plus grand nombre de ces facteurs, et subissent des remodelages lors de la spermatogenèse [31]. Cependant, nous ne connaissons pas le rôle de ces agrégats dépourvus de membranes. Ils pourraient jouer un rôle dans la dégradation des cibles des piARN ou dans la dégradation des piARN et des protéines associées, mais aussi dans le stockage des piARN pour une future utilisation. En effet, la séquestration des acteurs de cette machinerie dans différentes structures permet de limiter leur action à une fenêtre temporelle précise et évite l’interférence avec les machineries associées à d’autres petits ARN. Ce niveau de régulation supplémentaire reste mal défini et mérite de nouvelles investigations.
Défauts de la machinerie piARN et stérilité masculine
Chez la souris, de façon assez surprenante, toutes les mutations à l’origine d’une perte de fonction des éléments de la machinerie piARN étudiées à ce jour entraînent la stérilité du mâle par arrêt de la spermatogenèse à des stades différents [29]. Bien que les femelles expriment aussi une partie des éléments de la machinerie piARN, elles ne seraient pas affectées pour une raison encore inconnue. Il est suggéré qu’un système basé sur l’utilisation d’endo-siARN pourrait compenser l’absence d’une partie de la machinerie piARN [32, 33]. Cette classe d’endo-siARN pourrait renforcer la répression mise en place par les piARN ; elle nous rappelle que d’autres classes de petits ARN interviennent dans ce processus de répression des éléments transposables [34]. Puisque toute défaillance de la machinerie piARN se traduit chez les mâles par une dégénérescence sévère des cellules germinales, il serait pertinent de connaître l’implication de la machinerie piARN dans la stérilité de l’homme. Une étude a par exemple identifié, en étudiant 490 patients atteints d’azoospermie ou d’oligospermie, neuf marques de polymorphisme (SNP, single nucleotide polymorphism) dans les protéines PIWI [35]. En outre, une surexpression de HIWI, l’équivalent humain de Miwi, est associée à certains cancers [36], suggérant une possible implication des protéines PIWI dans les cellules souches cancéreuses [37]. De plus, l’expression parfois observée de piARN ou de protéines PIWI dans les cellules somatiques pourrait conduire à la pérennisation de certaines propriétés de cellules souches [37]. Au final, il est difficile aujourd’hui de mesurer toute l’étendue des conséquences d’un défaut de la machinerie piARN bien que son rôle dans la différenciation germinale soit clairement établi.
Conclusion
L’exploration de la machinerie piARN a longtemps été limitée parce que son expression est restreinte aux cellules germinales et que la diversité des piARN est importante, puisqu’on compte plus d’un million de séquences uniques, faiblement exprimées et sélectionnées par des mécanismes qui restent encore mystérieux. L’application de techniques modernes de séquençage et l’utilisation de trois modèles d’étude (souris, ver C. elegans et drosophile) nous ont permis de comprendre leur rôle crucial dans l’inactivation des ET. Ce rôle est particulièrement avéré lors d’une phase de la gamétogenèse pendant laquelle le code épigénétique est remodelé, mais aussi lors de la réactivation d’éléments transposables ou lors de contacts avec des vecteurs de ces éléments transposables, tels que les rétrovirus. En cela la machinerie piARN peut être envisagée comme un système immunitaire à l’échelle du génome. À l’image de l’immunité innée, les piARN constituent une première barrière contre les copies actives de rétrotransposons que porte le génome. La machinerie piARN distingue aussi le soi du non-soi par des mécanismes encore peu compris qui reposent notamment sur d’autres petits ARN, tels que les endo-siARN. Dans ce cadre, les piARN guident un complexe portant une activité endonucléase sur la cible à dégrader. Une étape d’amplification garantit l’efficacité de cette action. D’autre part, à l’image de l’immunité acquise et de l’expansion clonale observée avec les lymphocytes produisant les anticorps, la machinerie piARN propose un répertoire important de piARN. À cette variabilité de séquences s’ajoute la possibilité de mésappariements de nucléotides permettant l’accès à de très nombreuses cibles. Au final, seuls sont amplifiés les piARN qui reconnaissent leur cible. Dans ce cadre, en plus de la répression post-transcriptionnelle, les piARN dirigent dans le noyau un complexe qui modifie la méthylation de l’ADN. Ce contrôle épigénétique permet de mémoriser l’inactivation d’une séquence maintenant insérée dans le génome, au sein d’une lignée cellulaire donnée, mais aussi sur plusieurs générations d’individus. Enfin, le fait que les piARN ciblent des éléments autres que les éléments transposables, tels que les gènes de protéines ou les ARN non codants, de même que la présence énigmatique de la machinerie piARN dans les cellules somatiques (et en particulier dans des cellules souches à l’origine de cancer), nous promet encore de nombreuses surprises expérimentales.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
S. Muller a été financé par l’ARC (Association pour la recherche sur le cancer) et R.R. Pandey a été financé par l’EMBO (European molecular biology organization). Nous nous excusons auprès de ceux qui ont grandement contribué à l’exploration de cette voie piARN et dont les travaux, pour des raisons de place, n’apparaissent pas dans cette synthèse.
Références
- Chenais B. Transposable elements and human cancer: A causal relationship? Biochim Biophys Acta 2012 ; 1835 : 28–35. [PubMed] [Google Scholar]
- Kazazian HH, Jr. Mobile elements: drivers of genome evolution. Science 2004 ; 303 : 1626–1632. [CrossRef] [PubMed] [Google Scholar]
- Solyom S, Kazazian HH, Jr. Mobile elements in the human genome: implications for disease. Genome Med 2012 ; 4 : 12. [CrossRef] [PubMed] [Google Scholar]
- Aravin A, Gaidatzis D, Pfeffer S, et al. A novel class of small RNAs bind to MILI protein in mouse testes. Nature 2006 ; 442 : 203–207. [PubMed] [Google Scholar]
- Girard A, Sachidanandam R, Hannon GJ, Carmell MA. A germline-specific class of small RNAs binds mammalian PIWI proteins. Nature 2006 ; 442 : 199–202. [PubMed] [Google Scholar]
- Grivna ST, Beyret E, Wang Z, Lin H. A novel class of small RNAs in mouse spermatogenic cells. Genes Dev 2006 ; 20 : 1709–1714. [CrossRef] [PubMed] [Google Scholar]
- Watanabe T, Takeda A, Tsukiyama T, et al. Identification and characterization of two novel classes of small RNAs in the mouse germline: retrotransposon-derived siRNAs in oocytes and germline small RNAs in testes. Genes Dev 2006 ; 20 : 1732–1743. [CrossRef] [PubMed] [Google Scholar]
- Grimson A, Srivastava M, Fahey B, et al. Early origins and evolution of microRNAs and PIWI-interacting RNAs in animals. Nature 2008 ; 455 : 1193–1197. [CrossRef] [PubMed] [Google Scholar]
- Czech B, Malone CD, Zhou R, et al. An endogenous small interfering RNA pathway in Drosophila. Nature 2008 ; 453 : 798–802. [CrossRef] [PubMed] [Google Scholar]
- Ghildiyal M, Seitz H, Horwich MD, et al. Endogenous siRNAs derived from transposons and mRNAs in Drosophila somatic cells. Science 2008 ; 320 : 1077–1081. [CrossRef] [PubMed] [Google Scholar]
- Siomi MC, Sato K, Pezic D, Aravin AA. PIWI-interacting small RNA: the vanguard of genome defence. Nat Rev Mol Cell Biol 2011 ; 12 : 246–258. [CrossRef] [PubMed] [Google Scholar]
- Brennecke J, Aravin AA, Stark A, et al. Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell 2007 ; 128 : 1089–1103. [CrossRef] [PubMed] [Google Scholar]
- Nishimasu H, Ishizu H, Saito K, et al. Structure and function of Zucchini endoribonuclease in piRNA biogenesis. Nature 2012 ; 491 : 284–287. [CrossRef] [PubMed] [Google Scholar]
- Ipsaro JJ, Haase AD, Knott SR, et al. The structural biochemistry of Zucchini implicates it as a nuclease in piRNA biogenesis. Nature 2012 ; 491 : 279–283. [CrossRef] [PubMed] [Google Scholar]
- Ruby JG, Jan C, Player C, et al. Large-scale sequencing reveals 21U-RNAs and additional microRNAs and endogenous siRNAs in C. elegans. Cell 2006 ; 127 : 1193–1207. [Google Scholar]
- Kawaoka S, Izumi N, Katsuma S, Tomari Y. 3’ end formation of PIWI-interacting RNAs in vitro. Mol Cell 2011 ; 43 : 1015–1022. [CrossRef] [PubMed] [Google Scholar]
- Saito K, Sakaguchi Y, Suzuki T, et al. Pimet, the Drosophila homolog of HEN1, mediates 2’-O-methylation of PIWI-interacting RNAs at their 3’ ends. Genes Dev 2007 ; 21 : 1603–1608. [CrossRef] [PubMed] [Google Scholar]
- De Fazio S, Bartonicek N, Di Giacomo M, et al. The endonuclease activity of Mili fuels piRNA amplification that silences LINE1 elements. Nature 2011 ; 480 : 259–263. [CrossRef] [PubMed] [Google Scholar]
- Reuter M, Berninger P, Chuma S, et al. Miwi catalysis is required for piRNA amplification-independent LINE1 transposon silencing. Nature 2011 ; 480 : 264–267. [CrossRef] [PubMed] [Google Scholar]
- Aravin AA, Sachidanandam R, Girard A, et al. Developmentally regulated piRNA clusters implicate MILI in transposon control. Science 2007 ; 316 : 744–747. [CrossRef] [PubMed] [Google Scholar]
- Aravin AA, Bourc’his D. Small RNA guides for de novo DNA methylation in mammalian germ cells. Genes Dev 2008 ; 22 : 970–975. [CrossRef] [PubMed] [Google Scholar]
- Vourekas A, Zheng Q, Alexiou P, et al. Mili and Miwi target RNA repertoire reveals piRNA biogenesis and function of Miwi in spermiogenesis. Nat Struct Mol Biol 2012 ; 19 : 773–781. [CrossRef] [PubMed] [Google Scholar]
- Grivna ST, Pyhtila B, Lin H. MIWI associates with translational machinery and PIWI-interacting RNAs (piRNAs) in regulating spermatogenesis. Proc Natl Acad Sci USA 2006 ; 103 : 13415–13420. [CrossRef] [Google Scholar]
- Bagijn MP, Goldstein LD, Sapetschnig A, et al. Function, targets, and evolution of Caenorhabditis elegans piRNAs. Science 2012 ; 337 : 574–578. [CrossRef] [PubMed] [Google Scholar]
- Lee HC, Gu W, Shirayama M, et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell 2012 ; 150 : 78–87. [CrossRef] [PubMed] [Google Scholar]
- Ashe A, Sapetschnig A, Weick EM, et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell 2012 ; 150 : 88–99. [CrossRef] [PubMed] [Google Scholar]
- Brennecke J, Malone CD, Aravin AA, et al. An epigenetic role for maternally inherited piRNAs in transposon silencing. Science 2008 ; 322 : 1387–1392. [CrossRef] [PubMed] [Google Scholar]
- Grentzinger T, Armenise C, Brun C, et al. piRNA-mediated transgenerational inheritance of an acquired trait. Genome Res 2012 ; 22 : 1877–1888. [CrossRef] [PubMed] [Google Scholar]
- Pillai RS, Chuma S. piRNAs and their involvement in male germline development in mice. Dev Growth Differ 2012 ; 54 : 78–92. [CrossRef] [PubMed] [Google Scholar]
- Chuma S, Pillai RS. Retrotransposon silencing by piRNAs: ping-pong players mark their sub-cellular boundaries. PLoS Genet 2009 ; 5 : e1000770. [CrossRef] [PubMed] [Google Scholar]
- Tanaka T, Hosokawa M, Vagin VV, et al. Tudor domain containing 7 (Tdrd7) is essential for dynamic ribonucleoprotein (RNP) remodeling of chromatoid bodies during spermatogenesis. Proc Natl Acad Sci USA 2011 ; 108 : 10579–10584. [CrossRef] [Google Scholar]
- Tam OH, Aravin AA, Stein P, et al. Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes. Nature 2008 ; 453 : 534–538. [CrossRef] [PubMed] [Google Scholar]
- Watanabe T, Totoki Y, Toyoda A, et al. Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes. Nature 2008 ; 453 : 539–543. [CrossRef] [PubMed] [Google Scholar]
- Wang J, Li LC. Small RNA and its application in andrology and urology. Transl Androl Urol 2012 ; 1 : 33–43. [PubMed] [Google Scholar]
- Gu A, Ji G, Shi X, et al. Genetic variants in PIWI-interacting RNA pathway genes confer susceptibility to spermatogenic failure in a Chinese population. Hum Reprod 2010 ; 25 : 2955–2961. [CrossRef] [PubMed] [Google Scholar]
- Siddiqi S, Terry M, Matushansky I. Hiwi mediated tumorigenesis is associated with DNA hypermethylation. PLoS One 2012 ; 7 : e33711. [CrossRef] [PubMed] [Google Scholar]
- Bamezai S, Rawat VP, Buske C. The PIWI-piRNA axis: pivotal beyond transposon silencing. Stem Cells 2012 ; 30 : 2603–2611. [CrossRef] [PubMed] [Google Scholar]
- Papin C, Simonelig M. Contrôle du développement embryonnaire par des petits ARN issus de transposons. Med Sci (Paris) 2011 ; 27 : 1050–1052. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Dunoyer P. La bataille du silence : mécanisme et inhibition du RNA silencing au cours des interactions plante/virus. Med Sci (Paris) 2009 ; 25 : 505–511. [Google Scholar]
- Robert V, Bucheton A. Régulation de l’expression des séquences répétées et interférence par l’ARN. Med Sci (Paris) 2004 ; 20 : 767–772. [Google Scholar]
- Romero Y, Calvel P, Nef S. Petits ARN non codants et spermatogenèse. Med Sci (Paris) 2012 ; 28 : 490–496. [Google Scholar]
Liste des figures
 |
Figure 1. Modèle théorique de la biogenèse primaire des piARN et de l’amplification par cycle ping-pong. L’endonucléase Zucchini est responsable de la maturation de transcrits portant les clusters de piARN. Les pré-piARN sont reconnus par une protéine PIWI (ici PIWI A), qui a une préférence pour les séquences présentant un U en 1re position. Associés à PIWI A, les pré-piARN doivent encore subir une maturation par une exonucléase 3’-5’, ainsi que par HEN1 qui apporte un groupement méthyl en 2’O de l’extrémité 3’ ce qui les rend fonctionnels. Les piARN primaires ainsi générés peuvent s’engager dans le cycle ping-pong. Par complémentarité de séquence, le piARN primaire dirige le clivage du rétrotransposon ciblé (brin sens ou antisens) par la protéine PIWI A qui porte l’activité endonucléase. La nouvelle extrémité 5’ générée est reconnue par une autre protéine PIWI (ici PIWI B), mais cette protéine peut être la même que la première. Il est à remarquer que l’extrémité 3’ du piARN secondaire est maturée et modifiée comme l’est le piARN primaire. Le piARN secondaire peut diriger l’activité de clivage de PIWI B sur le transcrit portant les clusters de piARN, générant ainsi un nouveau piARN secondaire dont la séquence est identique au piARN primaire, et ainsi de suite. Les piARN dont l’extrémité 5’ est générée par l’action de Zucchini sont appelés piARN primaires, alors que les piARN dont l’extrémité 5’ est générée par les protéines PIWI sont appelés piARN secondaires. Ainsi, les piARN notés A10 représentent des piARN secondaires, alors que les piARN notés U1 peuvent être primaires ou secondaires. |
| Dans le texte | |
 |
Figure 2. Rôle des piARN dans l’inactivation de rétrotransposons chez la souris. A. Expression des protéines PIWI et des piARN au cours de la spermatogenèse de souris. Après leur migration vers les gonades, les cellules germinales primordiales se multiplient jusqu’à l’arrêt vers 15 jpc. Commence alors l’établissement de la méthylation de novo de l’ADN. Les spermatogonies reprennent leurs divisions à 3 jpp et débutent leur méiose à 10 jpp. Les premiers spermatides ronds haploïdes apparaissent à 14 jpp. jpc : jours post-coïtum ; jpp : jours post-partum. B. Biogenèse et fonction des piARN du pachytène. Après export du noyau, le transcrit portant les clusters de piARN génère des piARN primaires qui s’associent avec Mili. L’activité endonucléase de Mili clive sa cible pour donner naissance à un piARN secondaire. Notons qu’un cycle ping-pong entre deux protéines Mili peut amplifier la production de piARN secondaires. Le piARN secondaire associé à Miwi2 peut entrer dans le noyau et guider la méthylation de l’ADN des rétrotransposons en s’appariant à un transcrit en cours d’élongation pour recruter une machinerie encore inconnue conduisant à la modification des histones et à la méthylation de l’ADN. |
| Dans le texte | |
 |
Figure 3. Modèle présentant le rôle des piARN dans la distinction entre les ARN du soi et du non-soi chez le ver C. elegans. Dans les lignées germinales, un complexe piARN associé à PRG-1 inspecte les ARN pour identifier des cibles avec une complémentarité imparfaite. Un système basé sur l’utilisation de la protéine CSR-1 et d’endo-siARN permet de protéger les ARNm cellulaires (1a). La reconnaissance de la cible, élément transposable ou élément étranger, par le piARN (1b) permet de recruter une ARNpolymérase- ARN-dépendante (ADAP) (2). Cette dernière génère de multiples petits ARN guides appelés 22G-ARN (3) qui s’associent à Wago9, soit pour amplifier le signal en recrutant ADAP et en générant de nouveaux 22G-ARN, soit pour inactiver le transposon. Deux mécanismes de répression sont observés ; ils interviennent dans le noyau par modifications épigénétiques (4a), et dans le cytoplasme par la dégradation de la cible (4b). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




